Nathan Kidley†
† Cresset, New Cambridge House, Bassingbourn Road, Litlington, Cambridgeshire, SG8 0SS, UK
摘要
Forge可以用基于场的QSAR或机器学习方法来建立健壮、预测性的QSAR模型[1]。此类模型的构建要求数据集化合物具有共同的结合模式、合理的结合强度或活性值范围、而且化合物数量足够多。在本案例研究中,使用Forge对一个含有196种JAK1和JAK2抑制剂的数据集建立了预测性机器学习与场的QSAR模型,自动选择具有最佳预测能力的模型,并使用3D显示功能来可视化和解释模型。
前言
Janus激酶(JAK kinase)一直是免疫和恶性相关疾病研究的活跃领域。 Tofacitinib是一种已上市的JAK抑制剂,用于治疗类风湿性关节炎、强直性脊柱炎和溃疡性结肠炎。Tofacitinib[2]的副作用限制了该药的使用剂量,因此,发现没有此类不良作用的化合物是非常重要的。在本案例研究中,源数据是默克专利(US9394282)[3]和科学文献[4]。这些文献的重点是设计最大剂量比托法替尼更高的化合物,从而对类风湿关节炎的治疗更加有效。Tofacitinib是一种泛JAK抑制剂,有证据表明JAK2活性是临床上观察到贫血的原因,因此需要增加JAK1相对于JAK2的选择性和良好的ADME特性。
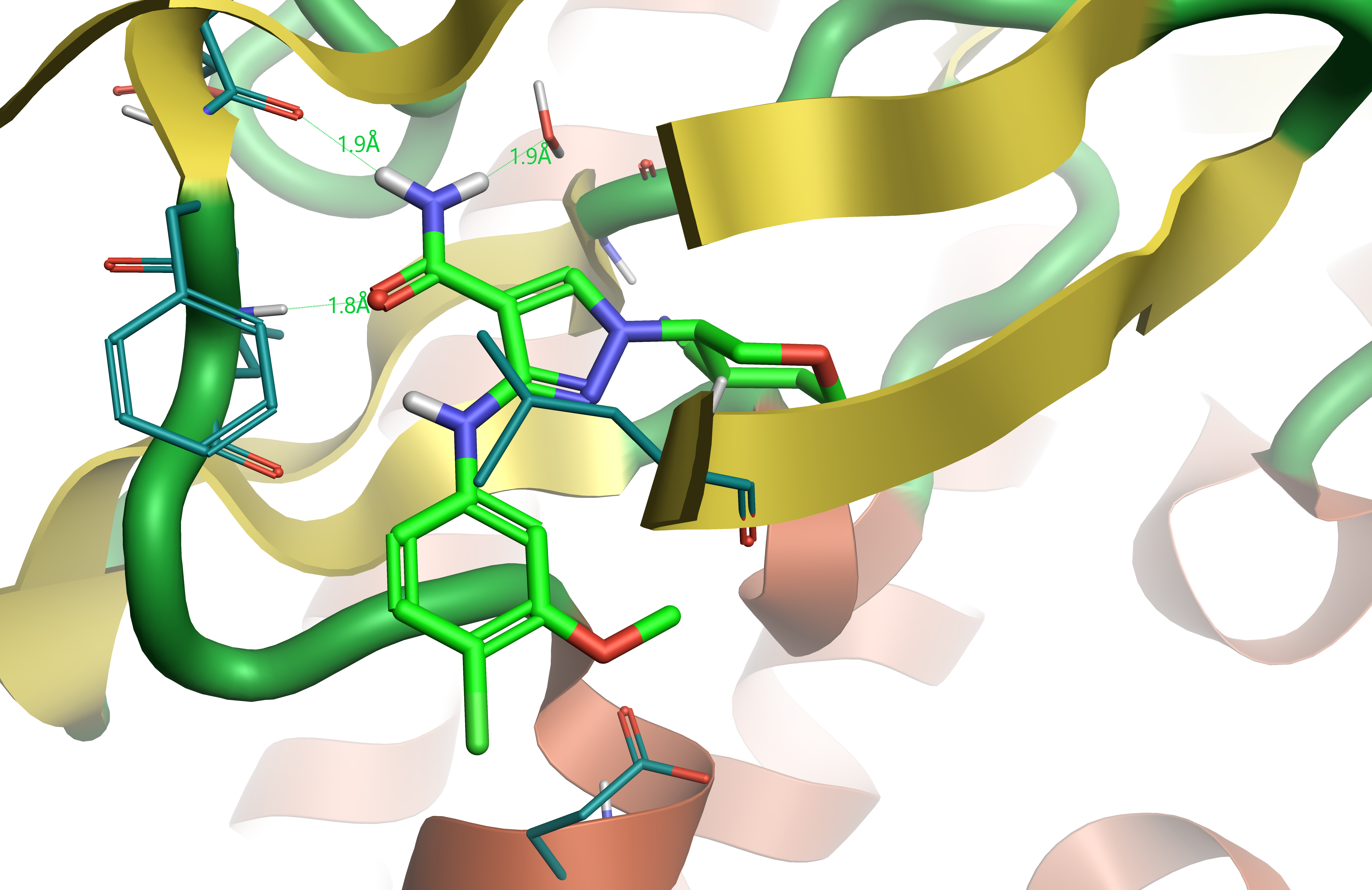
Figure 1. 化合物28与JAK1的共晶结构(PDB:5WO4),氢键用虚线表示。
分子的Cresset场点描述提供了分子周围空间区域的信息,这些信息与分子识别有关。 Forge中的QSAR方法直接使用从叠合过的分子场点确定的探针位置来采集训练集中每个分子的静电势或体积,并生成用于QSAR建模的描述符。
建立QSAR模型需要高质量的数据。使用3D描述符会增加另一个难度,要求生成合理的构象和有意义的叠合方式。通常,用于QSAR研究的良好叠合会非常紧密地将公共子结构或公共模板叠合起来,从而使得配体静电和体积变化更加易于解释活性差异的变化。
Forge可以使用基于场点的叠合技术[5]或最大公共子结构(MCS)算法来生成高质量的分子叠合。在叠合时还可以采用场和/或药效团约束以确保相互作用或子结构正确叠合。有时可以手动干预来改善叠合方式。但是,这么做必须小心,以避免创建出活性和非活性化合物人为地占据不同空间的QSAR模型。
构象搜索与分子叠合
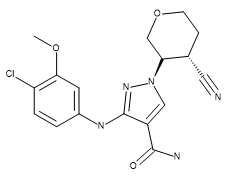
化合物28(表1)是感兴趣化合物的类似物。 JAK1与化合物284(图1)形成的复合物提供了其生物活性构象,可用作配体叠合的参比分子。
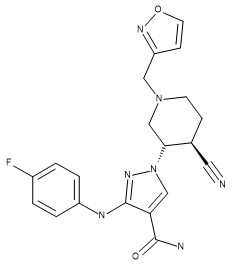
通过最大公共子结构方法将化合物US9394282,6-3和US9394282,6-36叠合到化合物28的活性构象上,并也用作参比分子以改善训练集化合物的叠合。
使用标准的“accurate but slow”模式进行构象搜索,使用标准的MCS叠合方法进行分子叠合,并将蛋白质排斥体积的硬度设置为“Soft”。
统计学分析与结果
将初始数据集(196个化合物)的80%划分为训练集(157种化合物),另外的20%划分为测试集(39个化合物)。全部196个化合物均已测定JAK1 pIC50值,其中172个化合物已测定JAK2 pIC50值。
Forge的Automatic QSAR采用默认选项、应用所有可用的机器学习方法、并选择具有最佳测试集预测统计性能的模型。此外,还建立了基于场的QSAR模型,并将其预测统计数据与机器学习方法的预测统计数据进行比较。表2列出了所有方法预测JAK1和JAK2活性的结果。
在这种情况下,表2中的所有模型在训练和测试数据集上的性能表现非常相似。 当看测试集的统计性能时,随机森林模型在总体上是最好的。 尽管Forge创建的所有模型都可以很好地用于预测新化合物的活性,但基于场的QSAR方法相对于机器学习方法具有更大的优势,即通过模型系数的视觉检查可以帮助模型的解释。 这极大地有助于理解什么驱动了模型中的活性,并对其进行解释以帮助设计新化合物。
Table 1.化合物28(PDB 5WO4的配体)、US93942化合物6-3以及US9394282化合物6-36的立体化学
| Compound 28 | US9394282, 6-3 | US9394282, 6-36 |
 |
 |
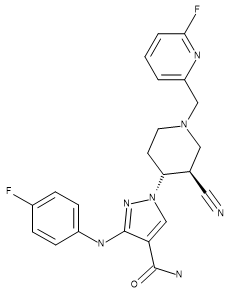 |
Table 2. 不同QSAR模型的预测与观测值的比较
| Model | Data set | R2 for JAK1 activity | R2 for JAK2 activity |
| Field QSAR |
Training |
0.792 | 0.794 |
| Cross validation | 0.589 | 0.541 | |
| Test | 0.634 | 0.586 | |
| KNN | Training | 0.6 | 0.536 |
| Test | 0.626 | 0.503 | |
| Random Forest | Training | 0.906 | 0.902 |
| Cross validation | 0.524 | 0.521 | |
| Test | 0.655 | 0.622 | |
| Relevance Machine | Training | 0.778 | 0.745 |
| Cross validation | 0.556 | 0.545 | |
| Test | 0.589 | 0.623 | |
| Support Vector Machine | Training | 0.83 | 0.788 |
| Cross validation | 0.55 | 0.526 | |
| Test | 0.636 | 0.625 |
模型可视化与解释
Forge中的Field QSAR是一种基于偏最小二乘分析的回归方法,因此描述符和活性的线性关系可用于模型的可视化解释。 “模型系数”图中的大点表示该模型在该位置的静电/空间场之间发现了很强的相关性,并且匹配这些特征的化合物具有很高的活性。
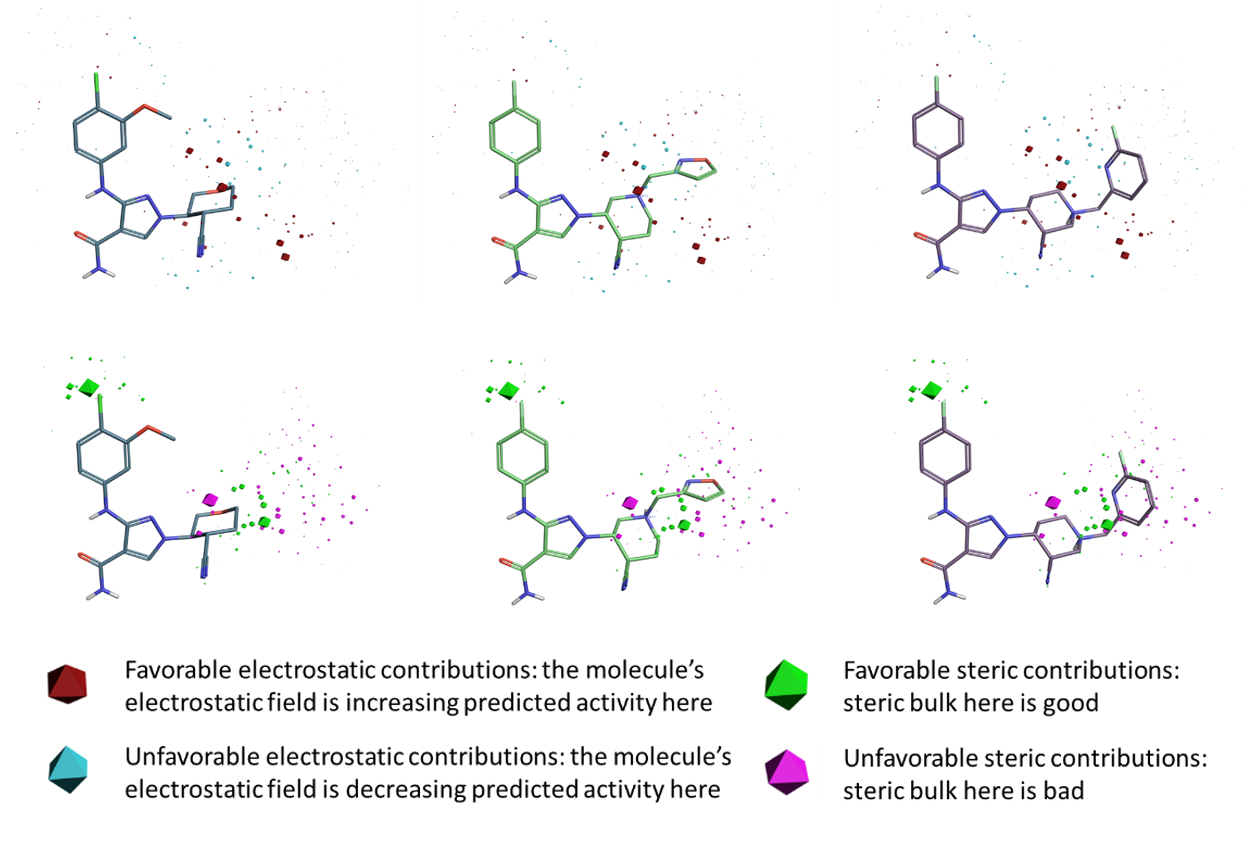
Figure 2.JAK1基于场的QSAR模型的模型系数图。包括静电系数(上)、立体系数(下)、以及化合物28(左)、US9394282 6-3(中)与US939482 6-36(右)。
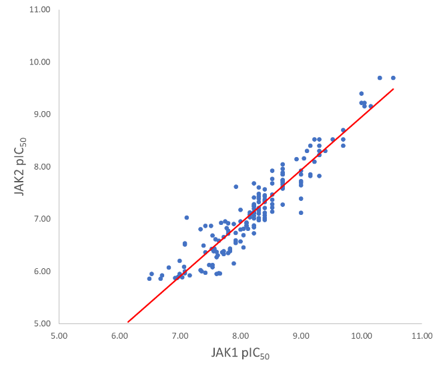
Figure 3.绘制JAK1与JAK2 pIC50活性值的相关性。拟合最好的线的R2为0.89,斜率不为1表明对化合物JAK1比JAK2具有更强的活性。
图2给出了主成分数为5时的JAK1模型的静电和立体模型系数。总体而言,在参比化合物的哌啶/四氢呋喃环与末端异恶唑/吡啶环之间的区域中,Field-Based QSAR模型的静电或空间系数没有特别紧密的分组。这可能是由于数据集中不同的环构象而难以获得非常紧密的叠合。
静电模型系数多面体比空间模型系数多面体要小得多,大小代表了它们对QSAR模型的重要性。最显著的正静电系数在哌啶环间位附近。
尽管在吡啶和异恶唑环附近有比较分散的立体系数多面体分布,但始终报告该区域存在不利的立体系数,这表明在该区域中大取代基对提高加化合物的活性不利。在苯环氯取代基附近可以发现存在有利的立体系数区域(如图2底部所示的绿色多面体)。另一个立体有利的区域在对位的哌啶环附近。而不利的立体效应(品红色多面体)区域是在哌啶环的左边间位。
在JAK2体外数据上观察到几乎相同的模式,这并不令人奇怪地看到JAK1和JAK2活性数据之间具有高度的相关性(R2=0.89,图3)。 分析结果还发现普遍性的JAK1选择性偏倚,该数据集中除了两个化合物外的所有化合物对JAK1的选择性明显高于JAK2。 Siu等人[4]认为JAK1中的E966变为JAK2中的D939是某些化合物选择性提高的原因。在本案例研究中使用的数据集中,苯环上的取代基变体很少,并且QSAR模型在此处未发现强SAR信号。 用Activity Atlas来探测探索到化学空间,从立体和静电的角度来看发现对氯取代基的值比较高。Activity Atlas分析结果还指出,从静电角度看,这些化合物的间位和对位尚未得到系统的研究。
结论
使用多种不同的QSAR方法,FORGE成功地构建了196个JAK1/2激酶抑制剂的3D-QSAR模型。Activity Atlas与基于场的QSAR模型便于模型的可视化和解释,从而深入了解你所探索了的化学空间、是什么对活性有贡献以及数据集的任何限制。该见解和预测值是一种强有力的组合适于待合成与测试的新化合物的设计与优先性排序。
在建立QSAR模型时应始终小心,确保数据足够一致、具有足够大的活性范围和分布。应仔细地审查任何模型以确保结果不是过度参数化的假象,该模型不存在插入现象,并能以稳健的方式进行外推。除了这些挑战之外,3D-QSAR模型还具有构象搜索和配体叠合等问题需要解决。
文献
- https://www.cresset-group.com/software/forge/
- Flanagan et al., J. Med. Chem. 2010, 53, 8468-8484
- Patent US9394282B2
- Sui et al., J. Med. Chem. 2017, 60, 9676-9690
- Cheeseright et al, J. Chem. Inf. Model., 2006, 46, 66