Nathan Kidley†
† Cresset, New Cambridge House, Bassingbourn Road, Litlington, Cambridgeshire, SG8 0SS, UK
초록
ForgeTM,1은 구조-활성 상관(SAR) 및 분자 설계를 위한 Cresset사의 ligand 기반 워크벤치로 Field QSAR 또는 머신 러닝 방법을 통해 신뢰성 및 예측성이 높은 정량적 구조-활성 상관(QSAR) 모델을 구축할 수 있습니다. 공통적인 결합 모드를 가지며 적당한 결합력 또는 활성이 있으면 어떤 데이터 세트에 대해서도 일정한 모델을 구축할 수 있습니다. 본 사례에서는 Janus Kinase JAK 1 및 JAK2에 대해 저해 활성을 갖는 196개의 분자로 구성된 데이터 세트를 이용하여 Forge를 통한 머신 러닝 및 Field QSAR 예측 모델을 구축합니다. Forge QSAR model information tool을 이용하여 예측 성능을 극대화할 모델을 선택하고 3D display 기능을 이용하여 모델의 시각화 및 해석을 진행하였습니다.
서론
Janus Kinase는 면역 및 악성 질환에 대한 활발한 연구 영역입니다. Tofacitinib은 류마티스 관절염, 강직성 척추염, 궤양성 대장염의 치료제로 사용되는 JAK 억제제입니다. Tofacitinib2은 유해한 부작용으로 인해 처방 용량이 제한되며, 이러한 부작용이 없는 화합물의 개발에 큰 관심이 있습니다. 본 사례 연구에서는 Merck사의 특허(US9394282)3와 문헌4의 데이터를 사용합니다. 이들의 연구 초점은 Tofacitinib보다 최대 처방 용량이 높고 더 효과적인 류마티스 관절염 치료제를 설계하는 것입니다. Tofacitinib은 JAK 범 억제제로 임상연구에서 JAK2 활성이 원인이 되어 빈혈을 유발하는 것으로 알려져 있어 우수한 ADME 특성과 함께 JAK2 대비 JAK1에 대한 선택성 향상이 요구됩니다.
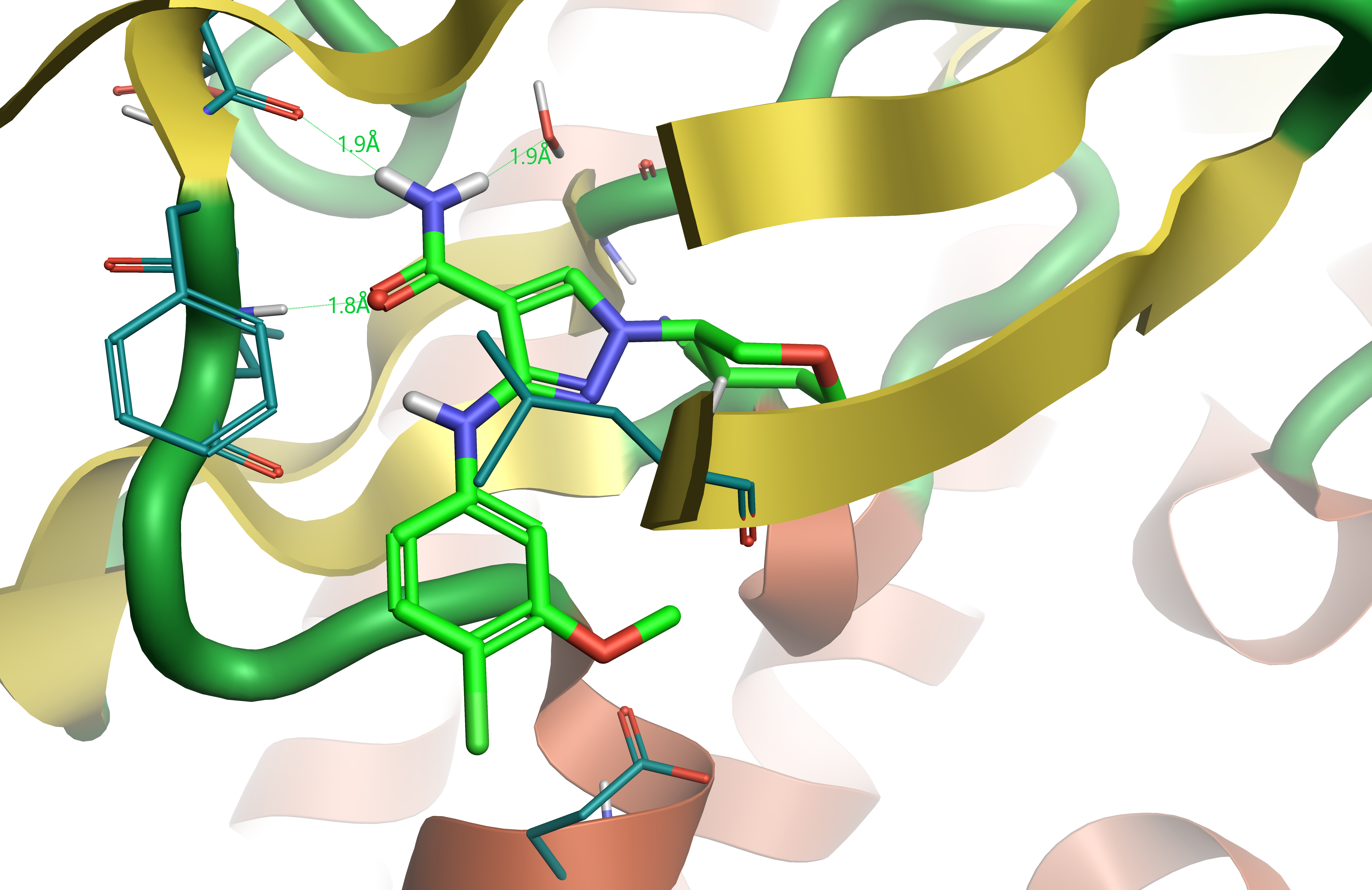
그림 1. JAK1 활성부위(PDB:5WO4)에 결합한 Compound 28. 수소 결합은 점선으로 표시하였음.
Cresset의 field point는 분자 인식과 관련된 분자를 둘러싼 공간 영역에 대한 정보를 제공합니다. Forge의 QSAR 방법은 align 된 분자들의 field point로부터 직접적으로 결정된 probe 좌표를 이용하여 QSAR 모델링을 위한 descriptor를 생성합니다.
좋은 QSAR 모델을 구축하기 위해서는 모델 구축을 위한 고정밀 데이터가 필요합니다. 3D descriptor를 이용하는 것은 각 분자의 적절한 conformation과 전체 분자에 걸친 정확한 중첩이 필요하며 그 자체로 어려운 문제입니다. 일반적인 QSAR에서는 공통 부분 구조 또는 공통 템플릿 분자에 대해 전체 분자를 정밀하게 align 하여 electrostatics와 부피 차이에 의한 활성에 미치는 영향을 명확히 파악하려는 노력을 합니다.
Forge는 field point overlay 기술5 또는 Maximum Common Substructure(MCS) 알고리즘을 이용하여 정밀도 높은 alignment를 생성할 수 있는 많은 옵션을 제공합니다. 또한, 이들 alignment 방법에 field와 pharmacophore constraint를 적용하여 상호작용 부위나 부분 구조를 더욱 잘 align 할 수 있습니다. 수작업으로 alignment의 수정을 필요로 하는 경우도 있습니다. 이때, 활성 및 비활성 화합물을 구별하여 의도적으로 분자를 배향시킨 모델을 만드는 일이 없도록 주의가 필요합니다.
화합물의 conformation hunt 및 alignment
Compound 28(표 1)은 관심 화합물의 유사체입니다. Compound28과 JAK1의 protein-ligand 복합체의 결정 구조(그림 1)4은 ligand alignment의 reference로서 유용한 bioactive conformation을 제공합니다.
표1의 화합물 US9394282, 6-3과 US9394282, 6-36을 상기 Compound 28의 X-ray conformation에 Maximum Common Substructure(MCS)로 align 하였고, training set 내 화합물의 alignment를 전체적으로 조정하기 위한 추가 reference로 이용하였습니다.
Conformation hunt 파라미터는 표준 ‘Accurate but Slow’, 표준 MCS alignment 방법, Hardness of protein excluded volume을 ‘Soft’로 설정했습니다.
통계분석 및 결과
초기 data set(196개 화합물)을 80%의 training set(157개 화합물)과 20%의 test set(39개 화합물)로 분할하였습니다. 모든 196개 화합물은 측정된 JAK1 pIC50을 가지며, 172개 화합물에 대해서만 JAK2 pIC50 값을 가집니다.
QSAR 모델 구축을 위한 Forge의 기본 옵션은 이용 가능한 모든 머신 러닝 방법을 자동으로 적용하고 최상의 test set 예측 통계를 갖는 모델을 선택합니다. 또한, Field QSAR 모델도 구축하여 머신 러닝 방법의 예측 통계와 비교하였습니다. JAK1 및 JAK2 활성에 대한 모든 QSAR 방법별 결과를 표 2에 나타냅니다.
이 사례에서 표 2의 모든 모델은 training set과 test set의 경향이 매우 유사한 것으로 나타났습니다. Test set에 대한 통계적 성능을 비교할 때, random forest 모델이 전반적으로 다소 우수해 보입니다. 여기에 Forge에 의해 생성된 모델은 모두 새로운 화합물의 활성을 예측하기에 충분히 적합합니다. 또한, Field QSAR는 모델 해석에 시각적 검증을 가할 수 있다는 장점이 있어 활성 향상에 기여하는 화학적 요소를 이해하고 새로운 화합물을 설계하는 데 큰 도움이 됩니다.
표 1. Compound 28 (결정 구조 ligand), US9394282, 6-3, 및 US9394282, 6-36의 입체화학 구조
| Compound 28 | US9394282, 6-3 | US9394282, 6-36 |
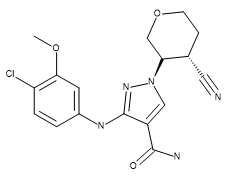 |
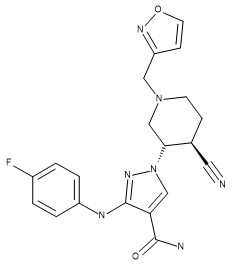 |
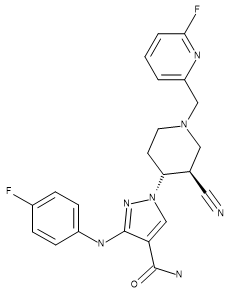 |
표 2. QSAR 모델의 실험과 예측 계산의 비교
| Model | Data set | R2 for JAK1 activity | R2 for JAK2 activity |
| Field QSAR |
Training |
0.792 | 0.794 |
| Cross validation | 0.589 | 0.541 | |
| Test | 0.634 | 0.586 | |
| KNN | Training | 0.6 | 0.536 |
| Test | 0.626 | 0.503 | |
| Random Forest | Training | 0.906 | 0.902 |
| Cross validation | 0.524 | 0.521 | |
| Test | 0.655 | 0.622 | |
| Relevance Machine | Training | 0.778 | 0.745 |
| Cross validation | 0.556 | 0.545 | |
| Test | 0.589 | 0.623 | |
| Support Vector Machine | Training | 0.83 | 0.788 |
| Cross validation | 0.55 | 0.526 | |
| Test | 0.636 | 0.625 |
모델의 시각화 및 해석
Forge의 Field QSAR는 부분 최소 제곱법(PLS) 분석에 기반한 회귀법으로 descriptor와 활성치의 선형 관계를 이용하여 모델을 시각적으로 해석할 수 있습니다. ‘모델 계수’ 플롯의 점의 크기는 그 위치에 electrostatic/steric field와 공통의 특징을 가지는 화합물이 실제의 활성과 얼마나 상관관계가 있는가 하는 정도를 나타냅니다.
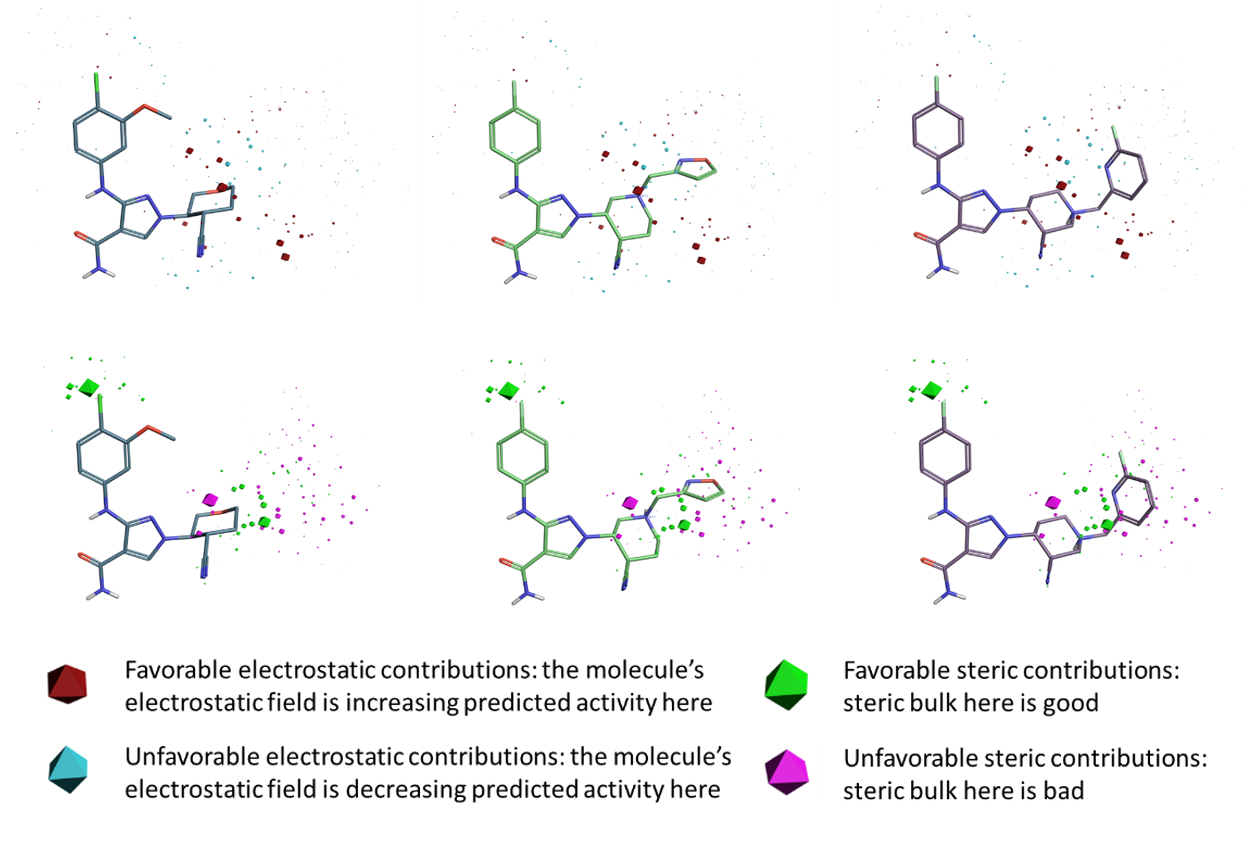
그림 2. JAK1 Field QSAR 모델의 모델 계수 플롯. Forge의 reference 구조(compound28(왼쪽)의 X-ray pose 및 align 한 US9394282, 6-3(중앙), US9394282, 6-36 (오른쪽) 화합물)의 electrostatic 계수(상단); steric 계수(하단)
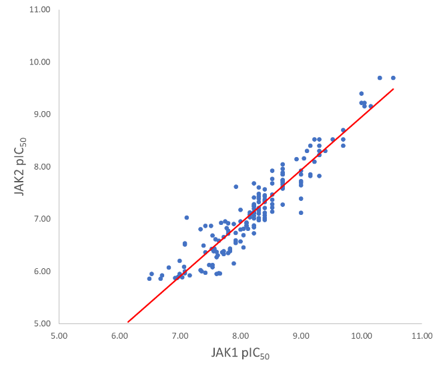
그림 3. JAK1과 JAK2간의 pIC50 활성의 상관관계. 회귀 결정계수 r2는 0.89. JAK2 보다 JAK1에 대한 활성이 더 높은 경향을 나타냄.
5 성분 JAK1 모델의 electrostatic 및 steric 모델 계수를 그림 2에 나타냅니다. 전반적으로 Field QSAR 모델은 Reference 화합물의 piperidine/tetrahydrofuran ring과 말단 isoxazole/pyridine ring 사이의 영역에서 electrostatic 또는 steric 계수에 대한 강한 상관관계를 보이지 않습니다. 이는 데이터 세트 내 분자 골격 고리 부분의 conformation 차이가 있어 정밀한 alignment가 어려운 것이 하나의 원인이라고 생각됩니다.
Electrostatic ‘모델 계수’ 다면체는 존재하지만 steric 다면체에 비해 훨씬 작습니다. 그 크기는 QSAR 모델에서의 활성에 대한 기여를 나타냅니다. 가장 중요한 positive electrostatic 계수는 piperidine ring의 같은 메타 위치 근처에 있습니다.
Pyridine 및 isoxazole ring 근처에 steric 계수의 핑크 다면체가 많이 흩어져 있습니다. 이처럼 일정 영역에 unfavorable steric 계수가 많이 발견되는 경우, 이 영역에 큰 치환기가 있으면 활성 발현에 불리해진다는 것을 의미합니다. Phenyl ring의 chlorine 치환기 근처에 favorable steric 계수 영역(그림 2 하단의 녹색 다면체)을 확인할 수 있습니다. 또 다른 영역은 piperidine ring의 para 위치 근처에 있습니다. 그에 반해 piperidine ring의 왼쪽 meta 위치에는 unfavorable steric 영역(magenta 다면체)이 있습니다.
JAK2 in-vitro 데이터에서도 거의 동일한 패턴이 관찰되며, JAK1과 JAK2 활성 데이터 간에 높은 상관관계가 있는 것은 놀라운 일이 아닙니다(R2 0.89, 그림 3). 또한, assay 결과는 일반적인 JAK1 선택성 편향을 보여주며 데이터세트 내 두 화합물을 제외한 모든 화합물이 JAK2 보다 JAK1에 훨씬 더 선택적입니다. Siu et al4는 JAK1의 E966과 JAK2의 D939, 단 하나의 아미노산 차이가 화합물의 선택성과 깊이 관련되어 있음을 발견하였습니다. 이 사례 연구에 이용된 데이터 세트에서는 phenyl ring 치환기의 다양성이 거의 없으며 따라서 QSAR 모델은 여기에서 강한 SAR signal을 검출할 수 없습니다. Activity Atlas는 탐색된 chemical space를 정밀하게 조사하기 위해 이용되며 steric 및 electrostatic 관점 모두에서 para-chloro 치환기의 높은 활성 기여를 확인할 수 있습니다. Activity Atlas는 meta 및 para 위치의 electrostatic 특징이 주어진 데이터 세트 안에서 충분히 관찰되지 않았다는 것도 알려줍니다.
결론
Forge를 이용하여 다양한 QSAR 방법을 적용한 196개의 JAK1/2 kinase 억제제 데이터 세트에 대한 3D-QSAR 모델을 성공적으로 구축할 수 있었습니다. Activity Atlas 및 field QSAR 모델은 모델의 시각화 및 해석을 용이하게 하며 탐색된 chemical space, 활성에 기여하는 요소 및 데이터 세트의 제한에 대한 다양한 정보를 제공합니다. 이들 정보와 예측치를 활용하는 것은 새로운 화합물의 설계를 용이하게 하고 합성과 테스트를 위한 우선순위를 정하는 데 큰 도움이 됩니다.
QSAR 모델을 만들 때는 항상 데이터가 충분히 일관적인지와 활성값의 범위가 충분히 넓고 고르게 분포되어 있는지 잘 확인해야 합니다. 결과 모델이 과도한 매개변수화의 산물이 아님을 확인하기 위해 선택한 모델로 내삽적이 아닌 외삽적인 활성 예측이 가능하다는 것을 제대로 검증해야 합니다. 이 외에도 3D-QSAR 모델은 conformation 탐색과 ligand alignment를 얼마나 정확하게 실시하는가 하는 복잡한 과제를 포함하고 있습니다.
Forge의 무료 평가
Forge는 Flare의 일부 모듈로 포함되어, Flare의 flexible licensing 옵션 중 Flare LigandPro에서 제공되고 있습니다. 이를 실제 신약 개발 프로젝트에서 테스트 및 평가해 보고 싶으신 분들은 무료 evaluation을 신청해 주시기 바랍니다.
References and Links
- https://www.cresset-group.com/software/forge/
- Flanagan et al., J. Med. Chem. 2010, 53, 8468-8484
- Patent US9394282B2
- Sui et al., J. Med. Chem. 2017, 60, 9676-9690
- Cheeseright et al, J. Chem. Inf. Model., 2006, 46, 66