본 사례에서는 Cresset사의 bioisostere 치환 및 scaffold hopping 도구인 Spark의 새로운 docking 기능을 이용하여 GSK-3β의 활성 자리에서 직접적으로 ligand-protein간 상호작용을 할 수 있는 대체 R-group을 발견하고 이들을 서열화 하는 것에 대해 소개합니다.
서론
GSK-3β는 다양한 세포 과정에 관여하는 Serine/Threonine kinase입니다. 특히, 성인의 뇌 속에 널리 존재한다는 점에서 뉴런의 신호 전달 경로에서의 중요한 역할이 시사됩니다. 또한, GSK-3β는 조울증, 우울증, 조현증과 같은 기분 장애와 관련이 있으며, 해당 질병의 치료에 대한 기대로 GSK-3β 억제제 개발이 활발히 이루어지고 있습니다.
1H-indazole-3-carboxamide는 최근에 발견된 ATP-경쟁적 GSK-3β 억제제의 새로운 구조 클래스입니다1,2. Cmpd 1(그림 1 – 좌 및 표 1)은 효능 있는 효소 및 세포 GSK-3β 억제제입니다. 또한, 해당 화합물은 선호되는 kinase 선택성 프로파일을 보여주며, 조증 동물 모델에서 PK 특성과 그 유효성이 확인되었으나 상당한 hERG 저해를 일으키는 것으로 보고됩니다.
hERG 저해를 회피하기 위한 Cmpd1의 lead 최적화2는 화합물의 염기도(basicity)를 낮추기 위해 (2-methoxyethyl)-4-methylpiperidine moiety를 oxanyl기로 치환하고 친지질성(lipophilicity)을 낮추기 위해 di-F-phenyl moiety에 극성이 더 높은 치환기를 도입하여 수행했습니다. 이러한 노력으로, 보다 높은 GSK-3β 저해 활성을 가지며 hERG 저해를 회피하는 Cmpd 14(그림 1 – 우 및 표 1)을 발견할 수 있었습니다.
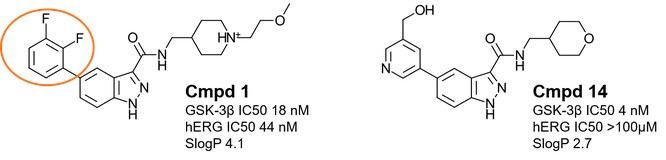
그림 1. 좌: Cmpd1은 GSK-3β 저해 활성은 높으나 hERG 저해를 야기. 우: 최적화된 Cmpd 14는 보다 높은 GSK-3β 저해 활성을 가지며 hERG 저해를 회피.
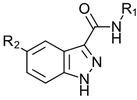
Cmpd 1의 유사체에 대해 상세히 검토된 SAR 데이터를 표 1에 나타냅니다.
표 1. 1H-indazole-3-carboxamide core의 R1 및 R2 위치의 SAR 탐색1,3
여기서는 Spark의 ‘docking’ 기능을 이용하여 di-F-phenyl moiety(그림 1 – 좌의 주황색 원)에 대한 대체 R-group을 신속하게 찾아냅니다. 이 방법을 이용하면 표적 단백질의 비어 있는 포켓 안으로 ligand와 fragment를 성장시킬 수 있으며, 기존 starter 또는 레퍼런스 분자에 의해 매핑되지 않은 protein의 활성 자리와 상호작용하는 새로운 결과를 찾을 수 있습니다. 더 구체적으로는 GSK-3β 활성 자리와 직접적으로 상호작용하여 저해 활성을 높이는 동시에 친지질성(lipophilicity)를 낮춰 hERG 저해를 줄이는 대체 R-group을 탐색합니다.
Protein-ligand 복합체
Spark에서 docking 방법을 이용하기 위해서는 정밀하게 준비된 protein 구조(수소 원자의 복원, 내부 수소 결합 네트워크의 최적화, 원자 충돌 회피, 적절한 양성자화 상태)를 이용하여 실험을 시작할 필요가 있습니다. GSK-3β와 결합한 Cmpd 1의 protein-ligand 복합체 구조(PDB: 6TCU)의 준비는 Cresset사의 구조 기반 설계 플랫폼인 FlareTM를 이용하여 수행했습니다.
Protein-ligand간 contact(그림 2)는 indazole carboxamide scaffold가 세 개의 수소 결합(Asp133의 주골격 carbonyl기, Val135의 주골격 NH와 주골격 carbonyl기)에 의해 GSK-3β의 hinge 도메인과 상호작용하고 있음을 보여줍니다. piperidine moiety는 용매에 완전히 노출되어 있고 di-F-phenyl ring은 촉매 작용 중심의 Lys85에 가까이 위치해 있습니다.
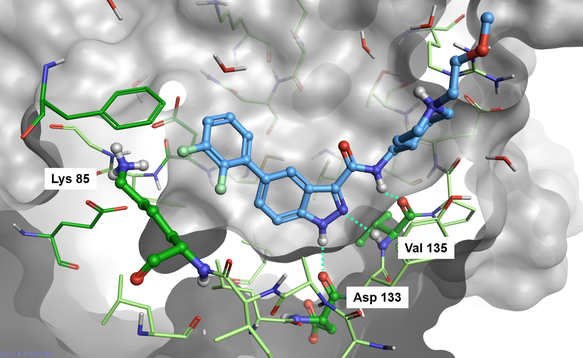
그림 2. GSK-3β 활성 자리에 결합한 Cmpd1(PDB: 6TCU)
Spark에서의 docking
Spark의 docking 워크플로우는 starter 분자에서 치환할 부분을 삭제하는 것에서부터 시작합니다(그림 3, A-B). 그 다음에 일부를 잘라낸 starter 분자에 새로운 fragment를 붙이고 Cresset XED force field로 계산된 protein electrostatics에 따라 protein의 활성 자리에 적절히 배치합니다(그림 3, C). 그리고 새로운 결과 분자의 pose가 Lead FinderTM의 ‘score only’ docking 모드를 이용하여 최적화됩니다(그림 3, D). 최종적으로, 알려진 ligand의 결정학적 pose를 정확하게 재현하도록 최적화된 Lead Finder의 ‘Rank’ scoring function을 이용하여 최상의 결과를 서열화 합니다(score는 negative 할수록 좋음).
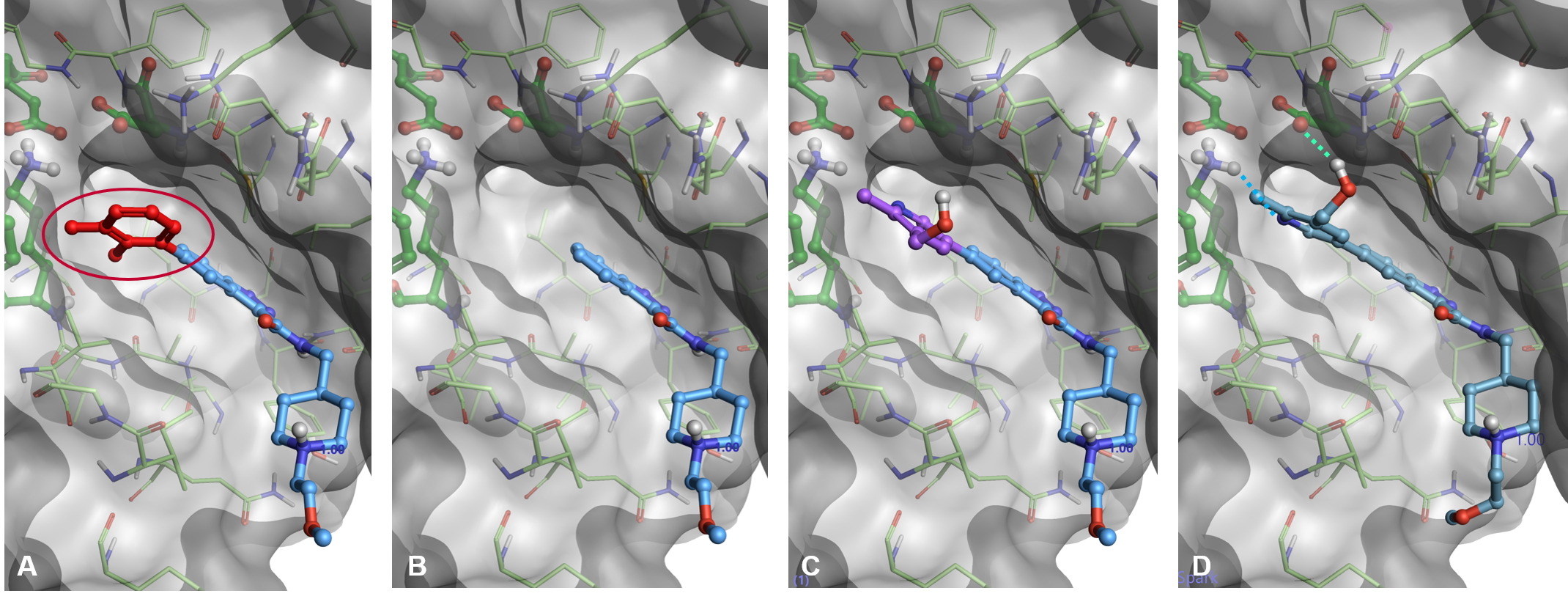
그림 3. Spark의 docking 워크플로우. A) starter 분자의 치환 부분의 선택 B) 선택한 fragment의 삭제 C) 새로운 fragment를 잘라낸 Starter 분자에 적절하게 결합 D) 새로운 결과 분자를 Lead Finder로 최적화
여기서는 간단한 절차를 거치는 것만으로 적절한 실험을 쉽게 준비할 수 있는 ‘Scaffold Hopping or R-group Replacement’ wizard를 이용했습니다(그림 4 – 좌). Flare로 최적화한 6TCU protein-ligand 복합체를 Spark로 input protonation state 옵션을 적용하여 가져오고, Starter molecule로 Cmpd 1을 선택하고 모든 결정학적 물 분자를 제거하였습니다. Starter 분자의 치환할 부분으로 di-F-phenyl moiety를 선택하였습니다(그림 4 – 우).
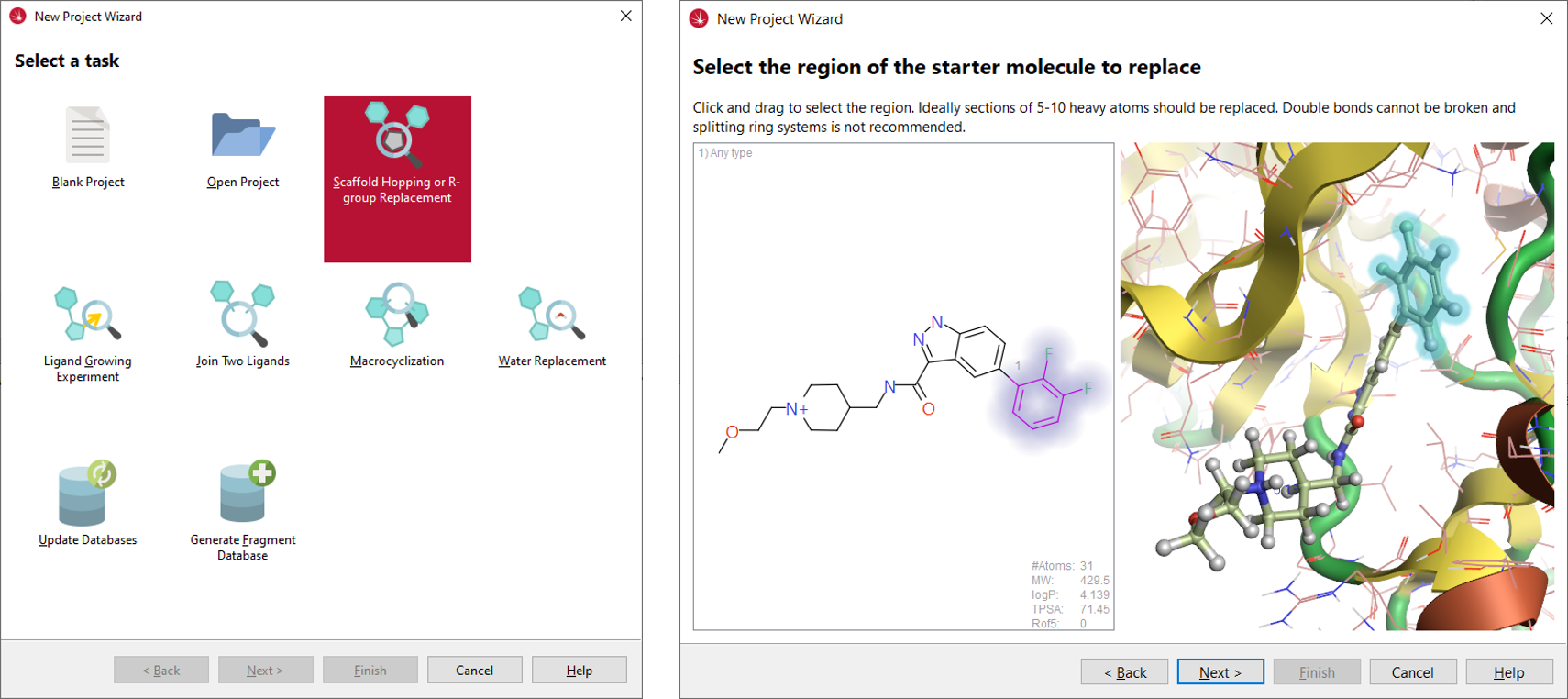
그림 4. 좌: Spark의 new project wizard. 우: Spark wizard에서 starter 분자의 치환 선택 영역이 강조 표시됨.
Wizard 조작을 완료하면 Spark의 calculation method가 ‘Docking’으로 설정됩니다. 이 방법은 ligand similarity에 기반한 기존의 Spark 탐색에 비해 계산량을 많이 필요로 하기 때문에 적절한 physico-chemical 필터를 설정하여 탐색 공간을 제한하고 계산 시간을 단축하여 목적에 맞는 실험을 수행하는 것이 권장됩니다. Starter molecule(MW 429.5, SlogP 4.1)을 guide로 하여 결과 분자의 분자량을 466 미만 그리고 SlopP는 3.5 미만으로 설정했습니다. 또한, 적절한 SMARTS 필터를 이용하여 치환된 phenyl기와 2-hydroxy-pyridine을 결과 리스트에서 배제하도록 하였습니다(그림 5).
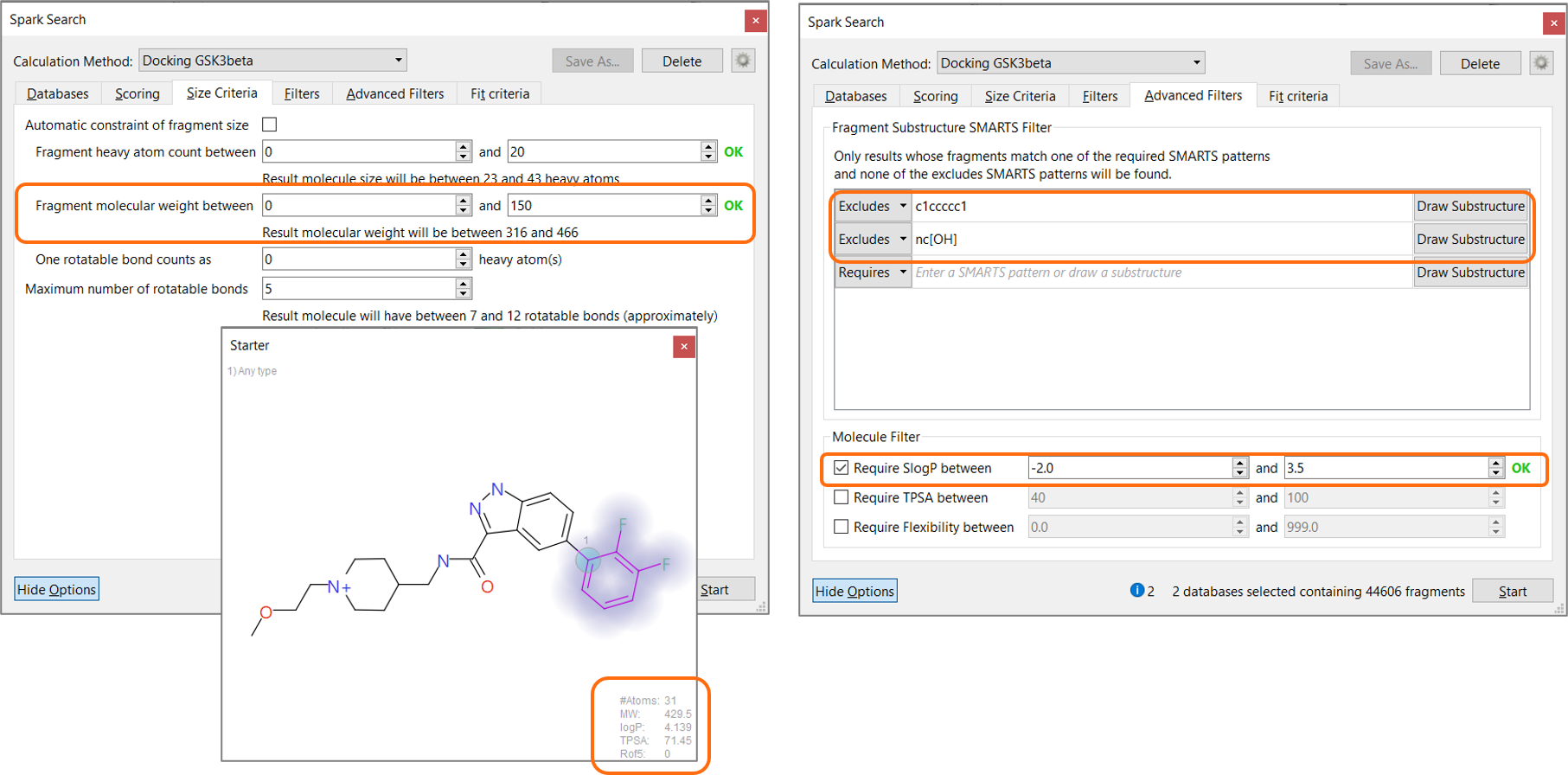
그림 5. 합목적적 탐색 및 계산 시간 단축을 위한 적절한 physico-chemical 필터의 설정
실험은 eMolecules building block4에서 유래된 4.5K aromatic boronic acid와 40K aromatic halide 데이터베이스를 이용하여 수행하였으며 문헌의 chemistry를 가능한 한 재현하도록 하였습니다2.
결과
Docking 실험의 결과로, top-scoring cluster(그림 6, 좌) 중에서 pyridine-3-yl 치환기를 발견할 수 있었으며, 표 1에서 보고된 것과 동일하거나 유사한 R-group(그림 6 – 중)을 포함하고 있음을 알 수 있습니다. 또한, Top scoring Spark 결과 중에서 제한된 분자량과 친지질성(lipophilicity)를 갖는 다양한 bicyclic heterocycle도 발견할 수 있었습니다. 이는 lead 시리즈의 추가 탐색을 위해 pyridine-3-yl 치환기에 대한 흥미로운 대안을 제공할 수 있습니다. 몇 가지 예를 그림 6의 오른쪽에 나타내었습니다.
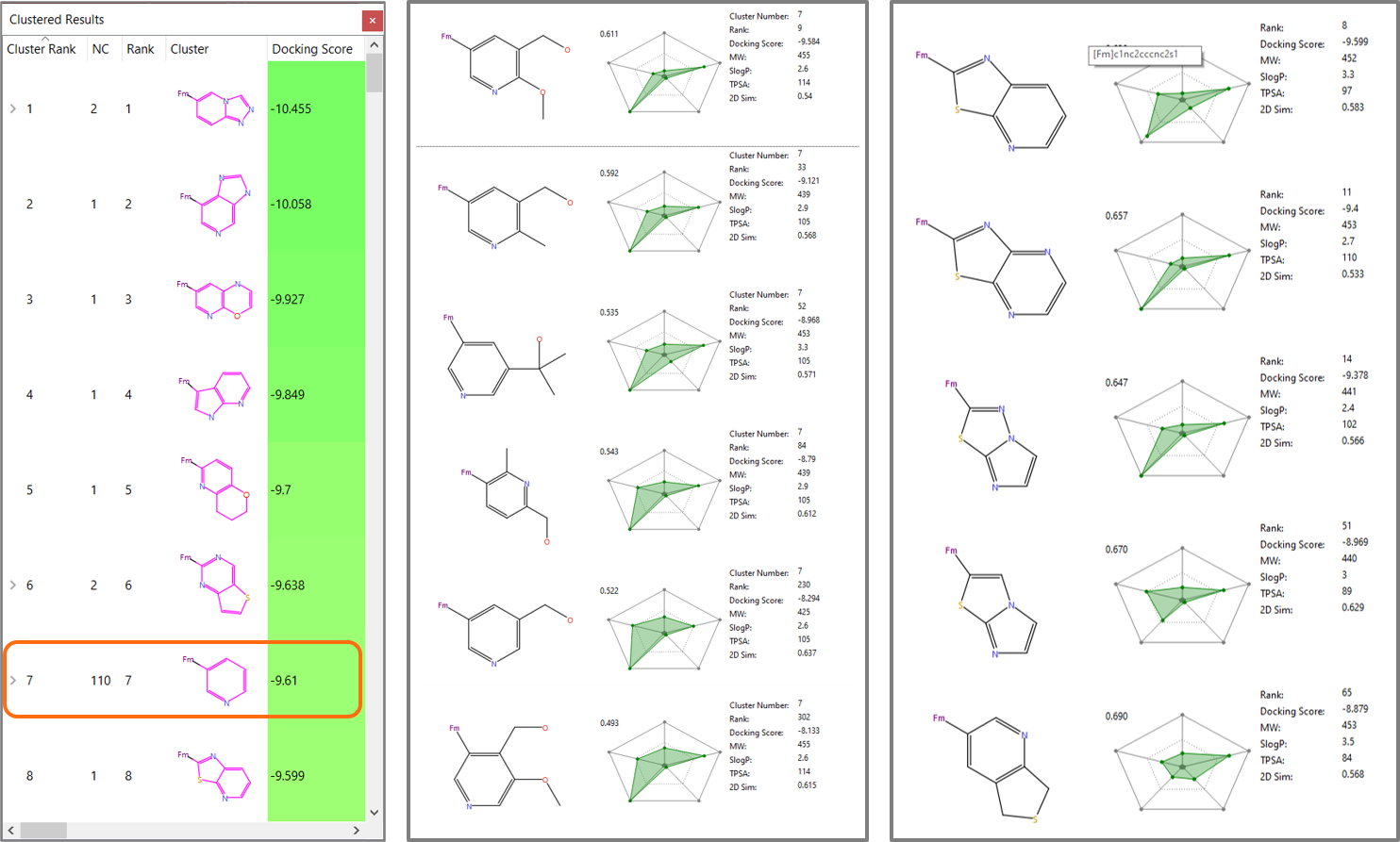
그림 6. 좌: Pyridin-3-yl 치환기2는 cluster rank 7. 중: 표1의 것과 유사한 pyridine-3-yl 치환기 cluster의 member. 우: 제한된 분자량과 친지질성을 갖는 bicyclic heterocycle.
흥미롭게도, pyridine-3-yl cluster의 많은 member에 대해서 Spark가 제시한 binding mode는 Lys85와 수소 결합 상호작용과 어떤 경우에는 Phe67과 aromatic-aromatic 상호작용을 포함하고 있으며, 화합물 OH84(PDB: 6Y9R, 그림 7)의 결정학적 binding mode와 일치합니다. 당연하게도 docking 방법은 residue와 추가로 수소 결합 상호작용을 할 수 있는 치환기(hydroxyl기와 같은)를 가진 3-pyridine을 선호하는 경향이 있으며, 이 경우에는 활성 부위에 있는 Asp200 등이 그것을 실제로 가능하게 합니다.
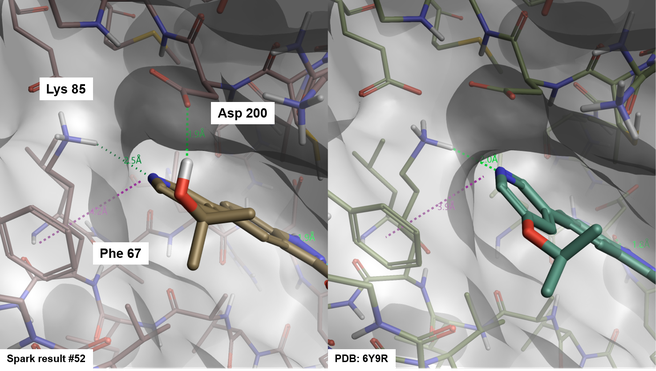
그림 7. Spark 결과 #52(좌) 및 PDB: 6Y9R(우)의 화합물 OH8의 binding mode
결론
Spark는 ‘docking’ 기능을 통해 protein 활성 자리에서 직접적으로 ligand-protein 상호작용을 가능하게 하는 fragment를 탐색할 수 있습니다. 이 방법을 이용하면 ligand와 fragment를 표적 protein의 비어 있는 포켓 안으로 성장시켜 기존 starter 또는 레퍼런스 분자에는 없던 상호작용을 하는 새로운 분자를 발견할 수 있습니다.
참고 문헌
1. Ombrato R. et al., J. Chem. Inf. Model. 2015, 55, 2540−2551
2. Prati F. et al., ACS Med. Chem. Lett. 2020, 11, 825−831
3. Buonfiglio R. et al., Molecules 2020, 25(9), 2163
4. https://www.emolecules.com/info/products-building-blocks.html