Ryuichiro Hara†, Stuart Firth-Clarke†, Nathan Kidley†
†Cresset, New Cambridge House, Bassingbourn Road, Litlington, Cambridgeshire, SG8 0SS, UK
초록
도킹은 단백질 구조를 활용하여 리간드의 결합 포즈를 생성하고 해당 상호작용을 평가하는 방법입니다. 이 방법은 신약개발에서 설계한 분자를 스크리닝하고 이들의 우선순위를 정하는데 널리 이용됩니다. 도킹 소프트웨어는 일반적으로 단백질을 고정된 구조로 처리하는 반면, 리간드는 입체형태적으로(conformationally) 유동적인 것으로 처리합니다. 경우에 따라서 이는 단백질 구조가 주어진 리간드의 생물학적으로 관련 있는 conformation이 아니기 때문에 문제가 되며 무의미한 도킹 결과를 가져올 수 있습니다. 본 사례 연구는 먼저 분자동역학을 이용하여 Flare의 앙상블 도킹을 위한 단백질 conformation들의 앙상블을 생성하는 접근 방법의 유효성을 검증합니다.
서론
결정 격자 내 상호작용으로 인한 결정 패킹 허상(crystal packing artifacts), 정제 또는 결정화 과정에서 사용된 보조 인자, 또는 활성 부위 내 리간드가 없는 상태에서 단백질이 용해된 경우 등 X-선 단백질 구조가 생물학적으로 유의미한 형태가 아닐 수 있는 몇 가지 이유가 있습니다. 또한, 결정 구조는 단백질의 동적 특성을 반영하지 않는 시간의 스냅 사진을 나타냅니다. 단백질을 고정된 구조로 처리하는 도킹에서 단백질 유연성(flexibility)을 고려하지 않으면 잘못된 결과를 초래할 수 있습니다. 단백질 유연성은 아미노산 잔기(residue)의 작은 움직임부터 단백질 구조의 더 큰 변화까지를 의미할 수 있습니다. Rigid 도킹에 영향을 주는 단백질 conformation의 적절한 변화에 대한 예시는 kinase에서 찾아볼 수 있습니다. 결합한 억제제에 따라 DFG-loop는 최소 두 가지 conformation 즉, DFG-In과 DFG-Out conformation으로 관찰될 수 있습니다. 이 변화를 고려하지 않으면 모든 화합물에 대해 활성과 관련 없는 도킹 결과를 생성할 수 있습니다.
Cambell 등1은 분자동역학을 이용하여 단백질 유연성을 샘플링하고 trajectory를 클러스터링하여 단백질 conformation들의 앙상블을 생성하는 방법을 보고했습니다. 이들은 리간드를 단백질 conformation들의 앙상블에 도킹(이 때, 각 단백질 conformation은 고정으로 처리)하여 간접적으로 단백질 유연성을 도킹 프로토콜에 포함시켰습니다. Cyclin-dependent kinase 2와 factor Xa를 예시로 들어 자가 도킹과 교차 도킹 실험을 수행했습니다. 이 접근 방법은 주어진 리간드에 대해 정확한 결합 모드를 가진 가장 낮은 스코어의 포즈를 확인하기 위해 개발되었습니다.
우리의 접근 방법의 유효성을 검증하기 위해 CDK2와 Factor Xa 데이터세트2를 이용했습니다. 모든 예는 Flare를 이용하여 X-선 단백질 구조를 준비하고 분자동역학 시뮬레이션을 수행했습니다. 시뮬레이션 trajectory를 클러스터링하여 고유 단백질 conformation들의 앙상블을 생성하고 이를 Flare 앙상블 도킹에 이용했습니다. MD trajectory의 클러스터링은 Flare 6.1버전부터 제공되는 새로운 기능입니다. 이 워크플로는 각 리간드의 도킹을 위한 최상의 단백질 conformation을 확인하고, 도킹 워크플로에 단백질 유연성을 포함시킬 수 있습니다.
Flare는 사용자 친화적인 인터페이스를 제공하며, 파이썬 API도 포함합니다. 사용자는 Cresset Engine BrokerTM의 분산 계산 기능을 통해 프로그래밍 방식으로 워크플로를 실행하거나 자동화할 수 있습니다. 프로토타입 워크플로에서 우리는 본 연구를 실현하기 위해 파이썬 스크립트를 개발했습니다.
방법
단백질 구조 준비
우리는 모든 단백질 구조를 Flare의 protein preparation 도구를 이용하여 준비했습니다. 구조를 다운로드하고, 아미노산 곁사슬의 대체 conformation들을 확인하고, 곁사슬의 공간 점유를 이용하거나 곁사슬이 형성한 접촉에 기반하여 선호하는 곁사슬 회전 이성질체(rotamer)를 선택했습니다. 이들은 Flare의 자동화된 protein preparation 도구를 이용하여 준비되었습니다. 이 워크플로에서는 수소 원자 추가, 이온화될 수 있는 잔기의 tautomer 상태 탐색, 누락된 곁사슬 추가, 두 아미노산 간의 틈(gap) 채우기, 말단 잔기 캡핑, 아미노산 잔기 간의 상호작용과 내부 수소결합 네트워크의 최적화 등이 수행됩니다. 대부분의 경우, Flare의 자동화된 protein prep은 도킹, 에너지에 관한 연구, 분자동역학 연구에 알맞은 설정을 지원하고 있지만 여전히 세심한 주의가 권장됩니다.
분자동역학
리간드 주변 결정학적 물 분자는 그대로 유지한 상태로 Flare MD 시뮬레이션에 이용했습니다. MD 파라미터 대부분은 Flare의 기본값을 사용했습니다. 저분자의 전하 부여 및 파라미터화는 AM1-BCC와 OpenFF 2.0을 각각 이용하였습니다. 단백질의 전하 부여 및 파라미터화는 amber14ffsb를 적용했습니다. Hydrogen-Mass Repartitioning(HMR)은 4fs 시간 간격(time step)을 이용했습니다. 시뮬레이션은 100ps 평형화(equilibration) 후 4ns 동안 수행했습니다. 전체 trajectory에서 2,000개의 프레임(프레임당 2ps)을 얻었습니다. 기본 설정을 이용 시, 사용자는 Flare 애플리케이션에서 몇 번의 클릭만으로 MD를 실행할 수 있으며 4ns 시뮬레이션은 단일 GPU가 장착된 PC에서 1시간 이내에 완료됩니다.
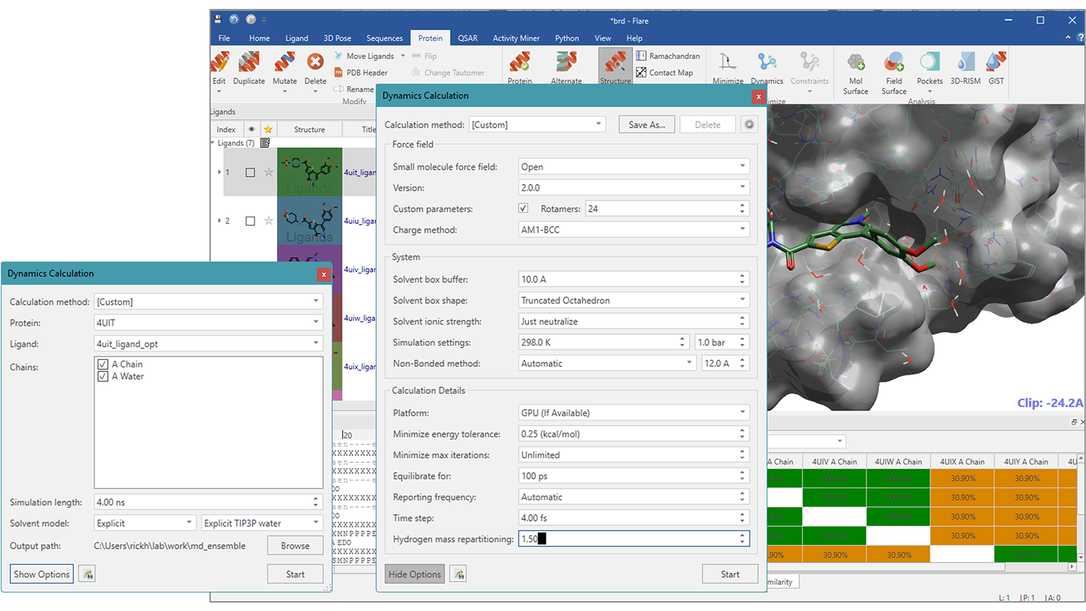
그림 1. Flare의 분자동역학 설정
Trajectory의 클러스터링
활성 부위는 공결정(co-crystal) 구조의 리간드를 기준으로 6 Å 거리 이내의 잔기들로 정의하였습니다. 활성 부위 잔기의 중원자(heavy atom)의 좌표 벡터를 질량 가중 평균 연결 RMSD 기반 클러스터링 알고리즘에 이용했습니다1,2. Cambell 등은 앙상블을 만들기에 충분한 클러스터의 수가 6 ~ 8개라고 언급하지만, 여기서는 trajectory로부터 20 개의 대표 conformation을 샘플링하기 위해 20 개의 클러스터를 취했습니다. 클러스터링은 Amber 도구 중 일부분으로 제공되는 cpptraj 함수를 이용하여 대체할 수도 있지만 이 예비 실험에서는 다양한 옵션을 탐색하고 클러스터링 결과를 최적화하기 위해 파이썬으로 클러스터링을 구현하기로 결정했습니다.
클러스터 간 시간 의존 전이를 그림 2에 나타내었고 활성 부위 내 중원자의 요동(fluctuation)을 RMSD로 추적했습니다. 그림 2의 플롯 색은 클러스터 멤버십에 의한 것이고 그래프의 노드 크기는 빈도에 해당하며 유방향 간선(directed edge)의 길이는 전이 빈도에 해당합니다(빈도가 높을수록 더 짧음). 이것은 분자동역학에서 단백질 구조 공간의 루프와 진행(progression)을 보여줍니다(명확성을 위해 매우 빈도 높은 자가 루프(self-loop)는 생략함). 평균 연결은 클러스터의 사이즈를 균등하게 분할하지 않고 전체 클러스터 내 RMSD 기반 분산(variance)이 클러스터의 사이즈에 상관없이 medoid를 식별하기 위해 계산됩니다. 이들의 medoid conformation은 모두 trajectory의 특정 스냅 사진입니다. 따라서, MD 프로세스에서 사용한 것과 동일한 force field를 이용하여 최적화 계산을 하는 것을 권장합니다. 우리는 활성 부위 가까이의 환경에 관심이 있기 때문에 리간드에서 8 Å 거리 내의 원자들의 최적화 계산을 모든 스냅 사진 구조에 대해 수행했습니다.
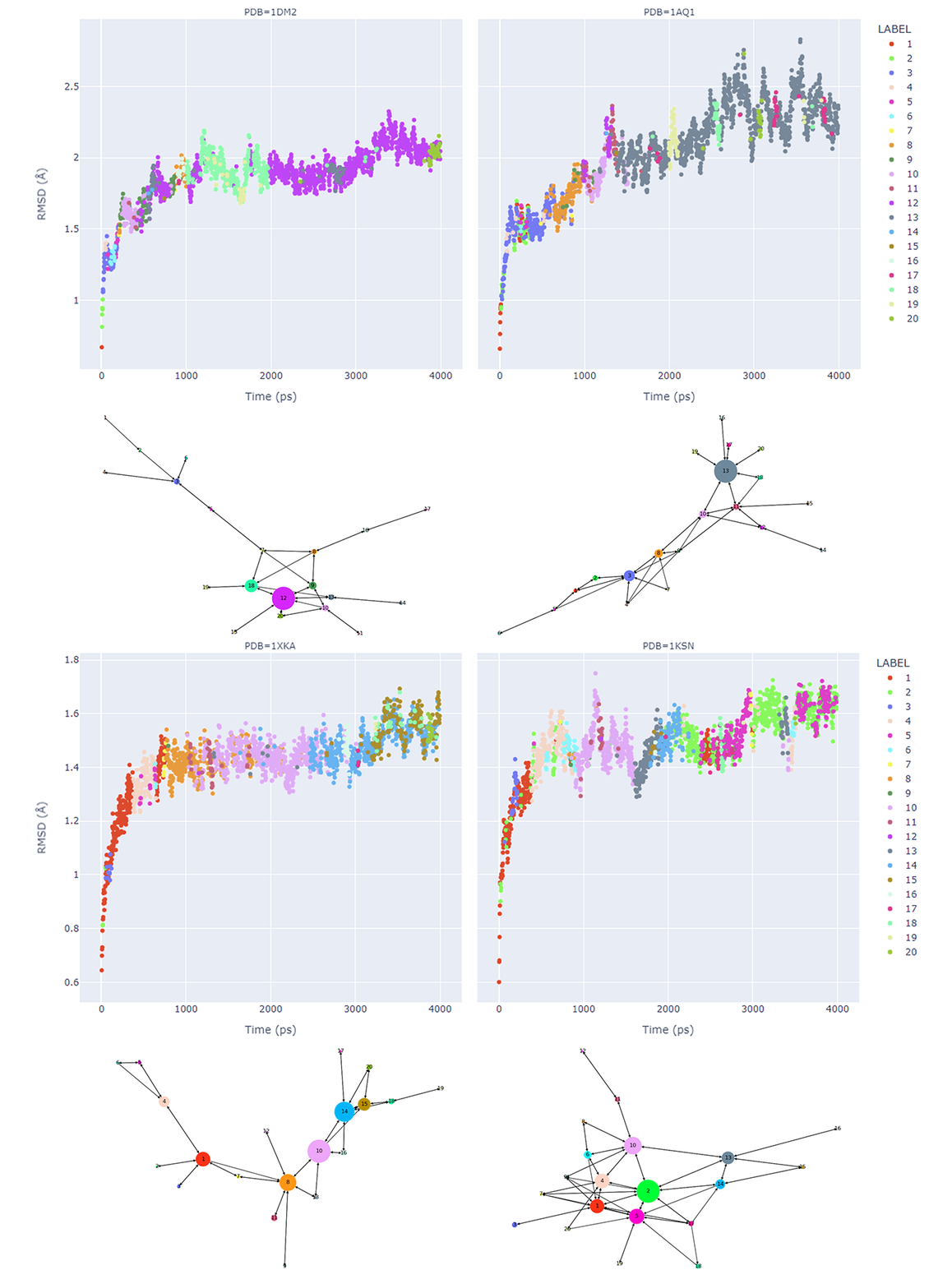
그림 2. 단백질 구조 1DM2, 1AQ1, 1XKA, 1KSN에 대한 MD trajectories의 분석, 시작 좌표 대비 RMSD 값을 보여줍니다. Trajectories가 클러스터링 되면 그 결과는 유방향 그래프로 보여집니다. 색상은 클러스터 노드와 클러스터 멤버십에 대해 각 예제 간에 일관되게 적용됩니다. 노드 사이즈는 클러스터의 멤버 수에 해당하고 간선의 길이는 전이 빈도를 나타냅니다(빈도가 높을수록 더 짧음). 이는 단백질 구조 공간의 루프와 진행을 보여줍니다(명확성을 위해 매우 빈도 높은 자가 루프는 생략함).
도킹 계산
모든 도킹 계산은 Flare에 탑재된 Lead FinderTM 알고리즘3을 이용했습니다. 각 선택된 단백질 conformation(클러스터 medoids와 기준 X-선 구조)의 도킹 실행은 리간드 세트에 걸쳐 반복됩니다. 전체 도킹 프로세스에서 결합 부위에 대한 적합성을 나타내는 스코어와 함께 리간드 포즈를 생성했습니다. Lead Finder는 다양한 스케일링 된 에너지 기반 스코어(dG, VSscore, Rank Score)를 제공합니다. 우리는 그 중에서 자가 및 교차 도킹 실험에서 예측한 리간드 포즈를 비교하기 위해 결정학적 리간드 포즈와 dG 스코어를 식별하도록 최적화한 Rank Score를 이용했습니다.
Flare의 앙상블 도킹 워크플로를 이용하면 하나의 단백질에 대한 다양한 conformation에 도킹할 수 있습니다. 우리는 Flare Python API를 이용하여 도킹 실험을 실행하는 pyflare 스크립트를 작성하고, 모든 단백질에 획일화된 제약을 추가하는 등 GUI에서 이용가능한 실험의 모든 설정을 조정하고, 결과를 사용자 맞춤 형식으로 작성했습니다. 높은 점수를 받은 모든 포즈를 앙상블 + X-선 단백질에 걸쳐 하나의 sdf 파일에 작성했습니다. 이를 통해 사용자는 Jupyter 노트북과 같은 다른 외부 도구를 이용하여 빠르게 분석을 진행할 수 있습니다.
본 연구의 프로토콜을 그림 3에 요약했습니다. 노란색으로 강조 표시한 단계는 Flare에서 수행되며, 클러스터링 프로세스는 파이썬에서 작성되고 pyflare API로 실행됩니다.
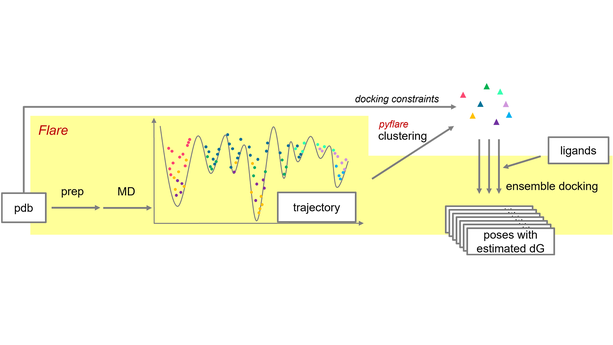
그림 3: Flare/pyflare를 이용한 MD/앙상블 도킹 워크플로
결과
MD 앙상블 도킹의 유효성 검증(verification)
Cambell 문헌에 설명한 바와 같이, 우리는 두 리간드 세트에 대한 자가 도킹과 교차 도킹을 통해 프로토콜의 유효성을 검증하였습니다: PDB 구조 1DM2와 1AQ1의 두 CDK2 리간드(그림 4 – 좌); PDB 구조 1XKA와 1KSN의 두 Factor Xa 리간드(그림 4 – 우). CDK2 리간드는 결합된 리간드의 큰 크기 차이의 예를 보여줍니다. 대신에 Factor Xa 리간드는 매우 유연합니다. 이 두 경우에서 교차 도킹은 보통 실패할 수 있습니다.
CDK2 예(그림 4 – 좌)의 경우, 결정학적 리간드 HMD(1DM2.pdb)와 STU(1AQ1.pdb)는 모두 1 Å RMSD 이내로 각각의 X-선 단백질 구조에 자가 도킹 되었습니다. 1AQ1.pdb에 대한 HMD 리간드의 교차 도킹은 RMSD = 1.554 Å로 상당히(reasonably) 성공적이었습니다. 그러나, 1DM2.pdb에 대한 STU의 교차 도킹은 큰 리간드 사이즈와 포켓 사이즈의 차이로 인해 실패했습니다.
각 단백질 구조의 MD 스냅 사진들의 앙상블에 대한 CDK2(HMD와 STU)의 자가 도킹 실험에서도 예상대로 좋은 결과를 얻었습니다. 이전에 실시한 직접적인 교차 도킹 실험과는 달리, MD 스냅 사진들의 앙상블을 이용한 교차 도킹 실험은 성공적이었습니다. 기준 X-선 리간드와 비교했을 때, 그 결과는 RMSD < 2 Å(STU 1.255 Å, HMD 1.654 Å)인 도킹된 포즈를 생성했습니다. 허용 가능한 RMSD 요건 외에 도킹된 포즈를 육안으로 검사하여 결정학적 포즈와 유사한지 확인했다는 점에 주목할 필요가 있습니다.
매우 유연한 리간드와 Factor Xa의 더 어려운 예의 경우, native 단백질 구조에 대한 리간드 4PP(1XKA.pdb)와 FXV(1KSN.pdb)의 자가 도킹은 성공적이었습니다(그림 4 – 우). Factor Xa의 교차 도킹 실험은 성공적이지 못했습니다. 두 리간드 모두 좋지 못한 도킹 스코어와 함께 RMSD > 2 Å인 포즈를 생성하였습니다. 이들 포즈의 육안 조사를 통해 그 결과는 어떤 리간드도 결정학적 포즈를 식별하지 못했음을 확인했습니다.
각 단백질 구조의 MD 스냅 사진들의 앙상블에 대한 리간드 4PP와 FXV의 자가 도킹 실험은 결정학적 단백질에 대한 rigid 도킹에 비해 약간 개선된 RMSD 값을 생성했습니다. MD 스냅 사진들의 앙상블을 이용한 Factor Xa의 교차 도킹의 경우, 자가 도킹 실험에서 관찰된 값에 가까운 LF dG 스코어와 허용 가능한 범위(RMSD FXV 1.385, 4PP 1.498) 내의 포즈를 생성할 수 있었습니다.
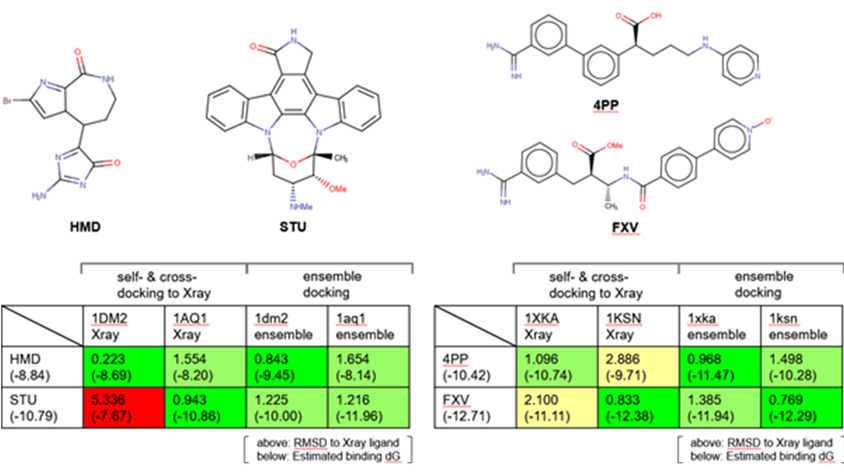
그림 4. MD/앙상블 도킹의 유효성 검증. 좌: CDK2 리간드의 자가 도킹 및 교차 도킹 결과. 우: Factor Xa 리간드의 자가 도킹 및 교차 도킹 결과.
종합적으로, CDK2와 Factor Xa 예의 결과는 Flare의 MD 클러스터링 앙상블 도킹 방법이 도킹 워크플로에 단백질 유연성을 고려할 수 있는 단백질 conformation들의 앙상블을 성공적으로 생성할 수 있음을 보여줍니다. 이 예에서 활성 부위의 차이는 주로 활성 부위의 곁사슬 운동으로 관찰되므로 여기서 이용한 비교적 짧은 4ns MD 시뮬레이션은 이들 변화를 샘플링하기에 충분합니다. 단백질 conformation의 더 큰 변화는 앙상블 도킹에 적절한 conformation들의 앙상블을 생성하기 위해 더 긴 시간의 MD 시뮬레이션을 필요로 할 수 있습니다.
결론
이 연구에서 우리는 단백질 유연성을 고려한 도킹 실험을 수행하기 위해 분자동역학과 Flare의 앙상블 도킹 워크플로를 이용한 접근 방법을 소개했습니다. 이 접근법은 이용가능한 단백질 구조가 아미노산 곁사슬의 운동 또는 더 큰 변화로 나타날 수 있는 리간드의 생물학적으로 관련 있는 conformation이 아닐 때 특히 유용합니다.
여기에서 이용한 MD 클러스터링은 Flare 6.1버전부터 제공되는 새로운 기능입니다. 이 방법은 도킹 실험에 단백질 유연성을 고려하기 위해 다양한 단백질 conformation을 생성할 수 있으므로 MD 시뮬레이션의 유용한 분석 방법입니다.
이 연구에서 우리는 단백질 구조 준비, 앙상블 도킹, 결과 분석의 전 과정을 자동화하는 파이썬 스크립트를 작성했습니다. 이들 스크립트의 저장 및 실행은 Jupyter 노트북을 이용했습니다.
참고문헌
- A.J. Cambell et al., J. Chem. Inf. Model. (2013)
- J. Shao et al., J. Chem. Theory Comput. (2007)
- Lead Finder, 2112 build 1, BioMolTech®, Toronto, Ontario, Canada