Stuart Firth-Clark†
†Cresset, New Cambridge House, Bassingbourn Road, Litlington, Cambridgeshire, SG8 0SS, UK
성공적인 가상 스크리닝 계획 및 실시의 열쇠는 구매할 화합물을 효과적으로 결정하기 위해 검토 중인 타겟에 대한 이용 가능한 모든 정보를 활용하고 가능한 한 다양한 chemical space를 탐색하는 것입니다. 본 사례에서는 BlazeTM를 이용한 리간드 기반 가상 스크리닝과 FlareTM를 통해 얻은 단백질 구조 정보를 함께 활용하여 FOXO 펩타이드의 대체가 될 수 있는 저분자 물질을 탐색합니다.
서론
신약 개발 프로젝트에서 생체 분자 구조를 표적으로 하는 접근은 관여된 결합 파트너의 종류에 따라 다릅니다(그림 1). 가장 일반적인 접근은 알려진 modulator의 결합 부위를 타겟으로 하여 같은 부위에서 경쟁하는 리간드를 도입하는 것입니다. 그러나, 생체 분자 중에는 명확한 저분자 modulators를 갖지 않고 핵산(DNA 및 RNA) 또는 단백질과 같은 다른 생체 분자와 상호 작용하는 것도 많이 있습니다. 이러한 계의 경우, 단백질 – 단백질 상호 작용(PPI)은 보통 두 단백질의 넓은 표면적에 걸쳐 발생합니다. 두 분자 사이의 상호 작용 에너지는 결합 부위와 같은 작은 영역에 집중되기보다는 큰 표면상의 서로 다른 많은 약한 contact에서 얻습니다. 큰 단백질에 강하게 결합할 만큼 충분한 상호작용을 할 수 없는 저분자의 경우에는 이러한 상호작용을 타겟으로 하는 것이 일반적으로 어렵습니다.
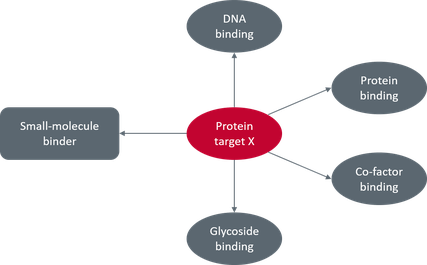
그림 1. 표적 단백질 X의 결합 파트너.
그러나, PPI 계면의 작은 특정 영역이 전체 결합 에너지 중 큰 비중을 차지하는 경우도 있습니다. 실험실에서는 작은 펩타이드 프로브를 이용하여 넓은 범위 안에서 더 중요한 결합 영역을 찾으려는 연구도 활발히 진행되고 있습니다. 결합 전체 중 기여가 비교적 높고 부피적으로 작은 영역을 찾을 수 있다면 그 영역을 표적화하는 것이 가능할 것으로 생각됩니다.
표적 영역이 특정되면 다음 단계로 본래 펩타이드를 ‘모방’할 가능성이 있는 저분자를 탐색하는 것입니다. 이에 대한 접근으로는 가상 스크리닝이 있으며, 결합 영역의 정보를 활용하여 표적 영역과 선호되는 상호작용이 가능한 저분자를 탐색합니다. Cresset사의 가상 스크리닝 플랫폼인 Blaze를 이용하여 쿼리 펩타이드의 정전기, 소수성, 반데르발스 특징과 유사한 특징을 갖는 시판 화합물 탐색을 아래에 설명합니다.
Forkhead box proteins(FOXO)의 신규 저분자 선도물질의 탐색
FOXO는 N-말단 DNA-결합 도메인을 갖는 전사인자(transcription factor)로 14-3-3 단백질과 같은 다양한 단백질 파트너를 인식합니다. FOXO1 경로의 파괴(disruption)는 당뇨병 등의 대사성 질환을 일으킬 수 있으며, FOXO3는 다양한 종양 개체(tumor entities)와 관련이 있으며 종양 혈관신생(angiogenesis)을 촉진하고 세포자멸사(apoptosis)에 관여하고 있습니다. 그러므로, FOXO 단백질 군(family)은 신약 개발 프로젝트에서 표적으로서 좋은 생체 분자 세트로 보이지만 이들의 주요 상호 작용은 DNA 등의 다른 생체 분자와의 상호 작용이므로 연구하기에 더욱 복잡한 시스템 중 하나입니다.
본 프로젝트의 첫 번째 단계는 FOXO 단백질과 관련된 데이터를 수집하는 것이었습니다. 알려진 저분자는 발견되지 않았지만, FOXO는 전사인자 단백질이므로 DNA와 결합합니다. UniProt을 통해 적절한 단백질 서열을 검색한 후, FOXO 단백질 군 중에서 이용 가능한 PDB 구조를 확인하였습니다.
DNA-결합 도메인을 표적으로
FOXO 단백질과 DNA 간의 상호작용을 나타내는 구조들을 발견했습니다. Cresset의 신약 개발 플랫폼인 Flare 상에서 FOXO 단백질 군 중 이용 가능한 DNA-결합 도메인 결정 구조들을 정렬(align) 및 중첩(superimpose)할 수 있었습니다(그림 2a). 중첩을 완료한 후, 작은 리간드 크기의 분자가 FOXO에 결합할 때 활용할 수 있는 특정 상호 작용이 거의 없음을 확인하였으며, 모든 잠재적 결합 부위의 크기가 매우 제한적이었습니다(그림 2b). 따라서, 다른 접근방식을 고려해야 했습니다.
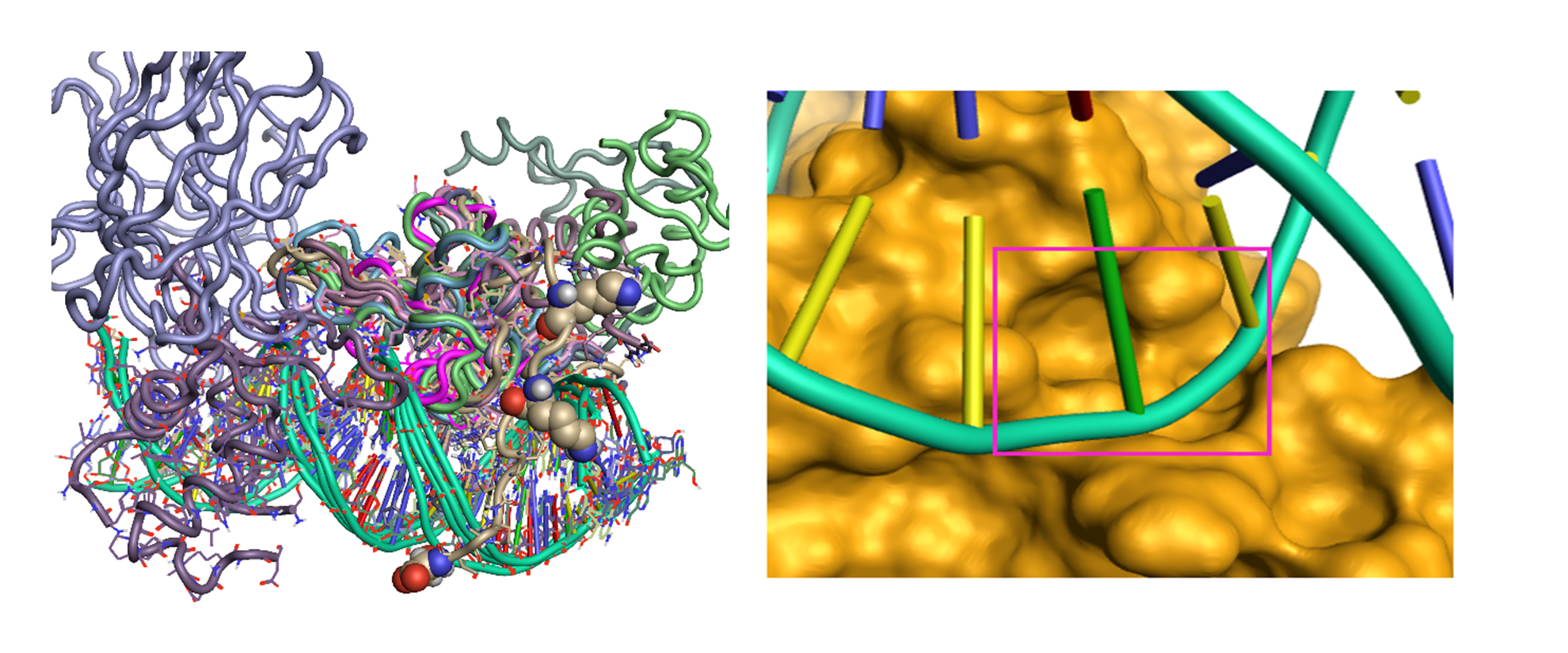
그림 2. (a). Flare에서는 결정 구조를 다운받고, 시퀀스를 정렬한 후 단백질을 중첩할 수 있습니다. (b). 상대적으로 작은 공동(cavity)은 가상 스크리닝 연구를 실시하는데 적합하지 않습니다.
14-3-3 인식 부위를 표적으로
FOXO 단백질은 또한 14-3-3 단백질의 결합 파트너이며, 14-3-3 단백질의 인식 부위는 FOXO 단백질의 N-말단 근처에 위치하고 있습니다. 14-3-3 단백질은 주로 이합체이며 phospho-peptide를 인식하여 FOXO를 억제합니다. PDB 구조 6QZR에서는 FOXO의 phospho-peptide fragment가 14-3-3 단백질에 결합되어 있고(그림 3), 이 구조를 기반으로 가상 스크리닝 전략을 고안하는 것이 가능합니다.
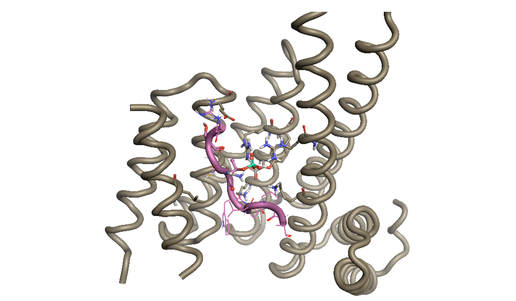
그림 3. FOXO1 (핑크)의 phospho-peptide fragment가 14-3-3 단백질(밝은 갈색)에 결합된 6QZR 결정 구조.
목표는 Blaze를 이용하여 14-3-3 단백질의 FOXO 결합 부위를 표적으로 하되, 결정 구조에 존재하는 FOXO 펩타이드의 구조적 특징을 고려하여 이를 수행하는 것이었습니다. 이를 위해, FOXO 펩타이드의 중앙 4개의 잔기(residue)에서 중요하다고 생각되는 특징 – 즉, 양전하 영역(빨간색 구), 음전하 영역(파란색 구), 소수성 특징(주황색 구) 및 형상 특징(노란색 구)을 추출하였습니다(그림 4).
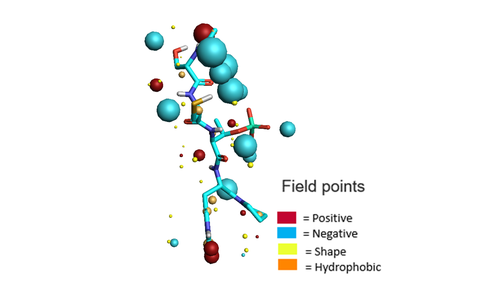
그림 4. FOXO 펩타이드의 분자적 특징을 설명하는 field point.
Blaze 인터페이스(웹 기반 포털 또는 Flare를 통한 엑세스 가능)는 pharmacophore 또는 field-point constraint를 도입하여 리간드 탐색의 가이드를 제공할 수 있습니다. 이것은 특정 상호작용이나 기능적 구조를 중요시하는 경우에 유용합니다. Blaze에서 가상 스크리닝을 실시할 때 특정 공급자 또는 분자량과 같은 속성을 추가하여 탐색에 이용할 상용 화합물 데이터베이스를 제한할 수 있습니다. 현재, Cresset Blaze collection에는 탐색에 이용할 수 있는 화합물 수로 약 2,000만 개가 등록되어 있습니다(그림 5).
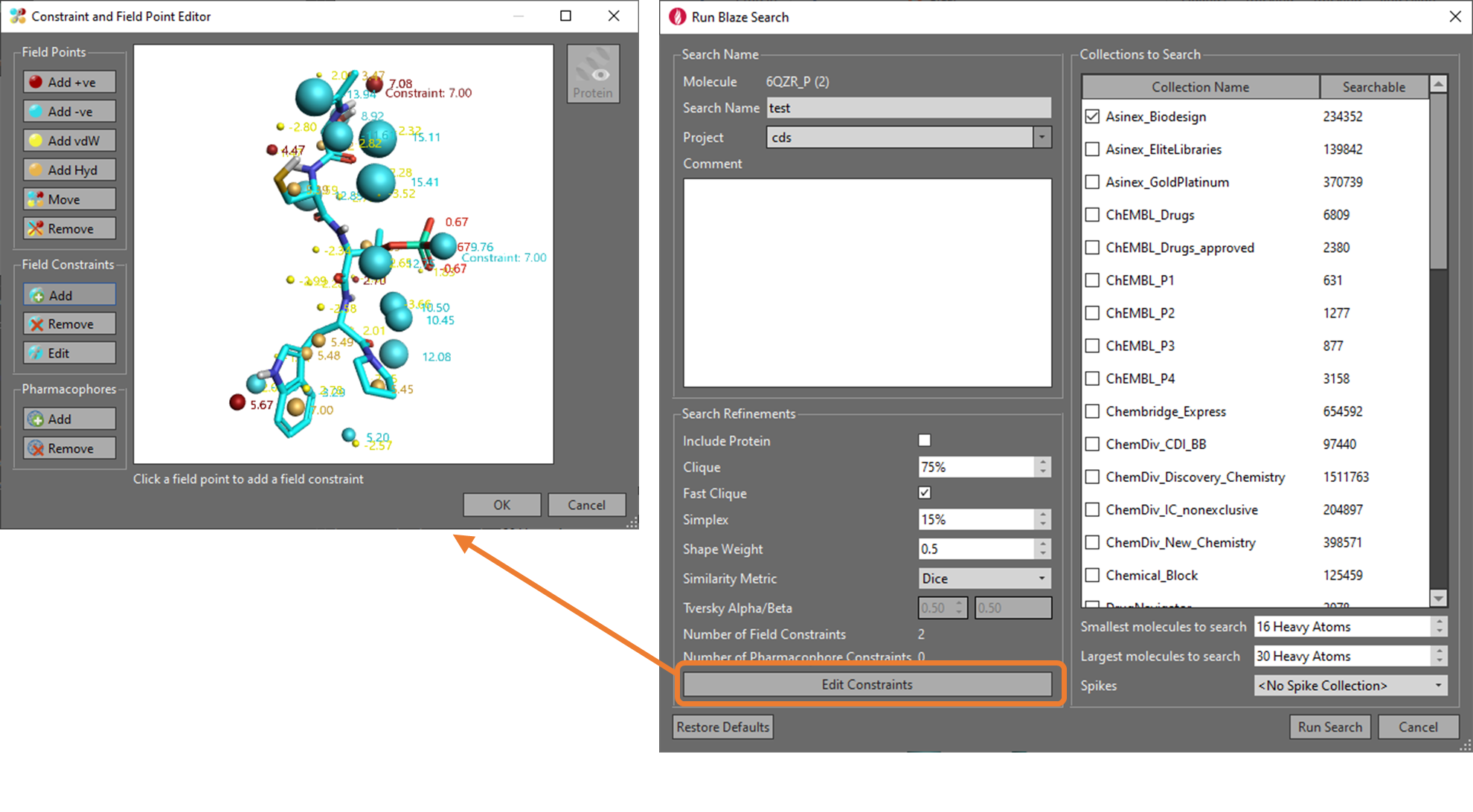
그림 5. Flare 인터페이스에서 Blaze의 입력 파라미터를 설정할 수 있습니다. 예를 들어, field 및/또는 pharmacophore constraint, 공급자 데이터베이스 정의 및 분자 크기 제한.
Top-scoring 히트 화합물을 Flare로 내려 받았고, 14-3-3 수용체와 함께 표시했습니다. 14-3-3 단백질 구조는 가상 스크리닝 시 직접적으로 이용되지 않았지만, 쿼리 자체는 6QZR 결정 구조로부터 얻은 것이므로 가상의 히트 분자는 14-3-3 단백질과 동일한 레퍼런스 프레임에 위치했습니다.
Top-scoring 히트 물질을 Flare로 내려 받은 후에는 추가 후처리 및 분석을 수행할 수 있었습니다(그림 6). 구체적으로, Pose 변형이 큰 분자를 육안으로 제거하거나 14-3-3 단백질에 도킹을 실시하고 신규 리간드의 결합 부위와의 정전기적 상보성을 조사할 수 있었습니다. Flare에서는 TPSA, logP 또는 flexibility와 같은 물리적 특성에서 이상적인 범위를 갖는 분자를 식별할 수도 있는데, 상기의 특성들에 대해 계산된 값이 적절한 분자만이 선택되도록 할 수 있습니다. 가상 스크리닝 실험의 결과 화합물을 구매할 때는 chemical space가 충분히 샘플링 되었는지 확인하는 것이 중요합니다. Flare에서는 V5 릴리즈부터 화합물 선택에 도움을 드릴 수 있도록 결과를 클러스터링할 수 있는 기능을 지원하고 있습니다.
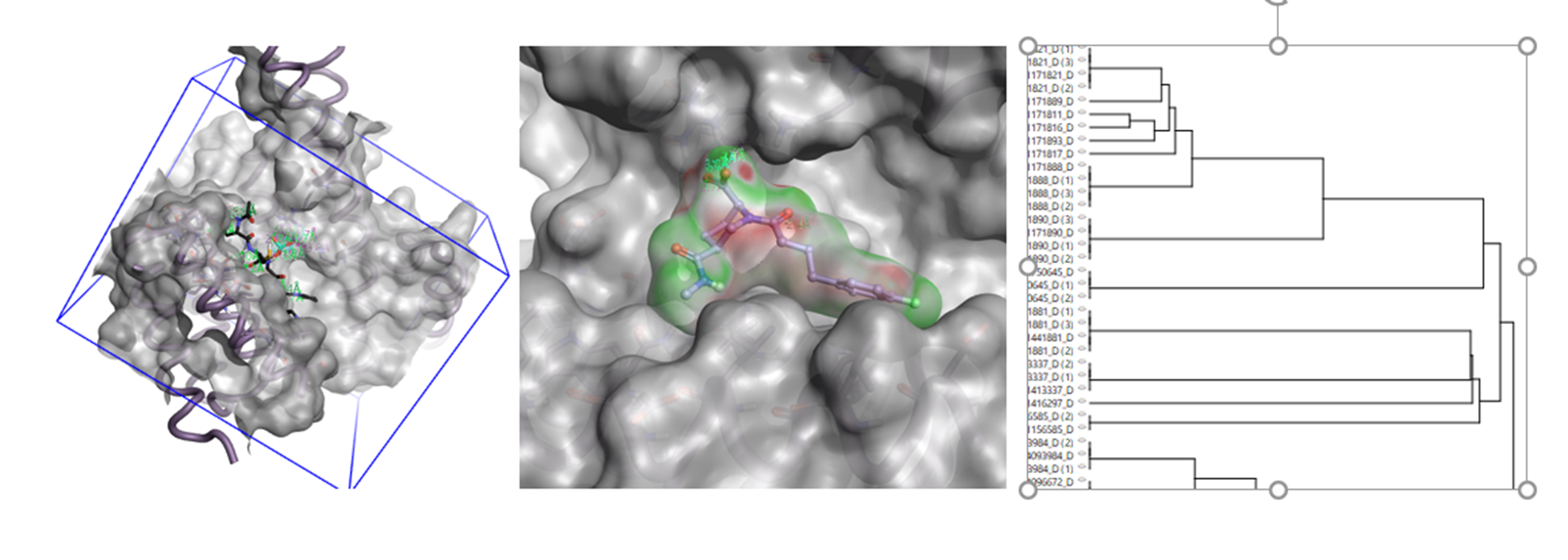
그림 6. Blaze 가상 스크리닝에서 얻은 화합물들의 선택을 가이드 하기 위한 Flare 기능의 그래픽 표현. (a) Docking, (b) Electrostatic ComplementarityTM, (c) Clustering.
그림 7은 Blaze의 탐색 결과의 예를 나타낸 것입니다. 왼쪽 최상단의 분자(1)은 쿼리 펩타이드입니다. 대부분의 예는 acidic기를 이용하여 phosphate 인식 부위와 상호 작용하고 있지만 일부 분자(6,9)는 그러하지 않습니다.
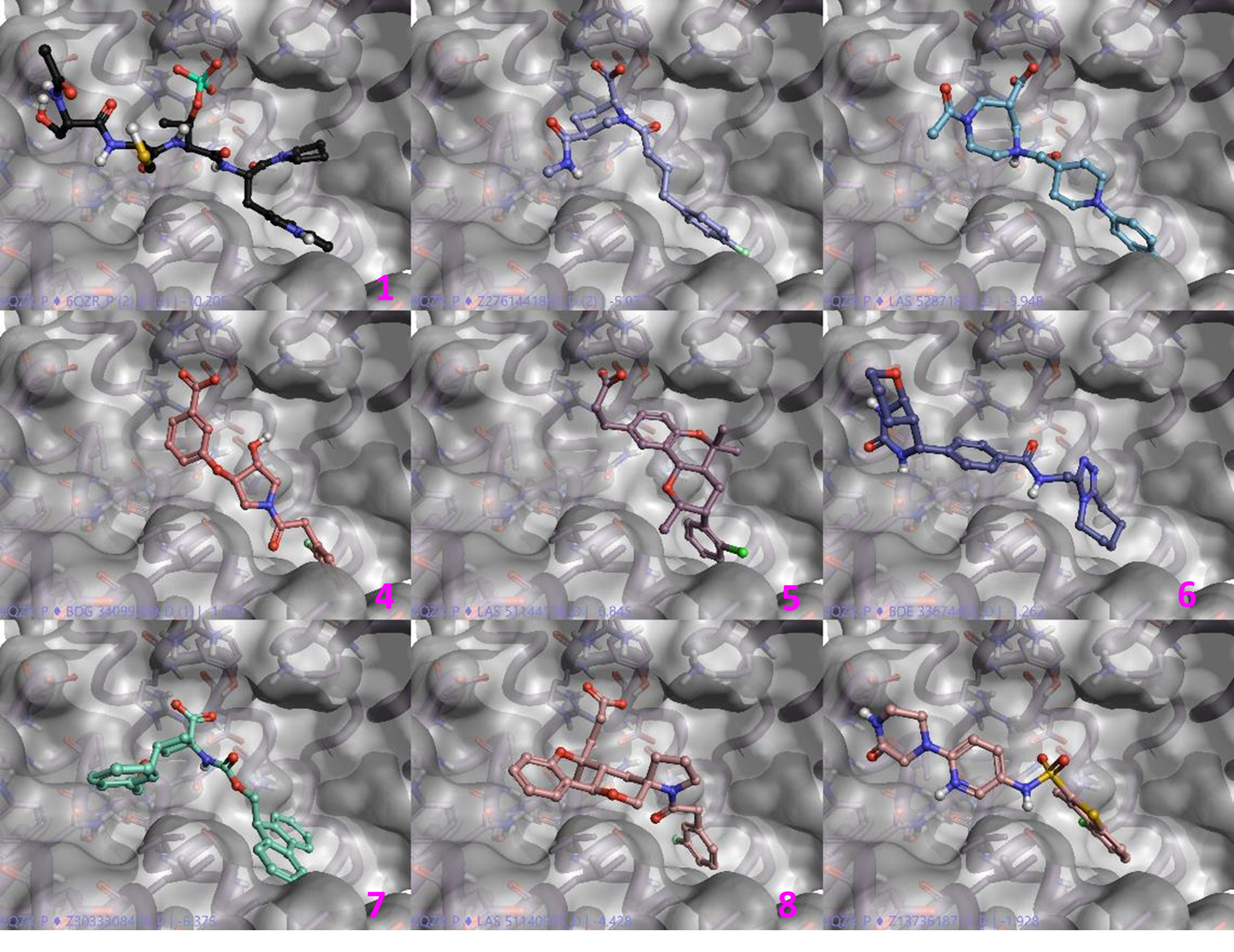
그림 7. Flare 상의 Blaze 가상 스크리닝 결과의 일부.
결론
리간드 기반 접근과 구조 기반 접근을 조합함으로써 이용가능한 모든 표적 정보를 활용할 수 있는 우수한 가상 스크리닝 전략을 설계할 수 있습니다. 잘 설계된 탐색에서는 핵산(DNA 및 RNA) 또는 단백질과 같은 다른 생체 분자와 상호작용하는 보다 도전적인 표적에 대한 저분자 히트를 발굴하는 것도 가능해집니다.