Nathan Kidley†
†Cresset, New Cambridge House, Bassingbourn Road, Litlington, Cambridgeshire, SG8 0SS, UK
초록
시장에 출시되었거나 개발 중인 새로운 공유 결합 화합물의 수는 의약 화학이 표적 공유 결합 억제제에 대한 관심을 다시 불러일으켰음을 시사합니다. 본 사례에서는 Bruton’s Tyrosine Kinase(BTK) 억제제 Evobrutinib1의 발견을 예로 들어 FlareTM,2의 공유 결합 도킹 워크플로가 공유 결합 화합물의 효율적인 설계 및 우선 순위 매김에 어떻게 활용될 수 있는지 소개합니다.
서론
Evobrutinib은 최근에 다발성 경화증 치료에 사용되는 BTK 공유 결합 억제제로 우수한 kinase 선택성(특히, EGFR 및 Tec 군 kinase에 대한)과 유망한 ADME 특성을 보여줍니다.
Merck사는 PDB:3GEN(그림 1)의 활성 부위에 결합한 BTK 경쟁 억제제 B43의 단백질 구조를 Evobrutinib 발견의 시작점으로 사용했습니다. 본 사례 연구에서는 해당 구조를 예로 들어 Flare의 공유 결합 도킹 워크플로가 화합물의 효율적인 설계 및 우선순위 매김에 어떻게 활용될 수 있는지 소개합니다.
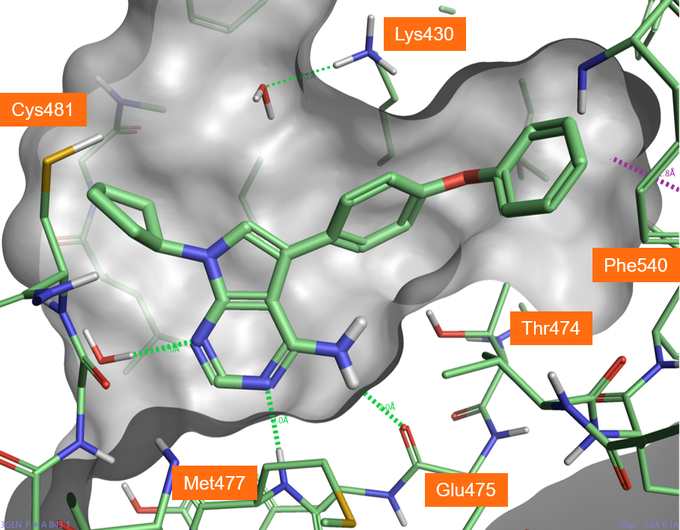
그림 1. BTK 활성 부위에 결합한 B43 화합물의 결정 구조(PDB: 3GEN), 수소 결합은 녹색 점선으로 표시하였음.
Flare 공유 결합 도킹 모듈
공유 결합 도킹은 Lead FinderTM의 도킹 알고리즘3을 이용하여 공유 결합 억제제의 binding pose 및 상호작용을 예측하는 Flare 모듈 중 하나입니다. Flare에서 공유 결합 도킹 실험을 하려면 리간드는 반드시 적절한 반응성을 갖는 친전자성 형태(공유 결합 warhead라고도 함)이어야 합니다. 이들은 화학 반응성이 낮은 친전자성기로, 리간드가 표적 단백질에 비공유적으로 결합할 때 활성 부위 내 특정 친핵성 잔기 근처에 위치 후 빠르게 반응하여 결합을 형성합니다. 그림 2 – 좌는 Flare에서 지원하는 공유 결합 warhead를 나타낸 것입니다. 또한, 사용자 정의를 통해 공유 결합 warhead를 쉽게 확장할 수 있습니다.
공유 결합 도킹 실험을 시작하려면 활성 부위 내 친핵성 아미노산(Cys, Lys, Ser 또는 Tyr 중 하나)를 반드시 지정해야 합니다.
그런 다음, 공유 결합 도킹 워크플로는 입력 화합물의 화학적 변형(즉, 공유 결합 warhead를 변형하여 유사-공유 결합 리간드 생성)을 수행합니다. 이때, 단백질과 공유 결합을 형성하지 않고 양성자화된 상기 아미노산의 친핵성 원자에 결합합니다(그림 2 - 우).
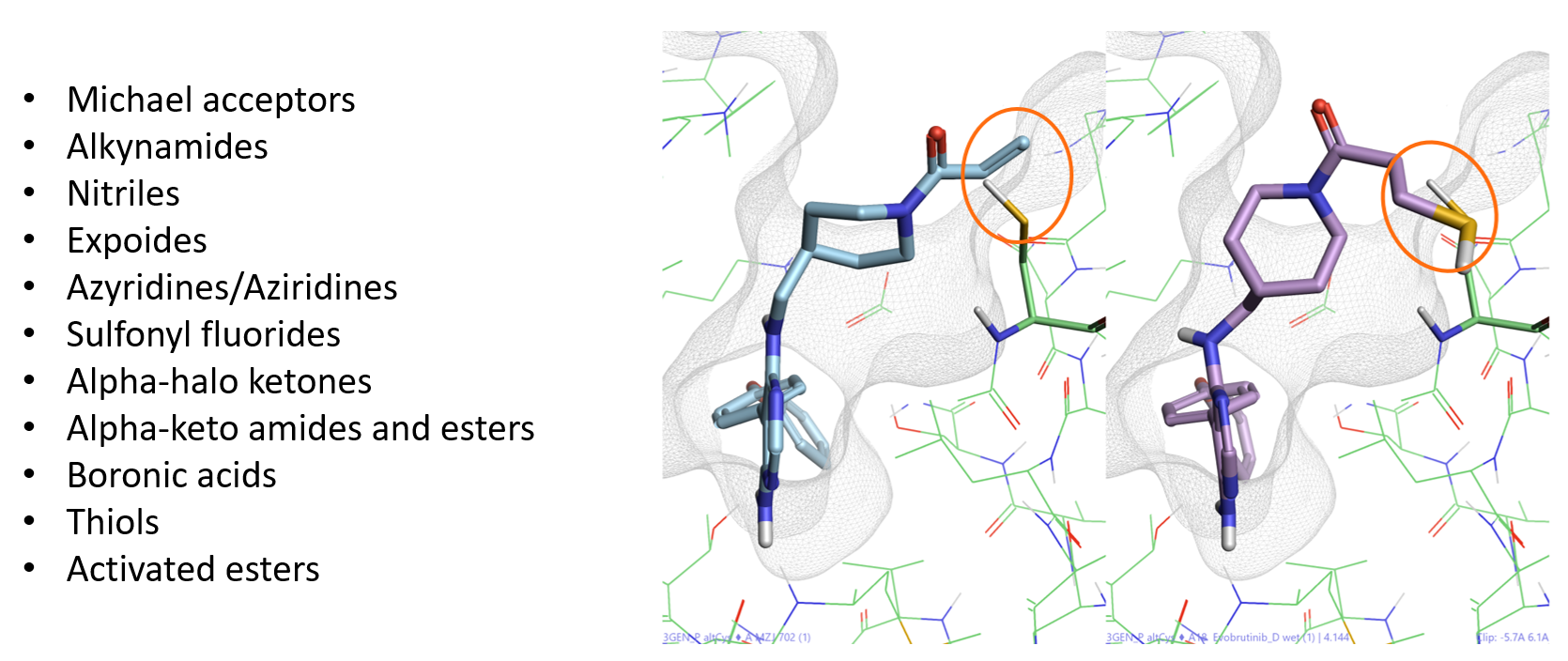
그림 2. 좌: Flare에서 지원하는 공유 결합 warhead. 우: Cys481에 공유 결합 도킹 시 Evobrutinib의 화학적 변형.
단백질, 리간드 및 도킹 준비
Flare에서 PDB:3GEN 단백질-리간드 결정 구조를 다운로드한 후, 수소 원자 복원, 수소 결합 네트워크 최적화, 원자 충돌 회피 및 적절한 양성자화 상태 옵션을 적용하여 구조를 준비하였습니다. 준비한 계가 더 유리한 리간드-단백질 수소 결합 상호작용을 생성하는 동시에 다른 BTK 단백질 구조와 일관성을 가질 수 있도록 Thr474 hydroxyl기를 수동으로 회전시키는 최적화를 더 진행하였습니다.
PDB:3GEN 단백질 구조(Caldwell 등1의 연구에서 Evobrutinib 발견의 시작점)는 공유 결합 리간드와 결합하지 않기 때문에 표적 친핵성 아미노산인 Cys481 곁사슬은 공유 결합을 형성하기에 적절한 conformation이 아닐 수 있습니다. 이러한 잠재적인 문제를 조사하고자 단백질 구조를 복제하고 Cys481에 대한 대체 회전 이성질체를 선택한 다음 최적화 작업을 수행하였습니다. 두 단백질을 이용하여 분자 도킹에 대한 표적 친핵성 잔기의 대체 회전 이성질체 선택 효과를 조사하였습니다.
Caldwell 등1의 화합물 A3-A26을 Flare로 rule-based protonation 옵션을 적용하여 가져왔습니다(그림 3). 가져온 리간드 구조의 양성자화 상태를 주의 깊게 확인했습니다. 화합물 A5(amino-pyridine hinge binder)의 pyridyl aza nitrogen은 수동으로 neutralize 하여 일관된 binding pose로 Met477 주골격 NH와 수소 결합을 유지하도록 했습니다. 데이터 세트(A18, Evobrutinib 포함) 내 모든 리간드는 공유 결합 warhead로서 Michael acceptor을 가지고 있습니다.
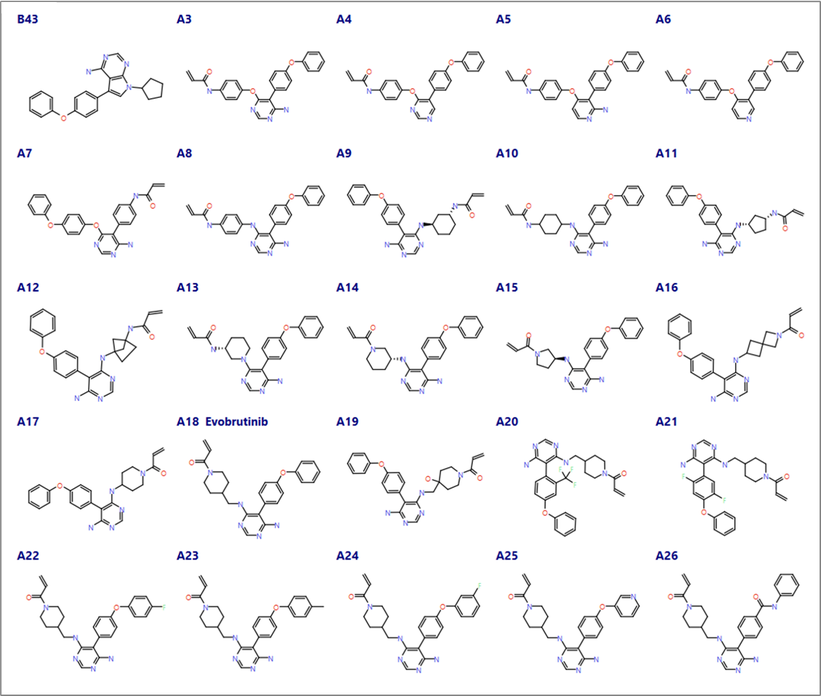
그림 3. Caldwell 등1의 연구에 이용된 다양한 코어를 가진 화합물(hinge 결합 도메인(A3-A8), warhead와 core 간 대체 링커(A9-A19), 선택성 포켓(A20-A26)).
예비 도킹 실험
Flare에서 그림 4와 같이 Covalent Docking Calculation 패널에서 “Normal” 방법, 결정 구조상 물 분자 배제, Cys481을 친핵성 아미노산 잔기로 설정한 후 3GEN 활성 부위 내 hinge 결합 설계(A3-A8)의 도킹 실험을 수행하였습니다. 초기 실험은 좋은 도킹 결과를 얻기 위해 다른 제한 조건(constraints)이 필요한지 또는 리간드나 단백질의 변경이 필요한지 여부를 신속하게 확인하기 위함입니다.
각 도킹 실험의 pose는 반경험적(semi-empiric) 분자역학 함수에 기반하여 결정학적 pose를 가장 잘 재현하도록 최적화된 Lead Finder의 ‘rank score’를 기준으로 그 우선순위를 매겼습니다.
결과는 긍정적이었으며, conformational sampling 최적화 및 더 유연한 링커를 포함한 다른 설계 결과와의 일관성 극대화를 위해 ‘Accurate but Slow’ 및 ‘Very Accurate but Slow’ 방법을 이용하여 공유 결합 도킹 실험을 추가로 진행하였습니다. ‘Accurate but Slow’ 방법은 계산 속도와 계산 정확성 간의 최적 균형을 제공합니다.
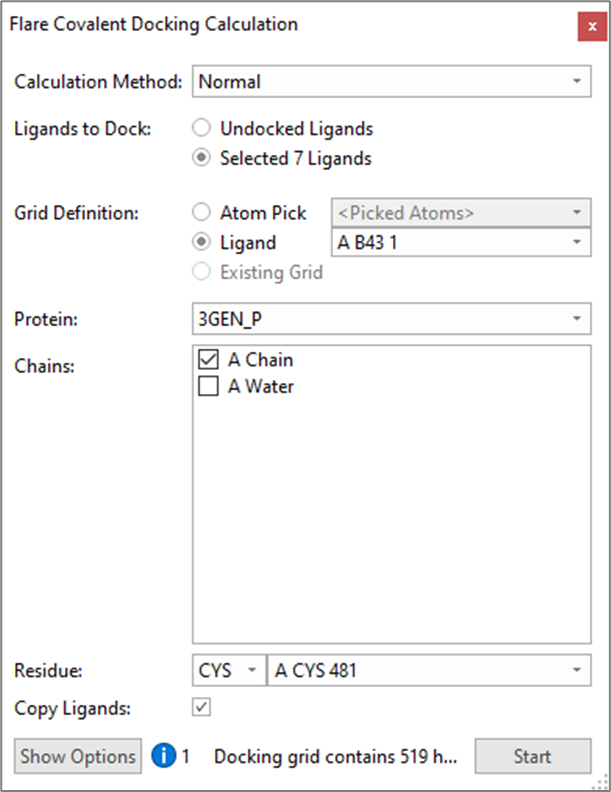
그림 4. Flare의 공유 결합 도킹 계산 패널
결과 및 분석
Hinge binder 설계(그림 3의 A3-A8)의 경우, 원래의 Cys481 회전 이성질체를 이용한 것에서 가장 좋은 결과를 얻을 수 있었습니다. 이들의 설계는 결정학적 리간드 B43에 가까운 유사체입니다. Flare 공유 결합 도킹 워크플로는 화합물 A3과 A5를 가장 좋은 것으로, A7을 가장 나쁜 것으로 평가했습니다.
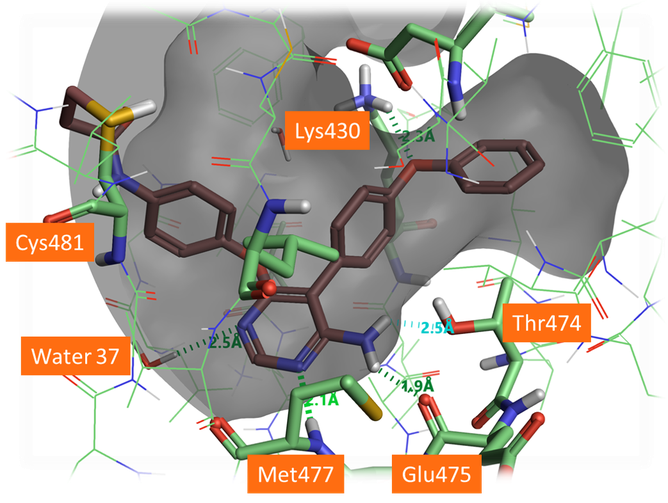
그림 5. 단백질 주골격 및 문지기 역할의 Thr474와 수소 결합을 형성하는 A3의 결합 pose. 5 위치의 aza-nitrogen은 활성 부위 내 특정 하나의 물 분자와 수소 결합을 형성함(도킹 계산에서는 이를 배제함). 해당 화학 계열의 도킹 계산에는 이 물 분자를 고려하는 것이 유익할 것임.
그림 5에 표시한 것처럼, A3와 A5는 Met477의 주골격 amine기, Glu475의 주골격 carbonyl기 및 Thr474(소위, 문지기 잔기)의 곁사슬 hydroxyl기와 좋은 수소 결합을 형성합니다. EGFR kinase에서는 문지기 잔기가 Met790이기 때문에 Thr474 잔기와의 수소 결합 형성은 EGFR kinase와 비교하여 BTK에 대한 고유 선택성을 얻을 수 있는 기회를 제공합니다. Phenoxyl phenyl moiety는 선택성 포켓을 채우고 Lys430과 수소 결합을 형성합니다. 흥미롭게도, pyrimidine 리간드의 5-aza nitrogen은 단백질과 직접적으로 상호작용하지 않지만 원래의 B43 화합물처럼 결정학적 물 분자와 수소 결합 상호작용을 합니다. Pyrimidyl hinge 결합 계열의 향후 도킹 실험에 이러한 물 분자를 고려하는 것은 가장 유망한 pose를 발견하고 평가하는데 도움이 될 것입니다.
화합물 A4와 A6는 2-amino 치환기를 가지고 있지 않아 Thr474와 수소 결합을 형성할 수 없지만 다른 상호 작용은 화합물 A3과 A5에서 관찰된 것과 동일합니다. 해당 신약 개발의 목표는 좋은 kinase 선택성을 타겟으로 하고 있기 때문에 BTK의 Thr474와의 상호작용을 형성하지 않으면 BTK vs. EGFR의 고유 선택성에 영향을 미칠 수 있습니다.
A7 화합물은 역 amino-pyrimidine moiety를 가지고 있어 hinge 도메인을 갖는 세트 내 다른 설계와 동일한 상호작용을 형성할 수 없습니다. 이는 Caldwell 등1이 보고한 BTK 및 EGFR에 대한 낮은 활성과 일치합니다.
링커 설계(그림 3의 A9-A19)의 공유 결합 도킹은 원래 및 모델링한 Cys481 회전 이성질체에서 모두 좋은 pose를 도출하였습니다. 대부분의 화합물은 모든 도킹 실험에서 유사한 pose를 보여 주었으며 링커 conformation의 차이만을 관찰할 수 있었습니다. 이들의 설계에서는 활성 부위 내 이 영역이 열려 있고 대개 용매가 접근 가능하기 때문에 우선 순위 매김이 더 어렵습니다. 도킹에서는 원래의 Cys481 회전 이성질체를 이용할 때 화합물 A11과 A13-15는 낮게 평가되고 대체 Cys481 회전 이성질체의 경우 A13과 A16이 낮게 평가된다는 정보만을 알 수 있을 뿐입니다. 단백질이 없을 때의 중복 pose를 minimization 하여 화합물 내 변형을 추론했습니다.
또한, 선택성 포켓의 설계는 phenoxy phenyl 고리의 마이크로솜의 안정성 개선에 중점을 두었습니다. Docking score 및 단백질-리간드 contact을 분석함으로써 A20과 A21의 phenyl 고리의 치환기에서 일부 입체적 충돌을 확인하였습니다. 이 세트(A22-A26) 내 다른 화합물은 말단 phenyl 고리에 변화를 가지며 모두 선택성 포켓에 알맞습니다.
기존 도킹과 마찬가지로 잘 준비된 단백질과 정확한 conformation은 결과에 상당한 영향을 미칩니다. 본 사례 연구에서는 덜 유연한 리간드의 경우 core와 warhead 사이에 aromatic 링커를 포함하는 hinge 결합 설계와 같이 원래의 Cys481 회전 이성질체를 선호하는 경향으로 나타났습니다. 링커 설계에서 더 유연한 리간드는 대체 Cys481 회전 이성질체를 이용할 때 더 좋은 docking pose를 도출했습니다.
Evobrutinib의 X-ray 구조와 docked pose 비교
BTK 활성 부위(PDB: 6OMU)에 공유 결합한 Evobrutinib의 결정 구조와 PDB: 3GEN 활성 부위 내 Evobrutinib의 docked pose를 비교할 때 두 결정 구조 간의 차이를 강조할 수 있습니다(그림 6). 특히, 6OMU의 Cys481 곁사슬 conformation은 3GEN과 다르며, 6OMU 활성 부위 내 Lys430 conformation과 일부 물 분자에서 약간의 차이를 보입니다.
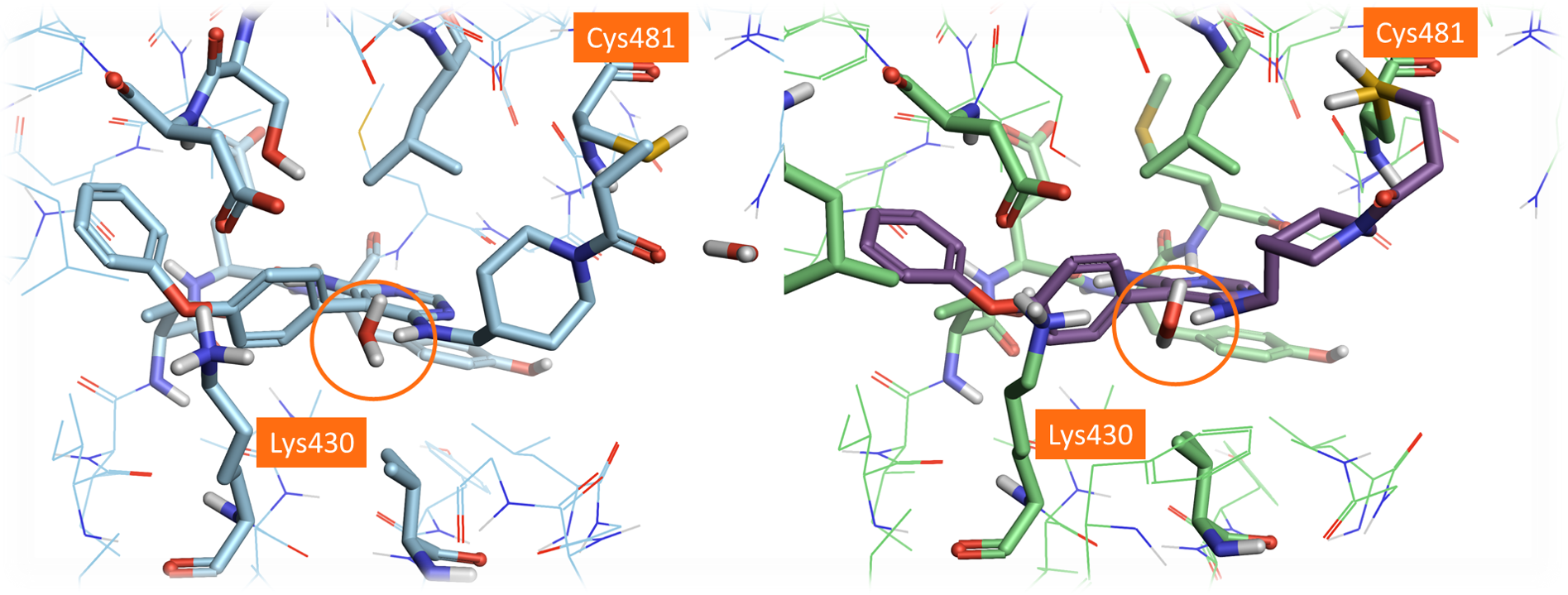
그림 6. 좌: Evobrutinib의 결정 구조(PDB: 6OMU). 우: PDB: 3GEN 활성 부위 내 도킹 시 Evobrutinib의 best scoring pose(‘Accurate but Slow’ 방법 이용). 두 구조를 비교할 때 Cys481의 곁사슬 conformation에 차이가 있음을 관찰할 수 있음.
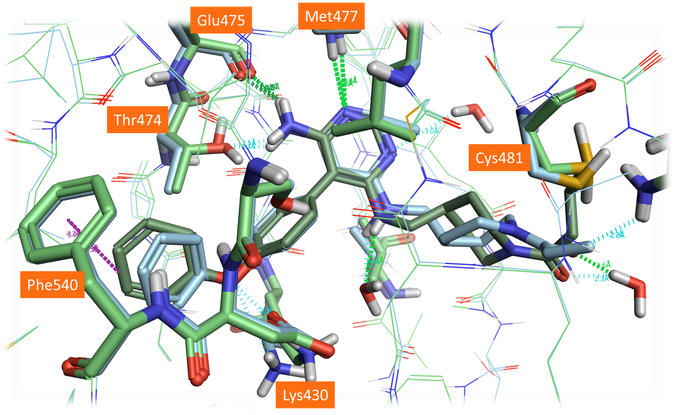
그림 7. Evobrutinib의 결정 구조(PDB: 6OMU, 연한 파란색)과 대체 Cys481 회전 이성질체를 적용한 도킹 시 Evobrutinib의 best scoring pose(녹색) 비교
Cys481의 대체 곁사슬 conformation은 6OMU의 실험적 위치에 더 근접해 있습니다. 당연하게도 친핵성 잔기에 대한 다른 회전 이성질체의 선택은 화합물의 전체 binding pose에 영향을 미칩니다. Cys481에 대체 회전 이성질체를 적용함으로써 결과를 크게 개선시킬 수 있었지만 링커 conformation은 여전히 결정 구조에서 관찰된 것과 약간의 차이를 나타냅니다. 이는 3GEN 단백질 구조의 활성 부위 내 특정 물 분자(특히, 링커 사슬의 amine기에 인접한 ‘1024’ 물 분자)를 고려함으로써 더욱 개선될 수 있습니다. 최종 pose는 결정 구조에서 관찰된 것과 매우 유사하며, 포화 고리 conformation에서 약간의 차이만 있습니다.
결론
Flare는 공유 결합 리간드의 준비, 설계 및 도킹 실험을 위한 사용자 친화 인터페이스를 제공합니다. 공유 결합 도킹 워크플로는 유사-공유 결합 리간드의 생성(도킹 결과를 Flare 프로젝트에 저장 가능), 다양한 docked pose의 탐색, 결과의 후처리 기능을 제공하여 신약 개발 프로젝트의 진행에 있어 가장 적합한 후보 물질을 선정하는데 많은 도움이 됩니다.
참고문헌 및 링크
- Caldwell et al., J. Med. Chem. 2019, 62, 7643-7655
- https://www.cresset-group.com/software/flare/
- https://www.cresset-group.com/software/lead-finder/