Anand Balupuri†, Giovanna Tedesco†
†Cresset, New Cambridge House, Bassingbourn Road, Litlington, Cambridgeshire, SG8 0SS, UK
摘要
化合物的代谢稳定性是药物发现和开发的关键因素。由于代谢稳定性差,一些具有良好药理特性的化合物未能成为药物。生物等排体替换是调节化合物特性的有力工具。在本案例研究中,演示了如何用Cresset的生物等排替代与骨架跳跃软件Spark[1]来解决代谢稳定性问题。
前言
丝裂原活化蛋白激酶(MAPK)通路是主要的细胞信号通路,该通路的异常激活经常驱动肿瘤生长[2]。细胞外信号调节激酶1和2(ERK1/2)在MAPK通路中发挥着核心作用[3],是治疗肿瘤有希望的靶点。
在过去的几年里,各种具有良好临床前疗效的ERK1/2抑制剂已进入临床试验阶段[4]。异吲哚啉酮是Astex Pharmaceuticals最近报道的一类新型ERK1/2抑制剂[5]。化合物7(见图 1–左和表1)是一种高活性、选择性的ERK1/2抑制剂,在小鼠中显示出有前景的药代动力学特征,然而,由于P450 3A4 (CYP3A4) 的氧化代谢,其体外半衰期较短(5.1 分钟)[6]。 对代谢物分析的结果表明,化合物7两端的甲苯基与氧杂环发生了显著的氧化代谢。
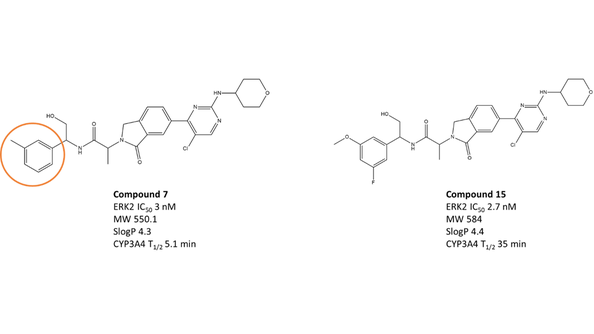
图1. 左:化合物7是一个半衰期短、强效、高选择性的ERK1/2抑制剂。右:优化过的化合物15不仅活性增强而且延长了半衰期。
提高化合物7代谢稳定性的先导优化策略包括:通过用更稳定的等排体取代以便直接阻断易损位点(类似物的SAR数据如表1所示)、构象刚性化和调节化合物的整体物理化学性质。这些努力发现了化合物15(见图1-右和表1),其显示出稍微更高的活性与显著改善的CYP3A4代谢稳定性,体外半衰期为35分钟。化合物15(ASTX029)目前正处于对晚期实体恶性肿瘤患者进行I-II期临床试验中[6]。
表1. 甲苯片段替换的SAR探索[6]
| Compound | R | ERK2 IC50 (nM) | CYP3A4 T1/2 (min) |
| 7 | 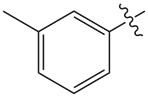 |
3.0 | 5.1 |
| 14 | 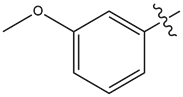 |
1.3 | 5.5 |
| 15 | 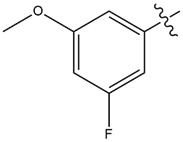 |
2.7 | 35 |
| 16 | 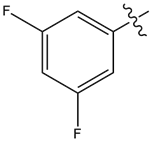 |
43%@1 | 小于5 |
| 17 | 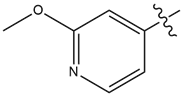 |
1.8 | 15 |
已公布的先导化合物7与ERK2复合物X-射线晶体结构(PDB 6G9N)表明,该分子同时占据铰链区和第二口袋(图2),人们认为这是引发双重药理学特性所必需的[6]。甲苯片段部分与第二口袋结合,并与TYR64发生芳香-芳香相互作用。含氧杂环与附近的铰链区结合,并与ERK2的活性位点没有发生任何的特异性相互作用。
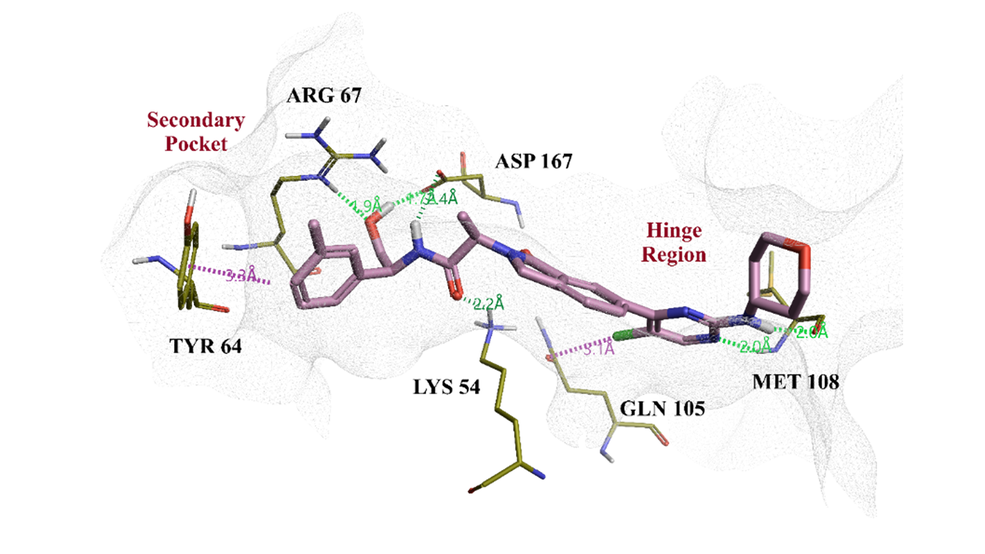
图2. 化合物7结合到ERK2的活性位点里(PDB 6G9N)
通过使用Cresset专有的分子场技术,SPARK给出了一系列在生物学上有价值的片段替换建议[7]。Spark用来自市售和文献化合物库的片段来替换给定起始物分子的特定部分,这些片段显示出相似的静电和立体特性。SPARK相对于同类工具的主要优点是它对分子的整体进行打分,而不是仅对替换片段进行打分。这种以产物为中心的方法将替代片段对分子其余部分的电子和构象效应考虑在内。在本文中,我们用Spark快速地识别甲苯基部分的替代R-基团(图1-左橙色圆圈),保留对ERK2活性,同时解决代谢敏感性问题。
方法
先导化合物7与ERK2的复合物X射线晶体结构(PDB 6G9N)用作本案例研究的起点。Spark提供了可以轻松设置各种实验的向导。 我们使用“Scaffold Hopping or R-group Replacement”向导(图3-左)来设置实验。
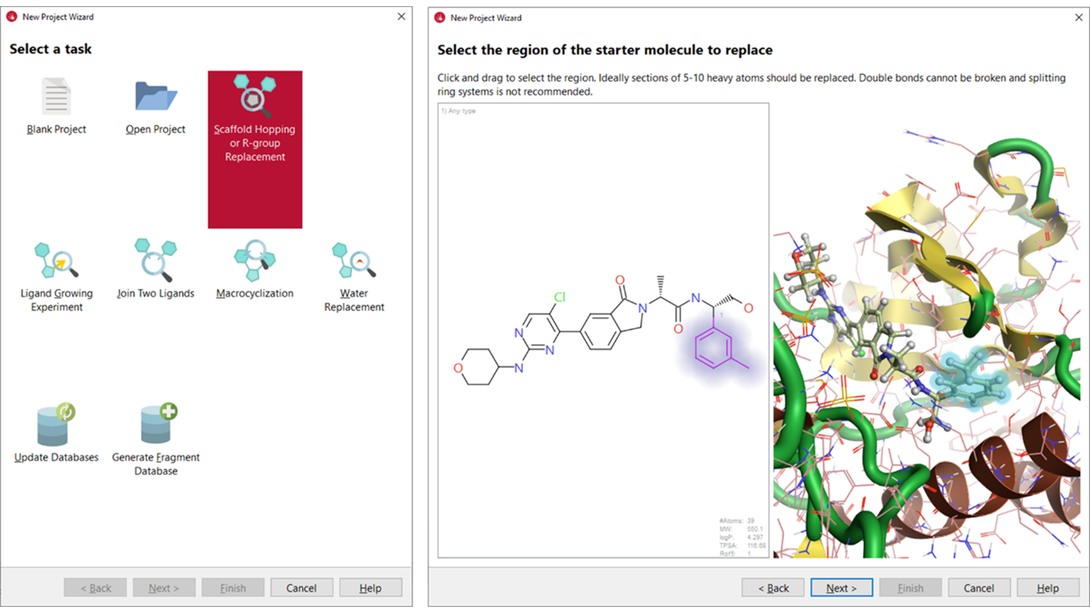
图3. 左:SPARK的项目向导。右:在Spark向导中,在2D和3D窗口中以光晕突出显示起始分子被选中进行替换的区域。
PDB 6G9N 直接从PDB下载到Spark中,并使用“Let Spark choose the protonation state”选项进行准备。选择共结晶配体(化合物 7)作为起始分子,并删除所有的结晶水分子。选择甲苯片段作为起始分子进行替换的区域(图 3–右)。
计算的时候,使用了下面的设置:
- Calculation method: Accurate but Slow
- Databases to search: ChEMBL_common, Very Common and Common
这几个Spark片段数据库共总包含了97,000多个片段可用于R基团替换实验。这些片段有的来自文献报道的化合物(ChEMBL 数据库)[8],有的来自市售化合物(VeryCommon 和 Common)[9]。
此外,基于起始分子的物化性质(MW:550,SlogP:4.3,TPSA:117,柔性:8.5),使用以下高级设置将Spark搜索聚焦于具有类药性质的结果上:
- Maximum number of heavy atoms: 50
- Maximum molecular weight: 610
- Calculated lipohilicity (SlogP) range: 3-5
- Calculated Topological Polar Surface Area range: 40-140
结果
使用以上详述的Spark设置对先导化合物7的甲苯片段进行R基团替换实验生成了1000个基于50%场与50%形状相似性的排序建议,其中生物等排因子 (BIF%) 值的范围为11到92。BIF%是一个标度分数,使得原始分子的打分为100%,删除要替换的部分的分值为零。正值表示有利的生物等排体,负值表示替换之后可以重现起始分子几何形状,但对删除部分(被替换部分)的模拟效果不佳。较大的结果聚类包括了苯基、噻吩、吡啶和呋喃骨架。毫不奇怪,大多数结果 (348个) 属于打分最高的苯基取代R基团类。
苯取代的R-基团
这个类别中的Spark结果包括许多有趣的变体,这些变体有助于以微妙的方式调节R基团的静电和形状,以解决代谢稳定性问题,利用药物化学知识来对分子进行优先级排序。例如,药物化学家通常使用氟和甲氧基来阻断代谢敏感点,因为碳-氟键比碳-氢键更能抵抗细胞色素P450的攻击。 此外,邻近或远离代谢攻击位点的氟取代会改变药物代谢[6,10,11]。有意思的是,Spark的结果包含了许多氟取代和甲氧基取代的片段,包括用于优化先导化合物7的R基团(见表1和图4)。
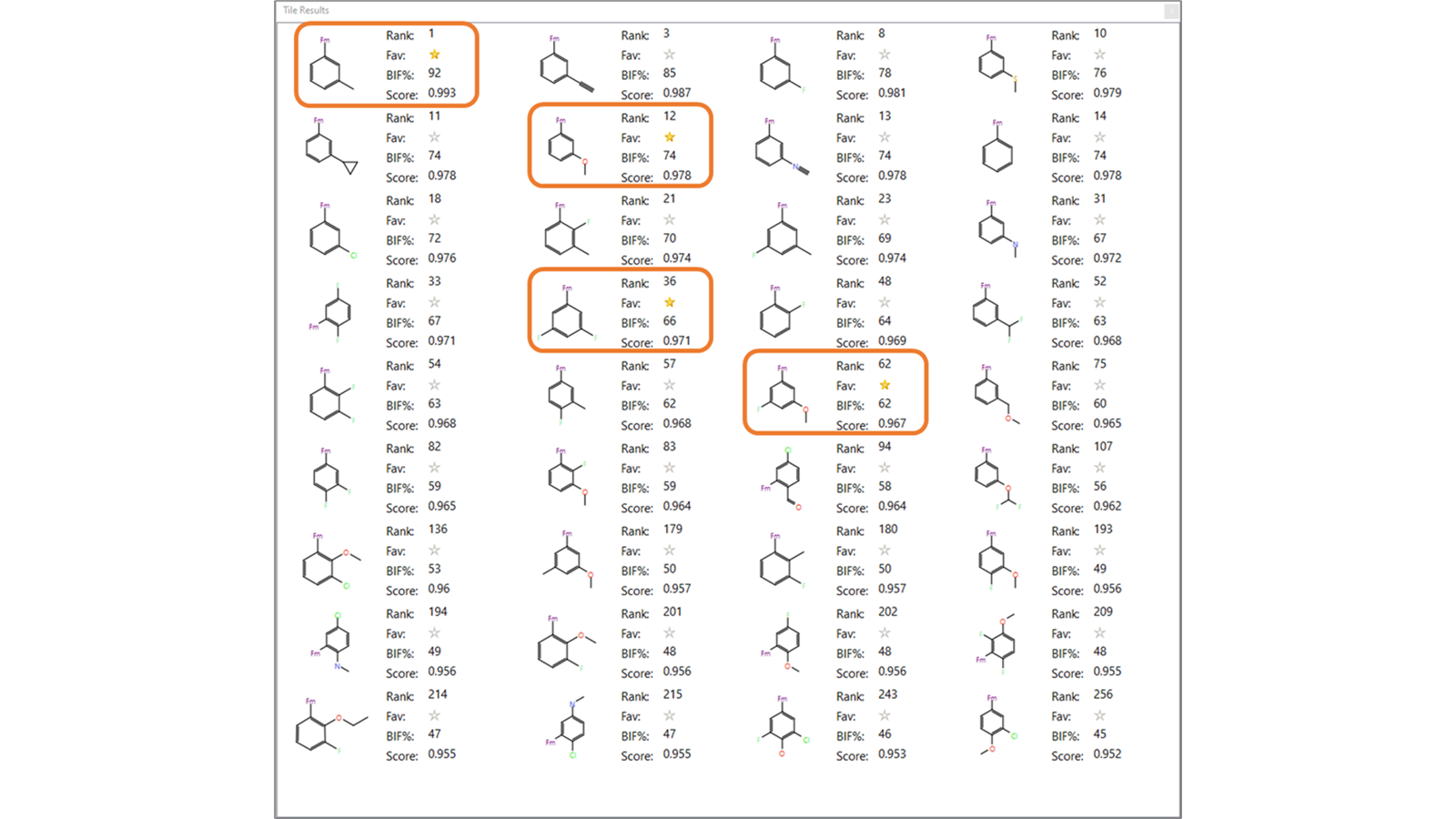
图4. 平铺视图呈现了Spark结果中的一些取代苯结构。其中高亮显示的R基团替换被Astex Pharmaceuticals用于先导优化[6]。
其它杂环
也用更缺电的环系替换芳香系来提高对细胞色素P450介导的氧化的稳健性。特别是,用吡啶基替换苯环是解决代谢问题的最常见策略[12]。在这种情况下,Spark提出了许多有趣的杂环替换甲苯的方案,包括各种邻-、间- 、对-吡啶衍生物结构(图 5)。
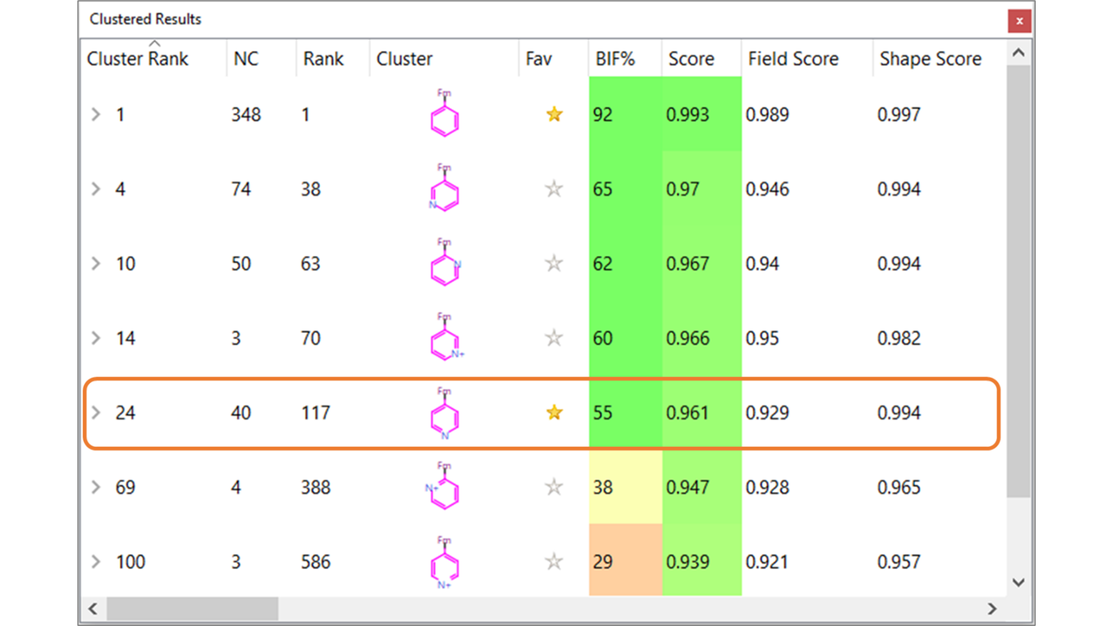
图5. Spark结果的top-score聚类视图。请注意,为了清晰起见,不包括双环和五元杂环化合物。
特别是对位吡啶取代基这个类别包含了40个片段,与原始论文[6]中报道的那些结构相似,按类排序为24。该类片段包含了表1中的化合物17,BIF% = 46较低,因其在静电上与化合物7的相似性低于表1中的其它化合物(图 6)。
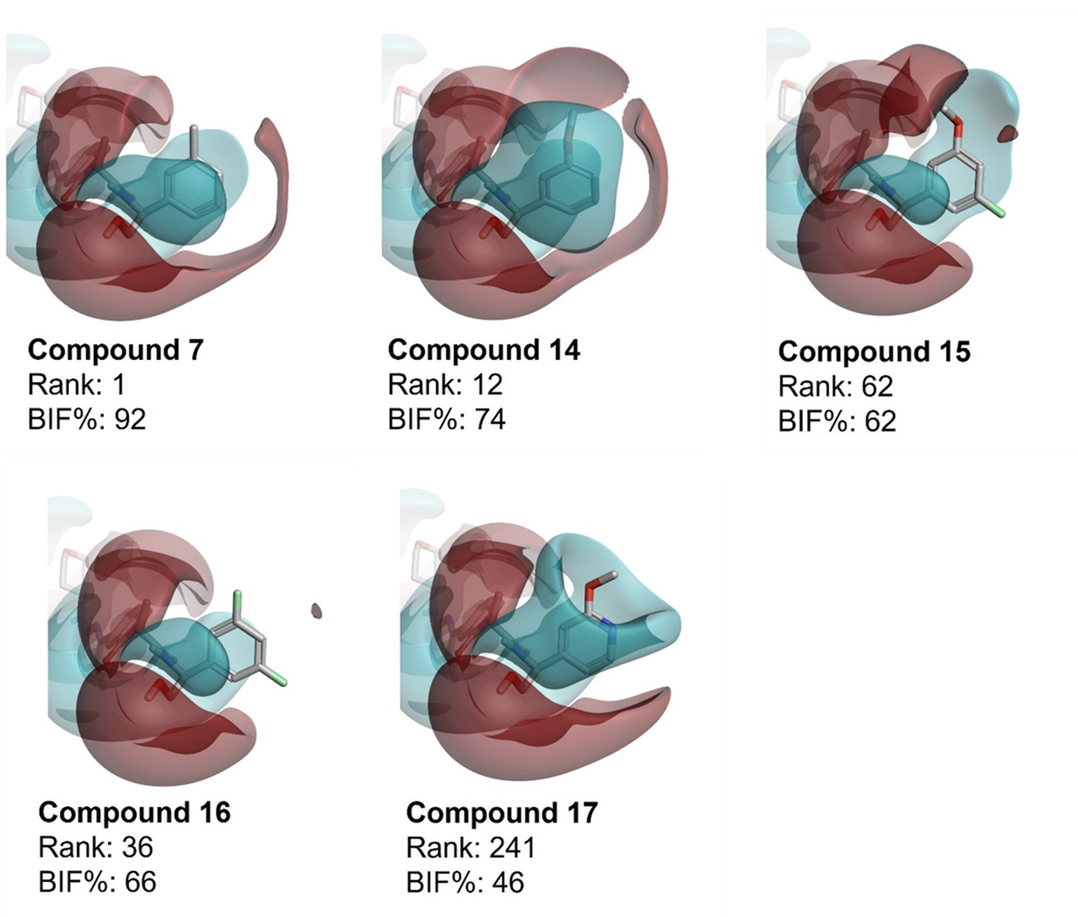 图6. 表1所列化合物的正(红色)和负(蓝色)静电表面的比较。化合物17 与其他化合物最不相似的区域是吡啶氮原子前面的负静电区。
图6. 表1所列化合物的正(红色)和负(蓝色)静电表面的比较。化合物17 与其他化合物最不相似的区域是吡啶氮原子前面的负静电区。
其它令人感兴趣的结果
先导化合物7的甲苯基片段经历了广泛的氧化代谢[6]。用环丙烷替换甲基可以抑制细胞色素P450介导的代谢,因为从环丙烷环中拔掉质子比从甲基中拔掉质子更加困难,拔掉质子是氧化代谢所需的[13]。在打分最高的SPARK结果中,我们可以找到环丙基取代的结果(图4),其打分排名第11。
化合物14用苯甲醚替换甲苯,显示出相似的代谢稳定性(表1)。然而,用二氟苯甲醚替换苯甲醚可以改善代谢稳定性[14]。二氟苯甲醚取代的结果也出现在SPARK的结果中(图4),打分排名为107。这些甲苯片段的替换方案为进一步探索该先导系列提供了令人感兴趣的选择。
结论
在本案例研究中,Spark成功地识别出了Astex Pharmaceuticals在发现临床候选药物ASTX029时使用的相同或非常相似的R基团替换物。该算例表明,使用Spark的生物等排体替换结合药物化学知识有助于解决在药物发现过程中与代谢相关的问题。
参考文献
-
Samatar A. A. et al., Nat. Rev. Drug Discov. 2014, 13(12), 928-942
-
Pearson G. et al., Endocr. Rev. 2001, 22(2), 153-183
-
Roskoski R. et al., Pharmacol. Res. 2019, 142, 151−168
-
Heightman T. D. et al., J. Med. Chem. 2018, 61(11), 4978−4992
-
Heightman T. D. et al., J. Med. Chem. 2021, 64(16), 12286-12303
-
https://www.emolecules.com/info/products-screening-compounds
-
Shah P. et al., J. Enzyme Inhib. Med. Chem. 2007, 22(5), 527-540
-
Park B. K. et al., Annu. Rev. Pharmacol. Toxicol. 2001, 41(1), 443-470
-
Lazzara P. R. et al., RSC Med. Chem. 2020, 11(1), 18-29
-
Talele T. T., J. Med. Chem. 2016, 59(19), 8712-8756
-
Xing L. et al., ChemMedChem 2015, 10(4), 715-726