Scott Midgley†, Nathan Kidley†
†Cresset, New Cambridge House, Bassingbourn Road, Litlington, Cambridgeshire, SG8 0SS, UK
초록
Fragment 기반 신약 개발은 hit를 lead 분자로 개발하는 데 있어 고유한 과제를 가지고 있는 진화하는 연구 분야입니다. 본 사례 연구에서는 bioisostere 솔루션인 SparkTM1을 이용하여 구조 기반 가상 스크리닝이 tuberculosis(TB) 치료를 위한 신규 InhA 환원 효소 억제제를 어떻게 효과적으로 찾아내는지 소개합니다. 우리는 Spark의 docking score 기능이 활성 부위의 새로운 영역으로 fragment를 성장시켜 화학의 새로운 영역을 확인하는 데 어떻게 이용될 수 있는지 설명합니다.
서론
TB는 21세기에도 여전히 주요 사망 원인으로 남아있습니다. Enoyl-[acyl-carrier-protein] reductase (InhA)는 TB 저분자 치료 연구에서 일반적인 표적입니다. 그러나, 선도 치료 화합물 중 대다수는 이 효소를 억제하기 전에 NAD 경로를 통해 활성화될 필요가 있습니다. 이 활성화의 차단은 TB 저항 메커니즘으로 알려져 있습니다. 따라서, 대사 활성화없이 InhA를 억제하는 치료는 흥미롭고 활발한 연구 분야입니다. 이는 Fragment 기반 스크리닝 연구를 수행한 후 hit fragment를 최적화한 Sabbah 등4의 목표였습니다. 본 연구에서는 Spark가 도킹 방법을 이용하여 실험적 hit 화합물을 독립적으로 식별하여 fragment를 InhA 활성 부위로 성장시킬 수 있음을 보여줍니다.
Spark에서 결과를 스코어링하는 종래의 방법은 주로 리간드 기반 방법입니다. Cresset의 field similarity 기술은 shape similarity metric과 함께 이용되어 결과를 starter 분자와 비교하여 bioisosteres를 식별합니다. 한편, Spark는 단백질과 리간드로부터 직접 도킹 스코어를 계산하는 대안적인 스코어링 프로세스에서 Lead FinderTM2 도킹 엔진을 이용할 수 있습니다. 이 접근 방식을 통해 사용자는 활성 부위의 미탐색 영역으로 리간드를 성장시키고 타겟 단백질과 새로운 상호작용을 형성하는 결과를 확인할 수 있습니다.
이 사례 연구에서는 ‘Site I’에 결합된 starter 리간드(PDB: 6SQ5)를 단백질 구조와의 상호작용은 유지하면서 carboxylic acid moiety를 치환하여 활성 부위의 ‘Site III’로 성장시켰습니다(그림 1). 이는 리간드를 ‘Site II’로 성장시키기에는 범위가 제한되었기 때문입니다. “Site III”로의 성장은 리간드의 고유 효능을 높일 수 있는 가능성을 제공했습니다.
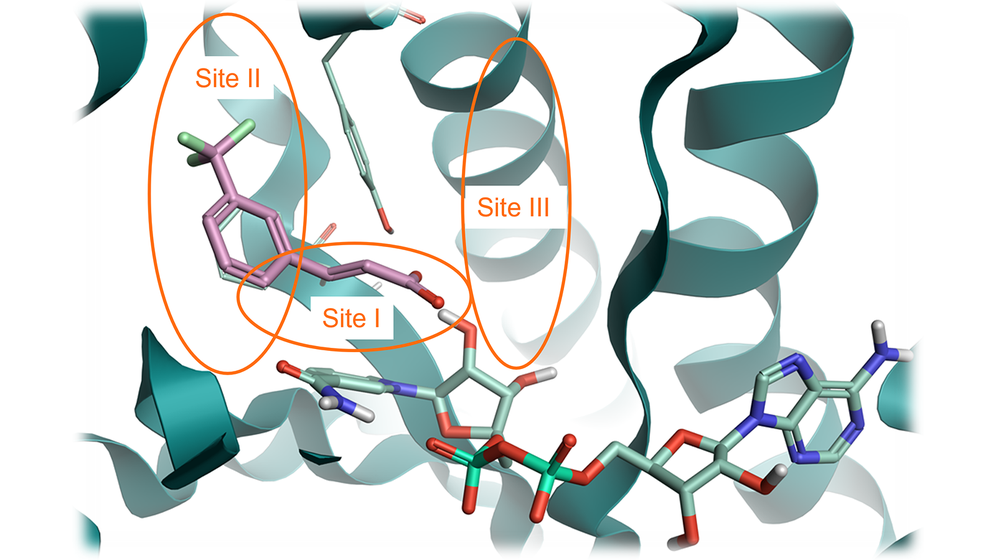
그림 1. 세 개의 주요 부위를 함께 나타낸 InhA 단백질(PDB: 6SQ5). Site I: 촉매 부위; Site II: 소수성 부위; Site III: 용매 노출된 영역. 분홍색: 성장시킬 fragment 구조. 옅은 녹색: NAD cofactor. 녹색 리본: InhA 단백질.
Spark에서의 전산 fragment 스크리닝
Spark 도킹 실험에 앞서, 종래의 Spark field 및 shape 검색을 수행하여 그림 1에 나타낸 fragment의 carboxylate group의 잠재적인 bioisostere들을 확인했습니다. 이를 통해 Spark 도킹 실험을 준비하기 위한 화학적 탐색 공간에 집중할 수 있었습니다.
이 탐색으로 sulfoxide, sulfone, sulfonamide, sulfoximine, imidazole, pyrazole 및 triazole fragment 등 다양한 bioisostere들을 확인하였습니다. 우리는 유사체 합성에 편리하고 합성적으로 접근 가능한 sulfone 및 sulfonamide bioisostere에 주목했습니다. 또한, 이들 fragment는 Site II 또는 III로 리간드를 성장시키는 데 적합한 벡터를 제공합니다.
도킹 실험을 위해 다음 세 개의 Phase로 구성된 단계적 접근을 수행했습니다.
- Carboxylate group의 bioisosteric 치환. 이 단계에서는 X-ray 리간드의 carboxylate를 치환하여 starter 분자를 성장시키기 위해 도킹 방법을 이용했습니다. Fragment 공간은 SO2 substructure를 포함하도록 필터가 적용되었습니다. 또한, 자동 fragment 사이즈 구속을 이용하여 치환된 fragment의 사이즈를 carboxylate group과 유사하도록 제한했습니다.
- Phase 1의 top-ranking 결과로부터 시작하는 fragment 스크리닝 실험. Spark 도킹 실험은 fragment를 더욱 성장시키기 위한 starter 분자로 phase 1의 top-ranking sulfone 결과를 이용했습니다. 이 실험에서는 fragment에 substructure 필터를 적용하지 않고 자동 fragment 사이즈 구속을 이용했습니다.
- 단백질 상호작용 구속을 이용한 phase 1의 top-ranking sulfone 결과에 대한 fragment 스크리닝 실험. 도킹 방법을 이용하였고, 결과가 활성 부위 내 NAD 분자의 중요 산소 원자와 수소 결합을 반드시 포함하도록 하기 위해 도킹 구속을 적용했습니다.
Phase 1: Carboxylate group의 bioisosteric 치환
활성 부위 내 리간드를 성장시키기 위한 목적으로, 우리는 carboxylate에 대한 bioisosteric 치환체들을 확인하기 위해 도킹 방법을 이용했습니다. 도킹 grid는 실험 리간드로부터 생성하였고, grid의 사이즈는 fragment가 Site III으로 성장할 수 있을 만큼 충분히 큰 8 Å으로 설정하였습니다. 검색에 이용한 fragment 데이터베이스는 ChEMBL, Commercial Very Common, Common이며, 이들은 약 100,000 개의 fragment를 제공합니다5,6.
검색된 fragment가 SO2 group을 포함하도록 하기 위해 substructure 필터를 고급 검색 옵션으로 추가했습니다. 치환 fragment들의 사이즈가 carboxylic acid group과 유사하도록 제한하기 위해 자동 fragment 사이즈 구속도 함께 추가했습니다. 이렇게 하면 검색 공간이 상대적으로 작은 SO2 포함 fragment들에 집중됩니다.
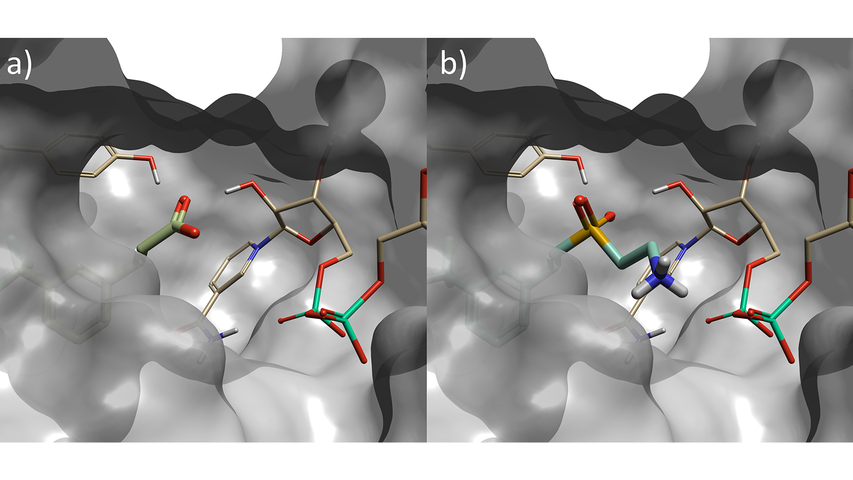
그림 2. a) PDB: 6SQ5의 단백질 내 starter 분자(결정학적 리간드), b) Phase 1 Spark 실험의 top scoring sulfone 화합물 – fragment가 starter 분자에 비해 NAD phosphorus 원자와 더 가까이 접촉하고 있다.
첫 번째 phase의 top scoring 결과는 carboxylate를 직접 치환한 SO2 group을 포함한 많은 hit을 산출했습니다. SO2가 활성 부위의 다른 부분 또는 carboxylate carbon에서 더 멀리 떨어진 원자를 점유한 결과도 있었습니다. 이러한 차이는 SO2 필터가 공간적으로 구속되지 않기 때문에 발생하며, 부착점 구속을 통해 개선할 수 있습니다.
Top scoring 결과를 그림 2에 나타내었습니다. 치환 fragment는 활성 부위에 tyrosine 잔기와 수소 결합을 형성하도록 배향된 SO2 oxygen 원자들 중 하나를 가집니다. 두 번째 수소 결합은 NAD sugar의 hydroxyl group에 형성되어 starter 분자의 carboxylate가 만들어내는 상호작용을 모방합니다. 또한, ethylamine group은 sulfone으로부터 확장되어 NAD cofactor의 phosphate와 추가적인 수소 결합을 형성합니다. 이것은 종래의 리간드 기반 방법으로는 발견할 수 없었던 새로운 상호작용으로 Starter 리간드가 활성 부위의 이 영역에 원자를 가지고 있지 않았기 때문입니다.
Phase 2: 확인된 sulfone에 대한 두 번째 fragment 스크리닝 실험
Phase 1의 top scoring sulfone-ethylamine 화합물을 두 번째 실험의 starter 분자로 이용했습니다(그림 3). Ethylamine group을 치환할 fragment로 선택했습니다(그림 3의 파란색 후광). 부착점 원자(그림 3의 빨간색 원)는 nitrogen 원자로 구속하여 실험의 결과가 carboxylate moiety의 sulfonamide bioisostere들만을 포함하도록 했습니다. 자동 fragment 사이즈 구속이 고급 옵션으로 사용되었습니다.
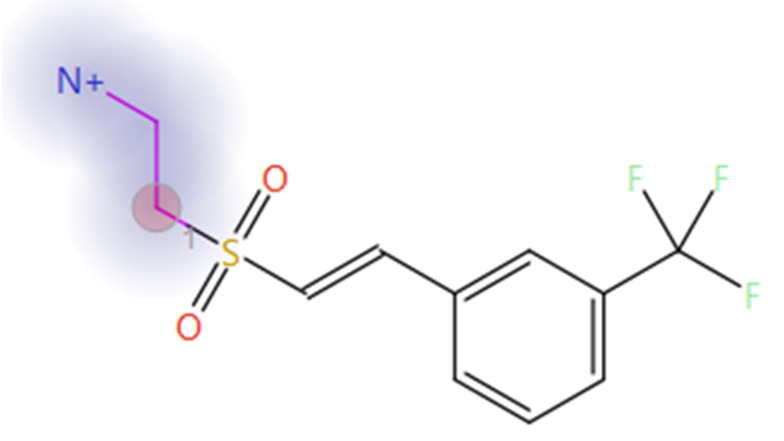
그림 3. Sulfone-ethylamine 화합물은 phase 1 Spark 실험에서 top scoring 결과이며, phase 2의 업데이트된 starter 분자로 이용되었습니다. 파란색 후광: 치환할 fragment. 빨간색 원: 부착점.
Phase 1에서와 마찬가지로 Very Common, Common ChEMBL, 및 Commercial fragment 데이터베이스를 검색에 이용하였으며, 이 경우에 새로운 starter 분자 주위에 5 Å grid를 이용했습니다. 작은 grid 사이즈가 도킹 실험의 계산 비용을 줄이기 위해 선택되었으며, 업데이트 된 reference 분자는 Site III(그림 1)에 대한 접근을 용이하게 합니다.
몇 가지 흥미로운 sulfonamide 치환체가 phase 2에서 확인되었습니다(표 1). 이들은 그림 4a 및 4b의 Imidazole 및 morpholine 구조를 포함하며, reference 구조에서 먼 거리에 위치한 단백질 및 NAD(황갈색으로 표시한 탄소 원자)와 상호 작용을 형성합니다.
표 1. Phase 2에서 확인된 상위 6개 fragment를 나타낸 Spark Results 테이블
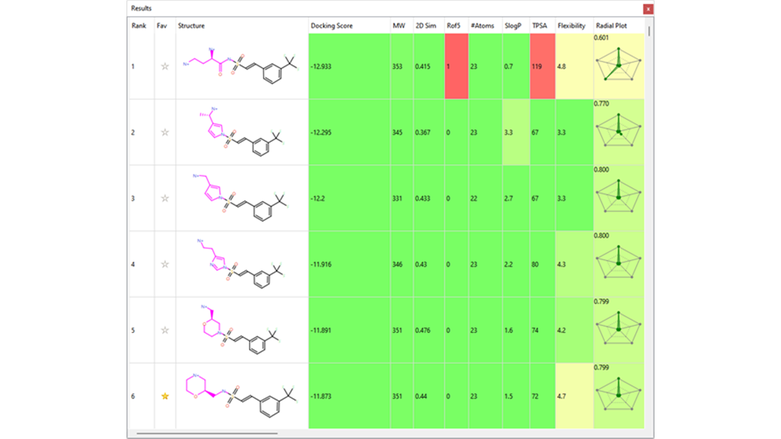
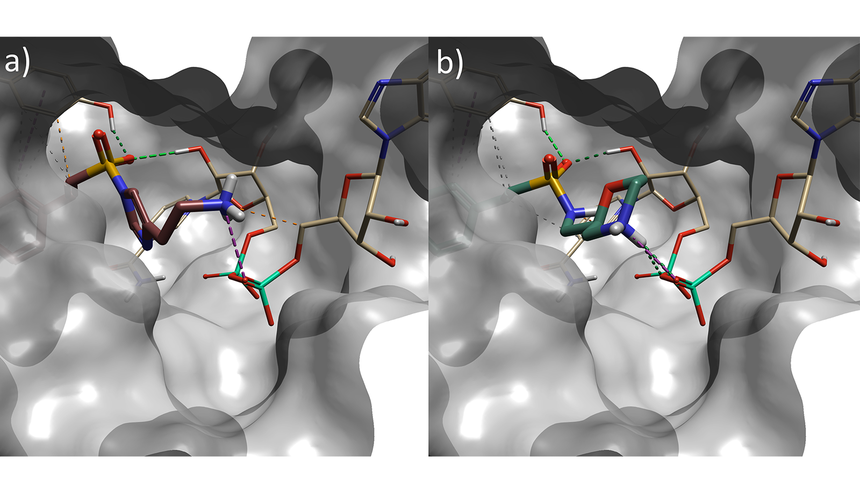
그림 4. a) Spark 결과 중 Imidazole sulphonamide(result 4), b) Spark 결과 중 Morpholine sulphonamide(result 6). 황갈색: NAD cofactor. 회색 표면: 단백질 활성 부위 표면
Phase 3: 확인된 sulfone에 대한 세번째 fragment 스크리닝 실험, 단백질 contact 구속 조건 적용
Phase 1 및 2에서 수행된 Spark 실험에서는 수많은 흥미로운 화합물(designs)이 확인되었습니다. 이러한 결과를 보완하고자 일반적인 반응으로부터 합성 가능한 화합물을 신속하게 확인하기 위해 Spark 시약 데이터베이스 중 하나를 검색하는 실험을 수행했습니다. Sulfonamide는 primary aromatic amine fragments(amine moiety를 유지)을 검색하여 형성되었으며, 약 24,000 개의 fragment가 이용되었습니다6. 이 실험에서는 필터를 적용하지 않았습니다. 기본 5 Å 도킹 계산 grid가 이용되었으며, cofactor와 수소 결합을 형성할 수 있는 화합물을 우선적으로 얻기 위해 NAD oxygen 원자에 도킹 구속 조건이 추가되었습니다.
이 실험의 결과로서 구속한 NAD 산소 원자 및 다른 단백질 잔기와의 수소 결합을 형성할 수 있는 흥미로운 sulfonamide 화합물(표 2)이 추가로 확인되었습니다.
흥미롭게도, 도킹 스코어가 6번째로 좋은 화합물은 Sabbah 등이 이 포켓에 대한 최적의 fragment로 확인한 것과 동일한 fragment를 가지고 있습니다. 그림 5에 나타낸 것처럼 이 화합물에 대한 Spark 결과는 말단 amine이 도킹 중 구속된 NAD oxygen 원자 및 근처 backbone carbonyl과 수소 결합을 형성하는 것을 보여줍니다.
표 2. Phase 3에서 확인된 상위 6개 fragment를 나타낸 Spark Results 테이블
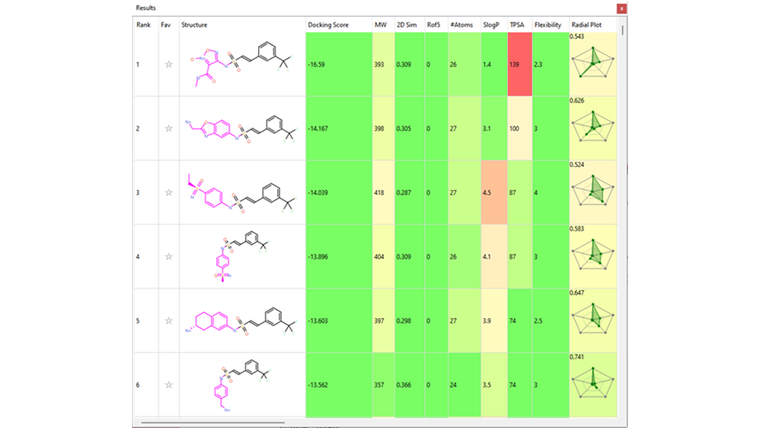
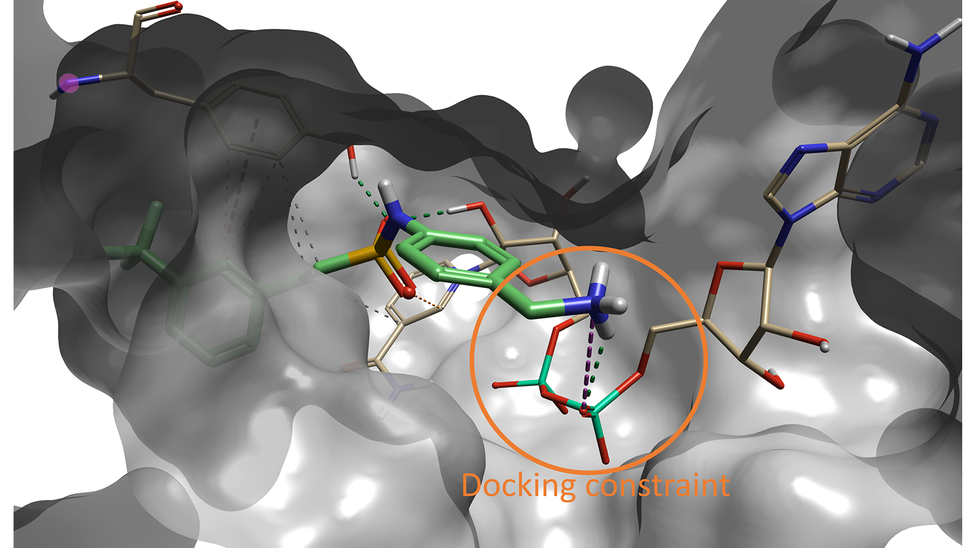
그림 5. Spark의 Compound #6(500개 중)은 Sabbah 등의 문헌4에서 확인된 lead 화합물임.
결론
이 사례 연구에서 우리는 Spark의 도킹 방법을 이용하여 PDB: 6SQ5의 starter 리간드를 InhA 활성 부위의 비어 있는 영역으로 성장시켜 InhA TB 타겟에 대해 발표된 실험 결과와 일치하는 새로운 리간드-단백질 결합 특성을 갖는 설계 아이디어를 확인했습니다.
도킹 방법은 Spark의 기능을 확장하며 활성 부위와의 원하는 상호작용에 대한 사전 리간드 정보가 없는 경우에도 fragment growing 실험을 실행할 수 있습니다.
필터 또는 구속 조건의 적용은 Spark 결과를 사용자가 원하는 물리 화학 및 반응 공간에 집중시키는데 큰 도움이 됩니다. 이번 사례에서는 화학 합성의 용이성과 활성 부위 내 새로운 상호작용 형성을 통한 고유 효능을 높이는 데 우선순위를 두었습니다.
이 사례 연구에서 이용된 워크플로는 활성 부위의 한 영역과 두 개의 관련 화학 하위 계열(subseries)에만 집중하여 lead를 생성했습니다. 이 워크플로는 대체 시약을 검색하거나 초기 fragment hit의 carboxylic acid의 대체 bioisostere로부터 시작하여 더욱 확장될 수 있습니다. 또한, 활성 부위의 다른 영역을 타겟하는 화합물을 탐색하고, 라이브 신약 개발 프로그램의 접근 방식과 유사하게 결합 부위를 전산적으로 조사하여 대체 화학 계열을 확인하여 추가 개발을 위한 fragment hit을 확장할 수 있습니다.
참고 문헌
- Spark™ V10.7.; Cheeseright T., Mackey M., Rose S., Vinter, A.; Molecular Field Extrema as Descriptors of Biological Activity: Definition and Validation, J. Chem. Inf. Model. 2006, 46 (2), 665-676,
- Lead Finder™ (accessed 12 May 2022); BioMolTech®, Toronto, Ontario, Canada
- Cresset science overview (accessed 12 May 2022)
- M. Sabbah, V. Mendes, R. G. Vistal, D. M. G. Dias, M. Záhorszká, K. Mikušová, J. Korduláková, A. G. Coyne, T. L. Blundell and C. Abell; Fragment-Based Design of Mycobacterium tuberculosis InhA Inhibitors, J. Med. Chem., 2020, 63, 4749–4761
- ChEMBL Database (accessed 12 May 2022)
- Products: Screening Compounds - eMolecules, (accessed 12 May 2022)