Nathan Kidley†
† Cresset, New Cambridge House, Bassingbourn Road, Litlington, Cambridgeshire, SG8 0SS, UK
概要
Forge™ は構造活性相関解析および分子デザインのためのCresset社のリガンドベース中心の創薬支援ソフトウエアです1。Forgeは、Cressetの3D-QSARの実装であるField QSARまたは他の機械学習手法によって、信頼性・予測性の高い定量的構造活性相関(QSAR)モデルを構築します。共通の結合モードをもち適度な結合力または活性値の分布があれば、どのようなデータセットに対しても一定のモデルを構築することができます。この事例紹介では、Janusキナーゼ JAK1およびJAK2阻害活性をもつ196個の分子からなるデータセットを使用し、Forgeによる活性予測機械学習およびField QSARモデルを構築します。QSARモデル情報ツールを使用して予測性能を最大化するモデルを選択し、3D表示でモデルを視覚化してその解釈を支援します。
はじめに
Janusキナーゼは免疫および悪性疾患関連の活発な研究領域です。トファシチニブ(Tofacitinib)は、関節リウマチ、強直性脊椎炎、潰瘍性大腸炎の治療薬として使用されるJAK阻害薬です。 トファシチニブには有害な副作用のため高用量では使えず、副作用を軽減する改良が強く望まれています2。 本事例紹介では、Merck社の特許(US9394282)3およびSuiらの論文4のデータを利用します。彼らの研究の焦点は、トファシチニブよりも最大用量が高くより効果的な関節リウマチの治療薬を設計することです。またトファシチニブは汎JAK阻害薬であり、臨床研究の中でJAK2活性が原因で貧血を誘発することがわかっており、優れたADMEプロファイルとともにJAK1/JAK2選択性の向上が求められます。
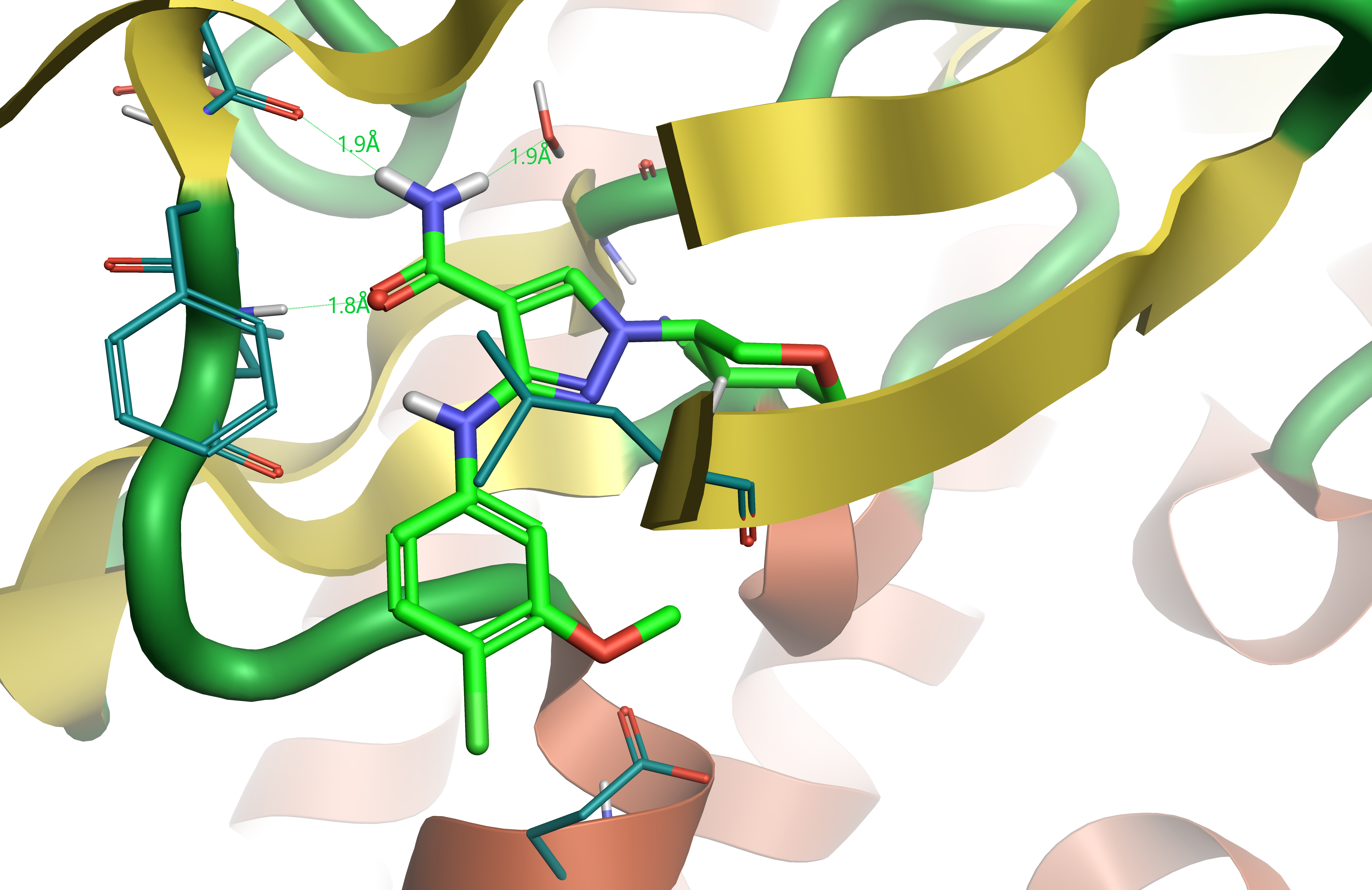
図1. JAK1活性部位(PDB: 5WO4)に結合したCompound 28 (破線は水素結合)
Cressetのfield pointは、分子の静電ポテンシャルおよび体積に基づき設定され、分子認識の鍵となる分子周囲の空間領域に関する情報を与えます。ForgeのQSAR手法は、まず分子を重ね合わせ、トレーニングセットの各分子のfield pointを集約したプローブの座標を用いてQSARモデリングのための記述子を作ります。
よいQSARモデルを構築するには、モデルを構築するための高精度のデータが必要です。 3D記述子を用いる上で、各分子の適切な配座と全分子にわたる確かな重ね合わせが必要であり、それ自体難しい問題です。通常QSARでは、共通部分構造または共通テンプレート分子に対し全分子を緻密に重ね合わせ、静電場と体積の違いによる活性への影響を明確に捉えようと努めます。
Forgeは、多くの設定項目を伴ってfield pointによる手法5または最大共通部分構造(MCS)による手法いずれかを選択し、精度の高い重ね合わせを実現します。いずれの手法においても、fieldとファーマコフォアの制約を加えれば、作用中心または部分構造をよりよく重ね合わせることができます。手作業での重ね合わせの修正を必要とする場合もあります。ただし、活性・非活性化合物を区別して意図的に分子を配向させるようなモデルを作ってしまうことがないよう注意が必要です。
化合物の配座生成と重ね合わせ
Compound 28 (表1) は問題化合物の類似体です。化合物28とJAK1のタンパク質-リガンド複合体の結晶構造 (図1) 4は、リガンド重ね合わせのレファレンス分子としてとても貴重な活性配座の情報を提供します。
化合物US9394282, 6-3およびUS9394282, 6-36を、上のX線構造のリガンドに最大共通構造で重ね合わせ、トレーニングセット内の化合物の重ね合わせを全体的に調整するための追加レファレンスとして利用します。
配座生成パラメーターは、標準の「Accurate but slow」、標準のMCSアラインメント法、タンパク質の排除体積の厳格度を「ソフト」に設定しました。
統計分析と結果
196化合物のデータセットを、80%のトレーニングセット(157化合物)と20%のテストセット(39化合物)に分割しました。 196化合物全てにJAK1 pIC50の実験値があり、そのうち172化合物についてはJAK2 pIC50の実験値もあります。
QSARモデル構築のデフォルト選択は、利用可能な機械学習方法を全て試みた上で、テストセットの予測値の統計的最適なモデルを選択します。ここでは、さらにField QSARモデルも合わせて構築し、機械学習手法の予測精度との比較をしました。 JAK1およびJAK2阻害活性について、機械学習メソッドごとの結果を表2に示します。
この例では、全てのモデルでトレーニングセットとテストセット傾向はよく似ています。テストセットの統計的パフォーマンスを比較すると、ランダムフォレストモデルがやや優れているように見えます。Forgeによって作成されたモデルは、いずれも新しい化合物の活性を予測するのに十分適しています。それに加えてField QSARは、モデルの解釈に視覚的検証を加えることができるという利点があり、活性の向上に寄与する化学的要素を理解し、新しい化合物の設計をする上で大いに役立ちます。
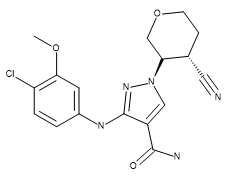
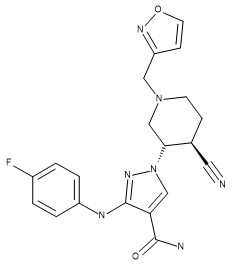
表1. 化合物28 (結晶構造配位子)、US93942, 6-3、およびUS9394282, 6-36の立体化学構造
| Compound 28 | US9394282, 6-3 | US9394282, 6-36 |
 |
 |
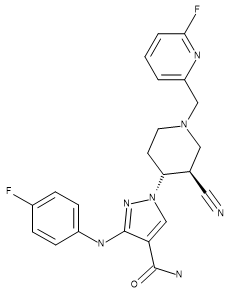 |
表2. QSARモデルの実験と予測統計の比較
| Model | Data set | R2 for JAK1 activity | R2 for JAK2 activity |
| Field QSAR |
Training |
0.792 | 0.794 |
| Cross validation | 0.589 | 0.541 | |
| Test | 0.634 | 0.586 | |
| KNN | Training | 0.6 | 0.536 |
| Test | 0.626 | 0.503 | |
| Random Forest | Training | 0.906 | 0.902 |
| Cross validation | 0.524 | 0.521 | |
| Test | 0.655 | 0.622 | |
| Relevance Machine | Training | 0.778 | 0.745 |
| Cross validation | 0.556 | 0.545 | |
| Test | 0.589 | 0.623 | |
| Support Vector Machine | Training | 0.83 | 0.788 |
| Cross validation | 0.55 | 0.526 | |
| Test | 0.636 | 0.625 |
モデルの視覚化と解釈
ForgeのField QSAR手法は、部分最小二乗法分析(PLS)に基づく回帰法であり、記述子と活性値の線形関係をもとに、視覚的にも可能なモデルの解釈を試みます。 「モデル係数」プロットの点の大きさは、その位置に共通の特徴を有する化合物と静電場/立体が実際の活性とどれだけ相関関係があるかという程度を表しています。
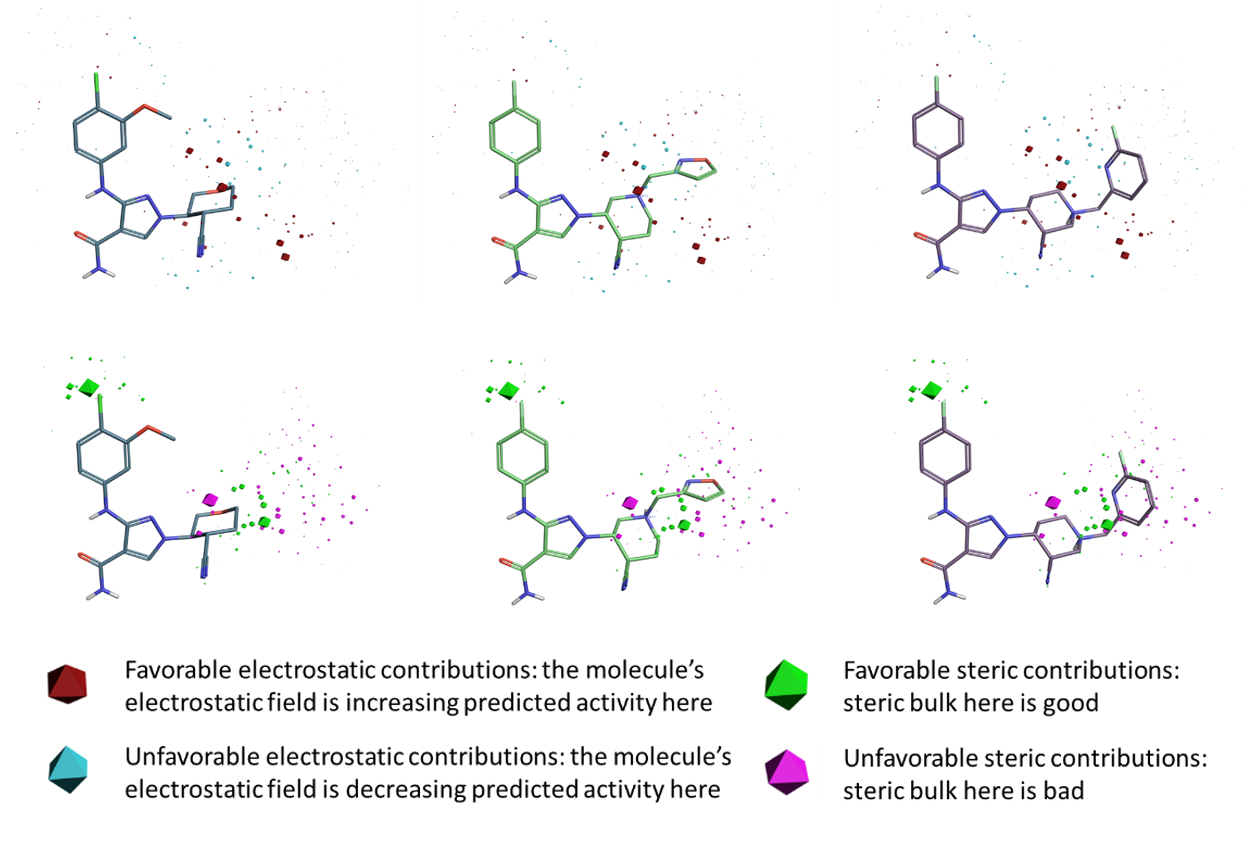
図2. JAK1 Field QSARモデルの係数プロット 上段: 静電係数 下段:立体係数
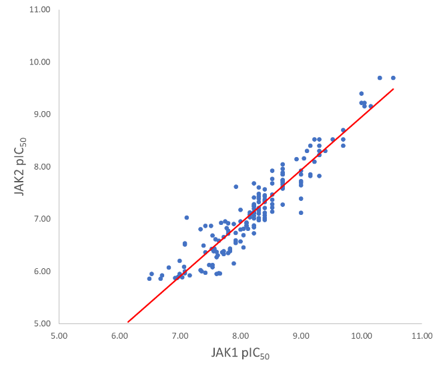
図3. JAK1とJAK2のpIC50活性の相関関係 回帰決定係数r2は0.89
JAK2よりもJAK1への活性がより高い傾向
PLSの5成分をとったJAK1阻害活性モデルでは、静電場および立体のモデル係数に良い定性的な相関が見いだされます(図2)。このField QSARモデルを俯瞰すると、ピペリジン/テトラヒドロフラン環と末端イソキサゾール/ピリジン環の間の中間領域には、静電場係数または立体場係数の活性に対する強い相関は見つけられません。これは、データセット内の分子の骨格環部分の配座の違いがあり、精緻な重ね合わせが困難であることが一つの原因だと考えられます。
静電場係数 (上段)は立体場係数 (下段)の多面体のサイズはQSARモデルでの活性への寄与を示しています。図からわかるように、全般的に静電場係数は小さいことがわかります。最も重要な正の静電場係数は、ピペリジン環の同じメタ位置の近くにあります。
ピリジン環とイソオキサゾール環近くに立体場係数のピンクの多面体が多く散らばっていますが、このように一定の領域に負の立体場係数が多く見つかるのは、この領域に大きな置換基があると活性発現に不利になるということを意味します。また、ベンゼン環の塩素原子の近くに、好ましい立体場係数の領域(図2の下段の緑色の多面体)が確認できます。もう一つの良好な立体場係数の領域は、ピペリジン環のパラ位の近くにあります。一方、ピペリジン環の左メタ位には好ましくない立体の領域(マゼンタ多面体)があります。
JAK2のin-vitroデータでもほぼ同じパターンが観察され、JAK1とJAK2の活性データの間に高い相関関係があることは不思議ではありません(R2 0.89、図3)。アッセイ結果は、このデータセット内の2つの化合物以外はJAK2よりもJAK1に対して有意に選択的であり、全体的にJAK1への選択性が高い傾向があります。 Siu et al4は、JAK1のE966とJAK2のD939の一つのアミノ酸の違いが、化合物の選択性と深く関わっていることを見出しています。この事例紹介で使用されたデータセットでは、ベンゼン環の置換基の多様性はあまりなく、QSARモデルはそこに強いSARシグナルを検出できません。一方で、Actiivity Atlasは探索された化学空間を定性的に精査するために利用され、立体的および静電的観点両方からパラクロロ置換基の高い活性への寄与を確認できます。また、Activity Atlasはメタ位とパラ位の静電的特徴がデータセットの中では十分に標本されていないことも教えてくれます。
結び
Forgeを利用することによって、196個のJAK1 / 2キナーゼ阻害薬のデータセットを用いて様々な3D-QSARモデルを構築することができました。 Activity AtlasおよびField QSARモデルは、モデルの視覚化と解釈を容易にし、探索された空間、活性に寄与する要素、およびデータセットの制限についての様々な情報を提供します。 この情報と活性予想モデルをうまく活用することは、新しい化合物の設計を容易にし、合成とテストに向けた優先順位をつけることことに大いに役に立ちます。
よいQSARモデルを作成するためには、データの化学的共通性が十分にあり、活性の分布が十分に広いことをよく確認する必要があります。 結果が過剰なパラメーター化による偶発的なものではないことを確認して選択されたモデルは、信頼性が高く、本質的に内挿的でなく外挿的である活性予測に威力を発揮します。ただし、3D-QSARモデルは、配座探索と重ね合わせをいかに正確に行うかという難しい問題を含んでいることに留意しないとなりません。
Forgeの評価利用
Forgeでは柔軟なライセンスオプション を用意しておりますので、実際の創薬プロジェクトでご試用・評価していただくことができます。ご関心のある方はぜひリクエストをお寄せください。
参考文献とリンク
- https://www.cresset-group.com/software/forge/
- Flanagan et al., J. Med. Chem. 2010, 53, 8468-8484
- Patent US9394282B2
- Sui et al., J. Med. Chem. 2017, 60, 9676-9690
- Cheeseright et al, J. Chem. Inf. Model., 2006, 46, 66