The adaptability of pyFlare to access advanced data visualization and calculation functionalities
We showcase some examples within a drug discovery context where the pyFlare environment has been further expanded with advanced data ...
News
리간드 기반 및 구조 기반 신약 설계를 위한 통합 소프트웨어 플랫폼인 Flare에서는 사용자가 electrostatic 및 shape 기반 3D descriptor를 이용하여 3D-QSAR 모델을 구축할 수 있습니다. 이들 모델에서는 데이터 세트 내 리간드들이 생물학적 표적의 활성 부위에 대해 공통 결합 모드를 공유한다고 가정합니다.
그러나 많은 경우에 설계자는 특정 결합 포즈나 이벤트가 없는 생물학적 특성에 대해 QSAR 모델을 구축하기를 원할 수 있습니다. 예를 들어, 그들은 활성 부위 결합 이벤트가 없거나 모든 리간드가 일관성이 없는 다양한 리간드 세트를 이용하여 ADMET 종점(endpoint)을 모델링하고 싶을 수 있습니다. 이러한 상황에서는 physico-chemical descriptor(예를 들면, 분자량(MW), logP, Topological Polar Surface Area(TPSA), 및/또는 회전 가능 결합 수와 같은 2D descriptor)를 대안으로 이용해야 합니다.
최신 Flare 업데이트에서는 RDKit 2D descriptor1 생성 기능이 완전히 통합되었으며 QSAR 모델 구축에 RDKit fingerprint1를 이용할 수 있는 기능이 추가되었습니다. 이전 기사에서는 사용자가 파이썬 스크립트를 이용하여 Flare로 가져온 2D descriptor를 이용하여 Blood-Brain Barrier (BBB) 투과성 QSAR 모델을 어떻게 구축할 수 있는지 소개했습니다. 이번 특집 기사에서는 RDKit descriptor를 빠르고 쉽게 생성하는 새로 추가된 기능을 이용하여 이전 기사를 다시 살펴볼 것입니다.
이 연구에서 이용된 BBB 데이터 세트는 Roy 등의 문헌2에서 가져온 분류 데이터 세트입니다. 결과 응답은 두 가지 범주로 분류됩니다: 분자가 BBB를 통과할 수 있으면 1의 값을 받고, 통과할 수 없다면 0의 값을 받습니다. 우리는 이러한 범주를 이용하여 분류 QSAR 모델을 구축할 것입니다.
먼저, 전체 데이터 세트를 7:3의 비율로 training 세트(1284개 리간드)와 test 세트(551개 리간드)로 나누었습니다. 그런 다음, ‘Column & Activity Editor’ 내 ‘Import RDKit descriptor’ 버튼을 클릭하여 RDKit 2D descriptor를 생성하였습니다. 사용자는 ‘Select All’을 선택하여 전체 RDKit descriptor를 가져오거나 체크 박스에 체크 표시하여 원하는 descriptor만을 가져올 수 있습니다.
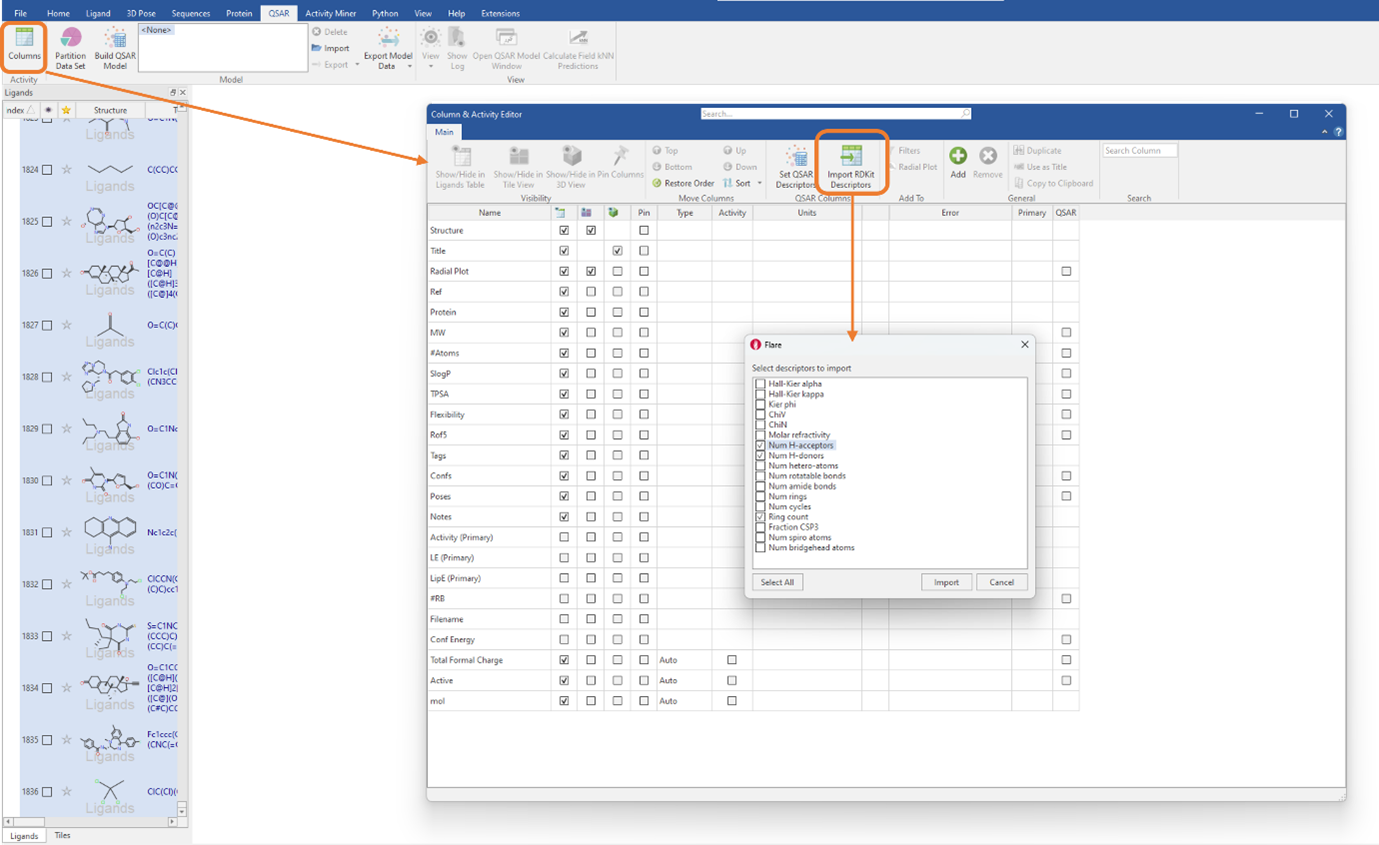
그림 1. ‘Column & Activity Editor’ 내 단일 버튼을 이용한 RDKit 2D physico-chemical descriptor 가져오기
가져온 각 RDKit descriptor는 Ligands 테이블에 molecular property 열로 추가될 것입니다. 이들 descriptor를 QSAR 모델 구축에 이용하기 위해서는 테이블에서 descriptor를 선택한 다음 Column & Activity Editor 내 ‘Set QSAR Descriptors’ 버튼을 클릭해야 합니다. 이 실험에서는 MW, SlogP, TPSA, Flexibility, #RB, Total Formal Charge, NumHAcceptors, NumHDonors 및 RingCount descriptor를 이용합니다. 후자 3개의 descriptor는 RDKit에서 가져온 것들입니다(그림 1).
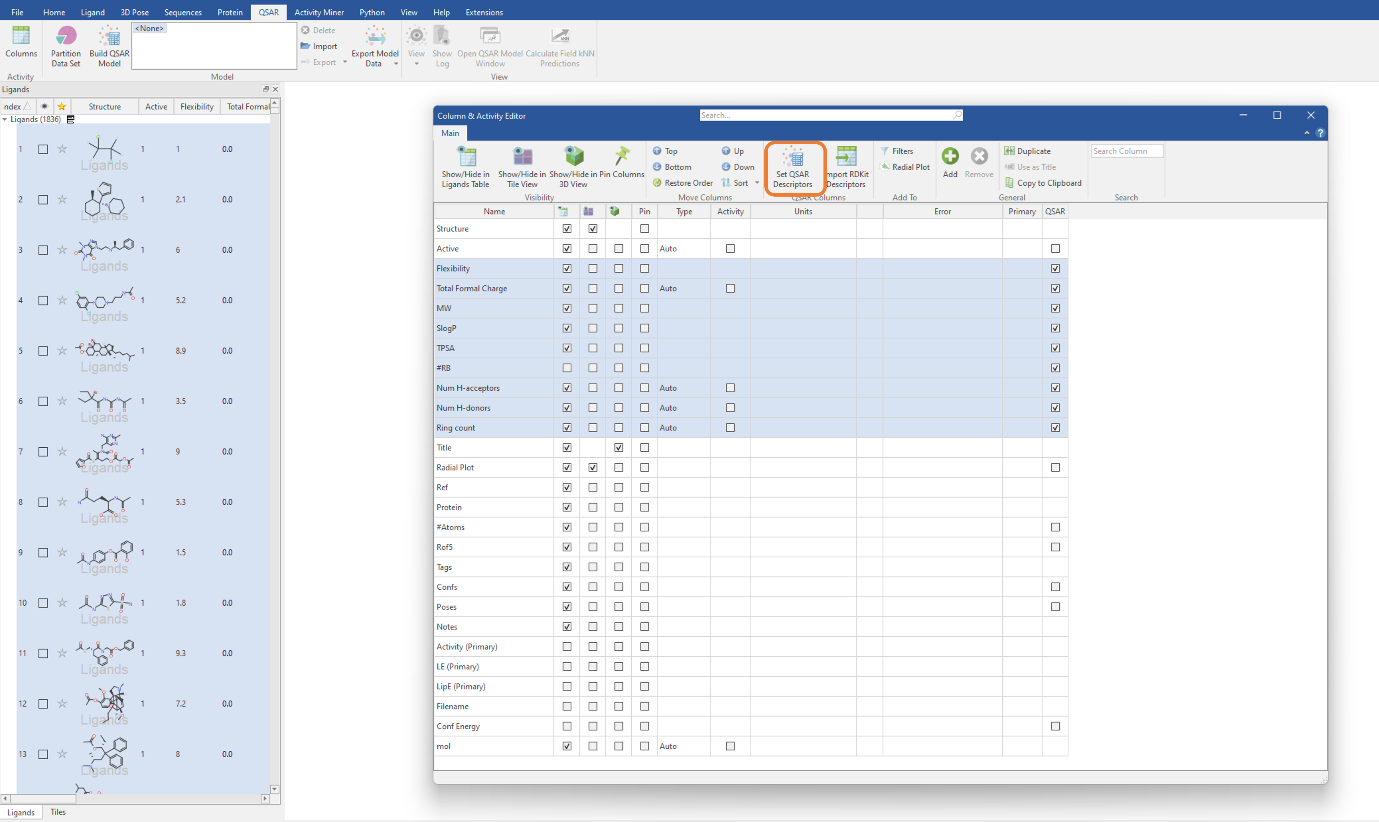
그림 2. 분류 QSAR 모델에 이용할 가져온 RDKit descriptor 선택하기
이전 연구와 유사하게, 상기 Flare 생성 및 가져온 RDKit descriptor의 조합을 이용하여 SVM 분류 QSAR 모델을 구축할 것입니다 (그림 2). ‘Electrostatic’ 및 ‘Volume’ 체크 박스에는 체크 표시가 해제되어 있는지 확인해야 합니다. 또한, electrostatic 및 shape field point 3D descriptor를 이용하지 않으므로 리간드 데이터 세트를 align할 필요가 없습니다 (그림 3).
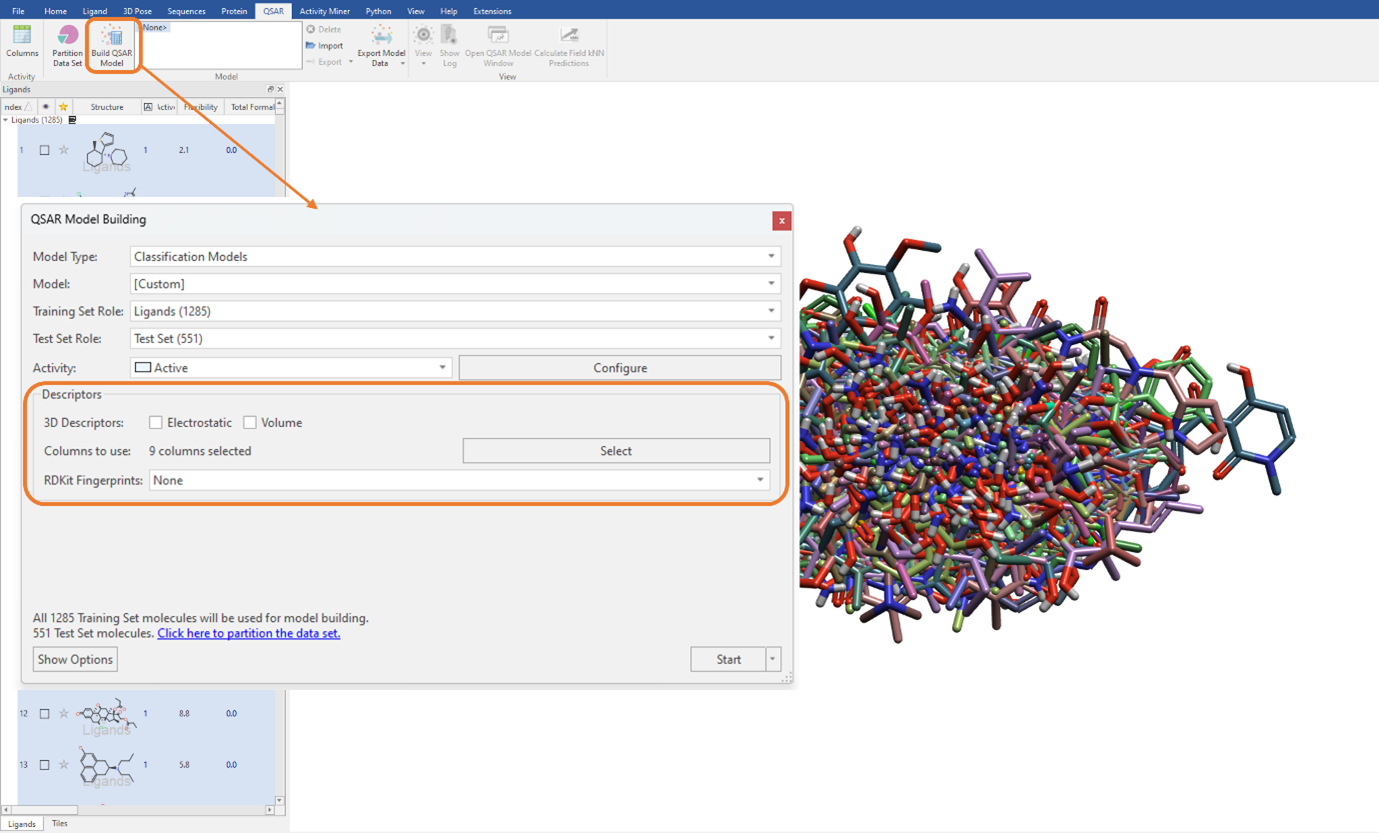
그림 3. Flare 생성 및 가져온 RDKit descriptor의 조합을 이용한 SVM 분류 모델 구축
Maximum number of optimizer iterations 옵션을 “no limit”으로 설정하여 QSAR 모델 구축 계산을 실행할 수 있습니다. 이 설정은 대규모 데이터 세트의 경우 기본 설정이지만 ‘Show Options’ 메뉴에서 직접 변경도 가능합니다 (그림 3). 분류 QSAR 모델의 성능, 즉 분류 모델이 테스트 세트 내 리간드에 대해 얼마나 정확하게 예측하는지는 Test Set Confusion Matrix로 요약됩니다(그림 4). 행렬의 비대각 요소는 범주 0에 속한 리간드가 범주 1로 분류되거나 그 반대와 같은 오분류의 빈도를 나타냅니다. 0의 비대각 값은 분류 QSAR 모델이 테스트 세트 내 각 리간드를 정확한 범주로 모두 분류했음을 의미합니다.
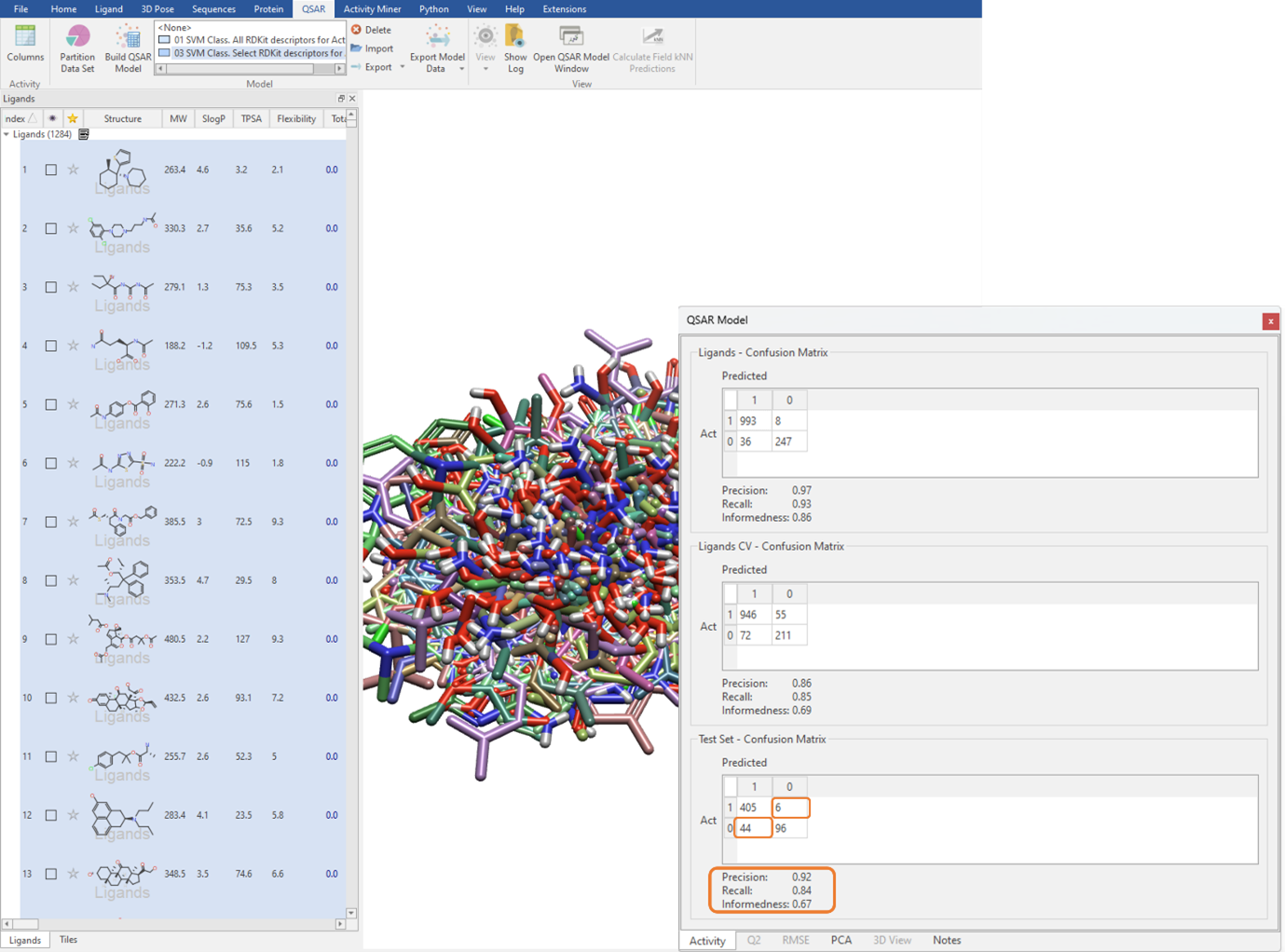
그림 4. Test Set Confusion Matrix를 이용한 분류 QSAR 모델의 예측 성능 평가 및 확인
Precision, Recall, 및 Informedness는 Confusion Matrix로부터 산출된 통계 평가지표(metrics)입니다(그림 4). Precision은 정확한 분류 비율을 나타냅니다. Recall은 이를 더욱 세분화하여 특정 범주에 대한 정확한 분류 비율을 보고합니다. 기본적으로는 범주 1에 대한 정확한 분류 수를 범주 1 리간드의 전체 수로 나눈 값입니다. Informedness는 무작위 추측이 아닌 정보에 입각한 의사 결정이 내려질 확률을 나타냅니다. 이들 값이 1에 가까울수록 분류 QSAR 모델에 대한 신뢰도가 높아집니다.
그림 4에서 테스트 세트에 대한 Precision, Recall, 및 Informedness는 각각 0.92, 0.84, 0.67로, 신규 리간드에 대해 BBB 투과성인지 또는 비투과성인지를 분류하는데 이 분류 QSAR 모델을 이용하는 것에는 통계적 확신을 가질 수 있습니다. 또한, Test Set – Confusion Matix에서 볼 수 있듯이 활성(투과성)인 6개의 리간드는 비활성(비투과성)으로, 비활성(비투과성)인 44개는 활성(투과성)으로 예측됩니다.
이 결과에서는 44개의 거짓 양성(false positive)을 예측하고 있지만, 거짓 음성(false negative)은 6개에 불과하다는 것을 시사합니다. 즉, 이는 BBB 투과가 원하는 특성인 점을 고려할 때 실험을 위한 분자의 우선순위를 정할 시에 잠재적으로 BBB를 투과할 수 있는 및 관심 있는 분자를 놓칠 가능성이 낮다는 의미입니다.
그림 3에서는 QSAR 모델 구축 시 descriptor로서 RDKit fingerprint를 이용하는 옵션도 있음을 확인할 수 있습니다. RDKit fingerprint는 분자의 유사성를 평가하는데 이용되는 substructure fingerprint입니다. 이 옵션을 선택하는 것으로 이들 fingerprint descriptor를 선택된 QSAR descriptor에 추가하거나 대체하여 이용할 수 있습니다. 2D physico-chemical descriptor(그림 1)처럼 ‘RDkit Fingerprints’ 드롭 다운 메뉴(그림 5)에서 원하는 옵션을 선택하는 것으로 모델 구축에 손쉽게 이용할 수 있습니다. 현재 선택 가능한 것에는 이 사례에서 이용한 ‘RDKit’ fingerprint와 Morgan fingerprint 및 MACCS key가 있습니다.
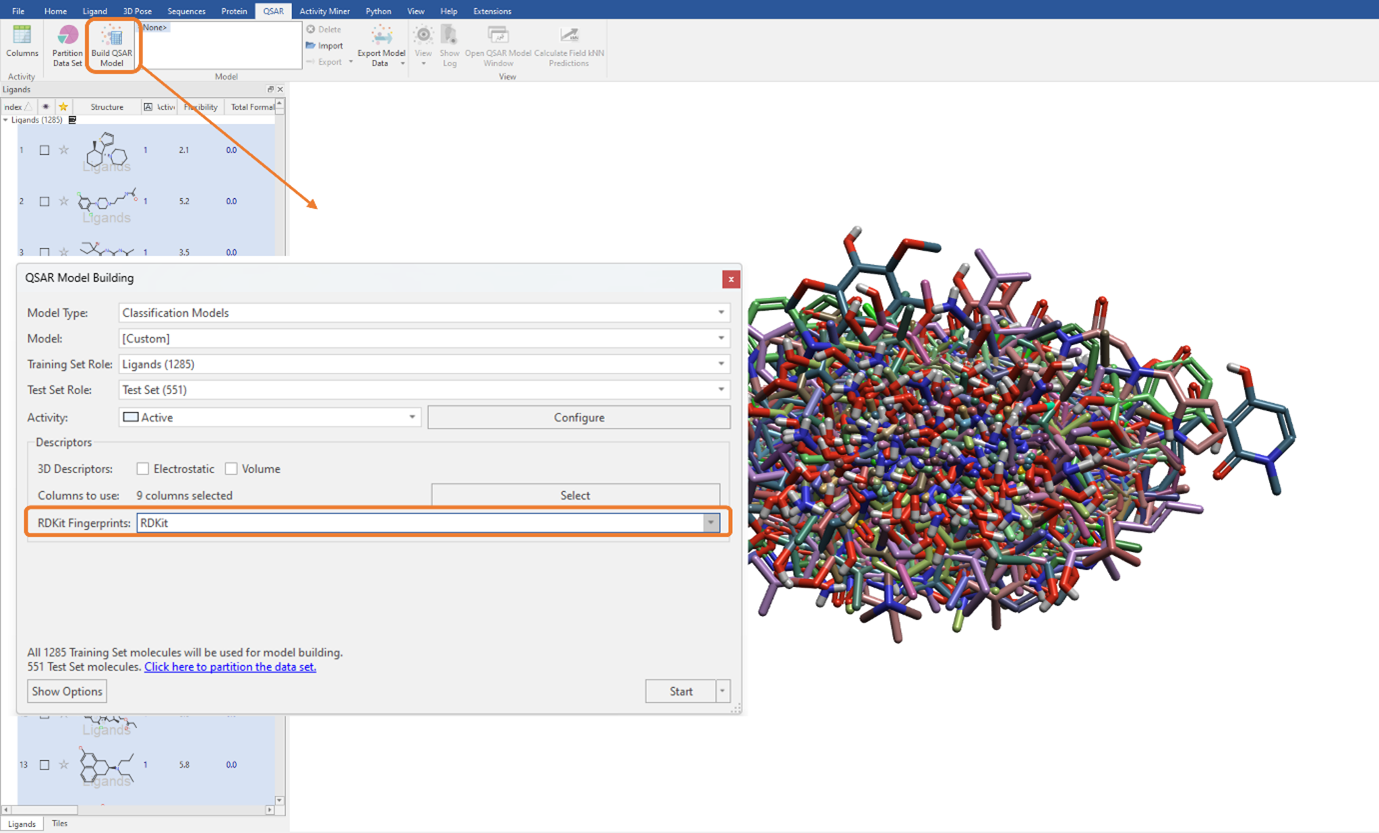
그림 5. RDKit fingerprint를 이용한 분류 QSAR 모델 구축. QSAR 모델 구축 시 3D 및 2D descriptor는 비선택(‘Columns to use’는 0으로 설정)하고 RDKit fingerprint만을 이용함
RDKit fingerprint를 이용한 분류 QSAR 모델에서는 Precision는 0.90, Recall은 0.86, Informedness는 0.72 값을 얻었습니다. 이는 RDKit 2D descriptor를 이용한 분류 QSAR 모델과 비교해 볼 때 BBB 투과성 예측력에 약간의 개선이 있음을 알 수 있습니다.
Flare에서는 RDKit 2D descriptor와 fingerprint를 쉽고 빠르게 가져와 QSAR 모델 구축에 이용할 수 있습니다. 이번 사례에서는 이들 descriptor를 이용하여 BBB에 대한 리간드의 투과성을 예측하는 분류 QSAR 모델을 성공적으로 구축하였습니다. RDKit descriptor 또는 RDKit fingerprint는 활성 및 ADMET 특성 예측 QSAR 모델을 구축 시에 Cresset 3D descriptor의 대안으로 이용될 수 있습니다.
지금 Flare 무료 평가판을 요청하여 분자 모델링 기능의 전체 포트폴리오를 자세히 살펴보십시오. 평가 과정의 일환으로 플랫폼 설치 및 다양한 기능에 액세스하는 데 필요한 모든 지원을 받을 수 있습니다. 또한, 결과물에 대해서는 논문 등에 자유롭게 게재하고 추가 연구에 이용할 수 있습니다.