The adaptability of pyFlare to access advanced data visualization and calculation functionalities
We showcase some examples within a drug discovery context where the pyFlare environment has been further expanded with advanced data ...
News
본 사례 연구는 Flare1의 Electrostatic ComplementarityTM을 이용하여 X-linked IAP(XIAP)-caspase 단백질-단백질 상호작용을 저해하는 작은 화합물 세트에 대한 단백질-리간드 정전기적 상호작용과 구조-활성 상관 관계(SAR)를 연구하였습니다. 또한, 해당 데이터 세트에 대한 XIAP-BIR3 친화도와 Electrostatic Complementarity 스코어 간의 좋은 상관 관계를 얻었습니다.
세포자멸사 단백질 억제제(IAPs)는 항세포자멸사와 생존 촉진 신호 전달 경로의 핵심 조절인자입니다.2-4 이들의 deregulation은 각종 암에서 발생하며, 종양 성장, 치료 저항, 저조한 예후와 관련이 있습니다. 이러한 이유로 IAP는 항암치료제 개발의 매력적인 표적이 되고 있습니다.5-7 가장 잘 특성화된 IAP인 X-linked IAP(XIAP)는 BIR 도메인을 통한 caspase 3, 7, 9의 결합과 비활성화에 의해 항세포자멸사 활성을 발휘합니다. XIAP를 저해하는 유망한 전략은 XIAP-BIR 도메인과 caspase 간의 단백질-단백질 상호작용(PPI)을 저분자를 통해 방해하는 것입니다. 그러나, PPI 약물 개발은 특히 일반적으로 평평하고 큰 고전적인 결합 부위와는 다른 특이한 결합 계면으로 인해 도전적인 과제가 될 수 있습니다.8
Astex의 문헌9은 표 1의 작은 길항제(antagonist) 데이터 세트에 대한 XIAP-BIR3 활성이 indoline에 전자를 끌어당기는 그룹을 도입함으로써 증가한다고 보고하며, XIAP-BIR3 pIC50와 Hammett’s σp 간의 좋은 상관 관계를 보여주었습니다.
본 사례는 Flare에서 Electrostatic Complementarity 맵을 이용하여 표 1의 분자에 대한 단백질-리간드 정전기적 상호작용과 SAR를 연구했습니다. 계산된 Electrostatic Complementarity 스코어는 XIAP-BIR3 pIC50를 정량적으로 모델링하기 위해 이용했습니다.
표 1. C-6 치환된 indoline8의 XIAP-BIR3 결합 친화도
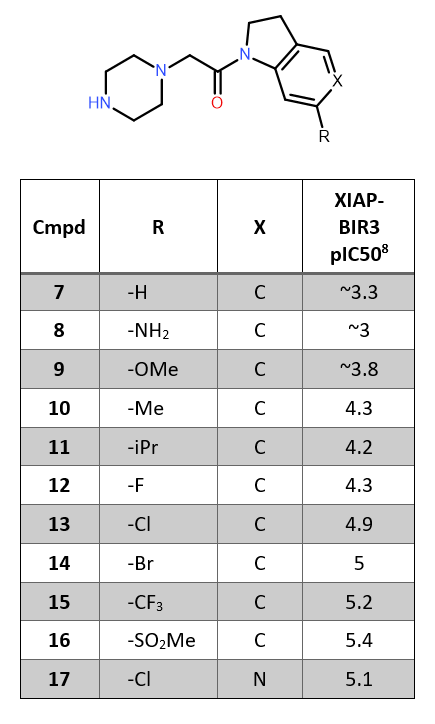
5C7A 리간드-단백질 복합체를 RCSB 사이트에서 Flare로 다운로드 하고, BioMolTech사11의 Build Model10 도구를 이용하여 수소 원자 복원, 수소 결합 네트워크 최적화, 원자간 충돌 회피, 적절한 양성자화 상태를 적용한 단백질 구조를 준비하였습니다. 구조 준비 과정 중 잘린 단백질 사슬은 모두 capping 처리하였습니다. 육안으로 결합 부위 내 리간드와 주위 아미노산 곁사슬이 적절한 양성자화 상태를 갖는지 확인하였고 물의 수소 결합 네트워크가 최적이 되도록 물의 배향을 다시 최적화하였습니다. 정전기적 상보성 계산에서는 단백질과 최소 2개의 수소 결합 또는 리간드와 단백질에 최소 1개의 수소 결합을 갖는 결합 부위 안과 결합 부위에서 가까운 물 분자만을 고려하였습니다. 모델링한 많은 결합 모드(예를 들어, 화합물, 9, 11, 15, 16)가 Lys297 잔기(그림 1)의 유연한 곁사슬과 충돌하기 때문에 각 리간드에 대해 해당 곁사슬 원자를 XED 힘장12를 이용하여 에너지 최소화 계산을 수행하였습니다. 생성한 수용체는 각 화합물의 정전기적 상보성을 계산하기 위해 이용하였습니다.
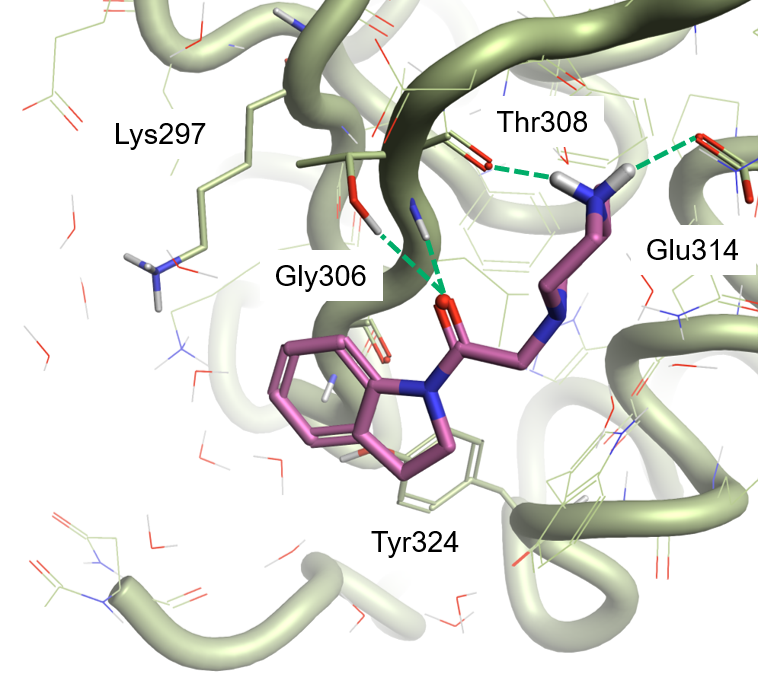
표 1의 화합물을 PDB: 5C7A의 리간드 결정 구조(표 1의 화합물 7)로부터 Flare의 molecule editor를 이용하여 그렸습니다. 그런 다음, Forge(현재는 Flare에 통합됨)13에서 11개의 화합물을 5C7A 리간드에 align했습니다. 이 때, 공통 indoline-piperazine 골격에 conformation에 의한 노이즈를 최소화하기 위해 Maximum Common Substructure(MCS) 방법을 이용했습니다.
Electrostatic Complementarity 맵과 scoring 함수는 Flare의 Cresset XED 힘장을 기반한 Protein Interaction Potentials(PIPs)를 확장한 것입니다. 원자 중심 전하에 의존한 고전적 힘장과는 달리, XED는 일반적으로 ab initio 접근에서만 가능한 원자 주위의 이방성 전하 분포를 설명할 수 있습니다. 분극 효과와 원자 전하 이방성의 설명은 방향족 또는 불포화 탄화수소, sp2 혼성 산소 원자, sp 또는 sp2 혼성 질소 원자, 방향족 할로겐 (CI, Br, I의 sigma hole)의 정전기적 특성을 계산하는데 특히 유용합니다.14-16
관심 단백질에 대한 리간드의 Electrostatic Complementarity 맵의 계산은 먼저 용매 접근 가능 표면(SAS)이 리간드에 배치됩니다. 그런 다음, 표면의 각 꼭짓점에서 리간드와 단백질로 인한 electrostatic potential을 계산합니다.
이들 potential은 스케일링, 합산, 정규화를 통해 Electrostatic Complementarity 스코어를 산출합니다. 완벽한 상보성은 각 꼭짓점에 대해 리간드의 electrostatic potential 값이 단백질의 electrostatic potential 값과 크기는 같고 부호는 반대로 쌍을 이룬 것을 의미합니다.
단백질과 정전기적 상보성이 있는 리간드 표면 영역은 녹색으로 표시되며, 해당 상보성에 충돌이 있는 영역은 빨간색으로 표시됩니다. Electrostatic potential 및 상보성 방법론에 대한 자세한 설명은 다른 곳에서 확인할 수 있습니다.17
Electrostatic Complementarity 스코어는 다양한 단백질-리간드 시나리오에 적합한 서로 다른 세 가지 메트릭스로 리간드-단백질 정전기적 상보성을 정량화합니다.
첫번째 계산된 스코어(‘Complementarity’)는 리간드 표면에 대한 상보성 스코어의 정규화된 표면 적분입니다(사실상, 리간드 표면에 대한 해당 스코어의 평균값).
나머지 두 스코어(‘Complementarity r’과 ‘Complementarity rho’)는 각각 Pearson’s correlation coefficient와 Spearman rank correlation coefficient이며, 표면 꼭짓점에서 샘플링된 초기 리간드 및 단백질 electrostatic potential에서 계산됩니다.
세 가지 측정값 모두는 1(완벽한 상보성)에서 -1(완벽한 충돌) 사이의 범위를 가지지만 그 특성은 서로 다릅니다. Complementarity 스코어는 탈용매화 효과를 일부 감안하며, 따라서 이러한 효과가 클 경우 더 강력할 수 있습니다. Pearson과 Spearman correlation coefficient는 경우에 따라 리간드 활성을 더 잘 나타낼 수 있지만 노이즈에 더 민감합니다(r이 rho보다 더 민감). Spearman’s rho 값은 백그라운드 전기장에 더 강력하므로 계산된 단백질의 electric potential이 단백질의 큰 총 전하 때문에 편향된 경우 유용할 수 있습니다.
계산은 빠르며 예측 가능합니다: 100개의 리간드를 평가하는데 노트북에서 몇 분도 채 걸리지 않으며, 일반적으로 화합물 활성과 상관 관계가 있는 단백질-리간드 정전기에 대한 중요한 정보를 제공합니다.
XIAP 활성 부위 내 화합물 7의 Electrostatic Complementarity 맵(PDB: 5C7A, 그림 2 – 좌)은 indole 고리의 위쪽 영역에 강한 정전기적 충돌(빨간색)이 있음을 보여줍니다. 이는 Gly360 잔기의 주골격 carbonyl기와 Tyr324 잔기의 phenolic oxygen에 의해 발생된 단백질 활성 부위 내 음(-)의 electrostatic potential 영역(그림 2 – 중앙)이 indoline 고리와 관련된 음(-)의 정전기장(그림 2 – 우)과의 충돌로 인한 것으로 해석됩니다. Lys297 잔기의 양성자화된 곁사슬의 양(+)의 정전기장(그림 2 – 중앙)과 indoline 고리의 σ 수소의 양(+)의 정전기장(그림 2 – 우) 간의 덜 뚜렷한 정전기적 충돌도 볼 수 있습니다.
상기 맵(보고된 상관 관계와 일치8)에 의하면 전자 끄는 기는 indoline 고리를 덜한 electron-rich한 상태로 만드므로 XIAP와의 결합 친화도를 증가시킬 것으로 예상됩니다. 더 음(-)(또는 덜한 양(+))의 정전기장과 관련된 치환기는 Lys297 잔기의 양성화된 곁사슬과의 상호작용에 유리할 것입니다.
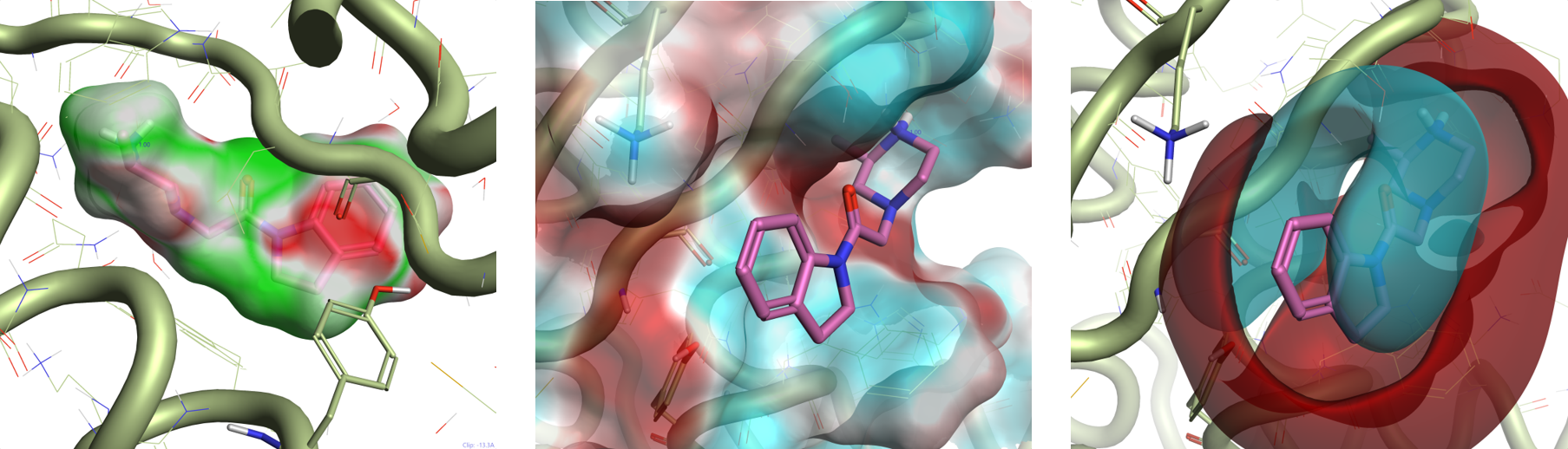
그림 3은 표1에 나와 있는 화합물의 Electrostatic Complementarity 맵을 XIAP-BIR3 활성이 증가하는 순(좌에서 우)으로 나타낸 것입니다.
전자 주는 기(-NH2, -OMe)에서 전자 끄는 기(-F, -Cl, -SO2Me)로 이동할 때 명확한 경향을 관찰할 수 있습니다. 전자 끄는 기는 indole 고리의 전자 밀도를 낮춰 XIAP 활성 부위의 음(-)의 정전기와의 충돌을 감소시킵니다.
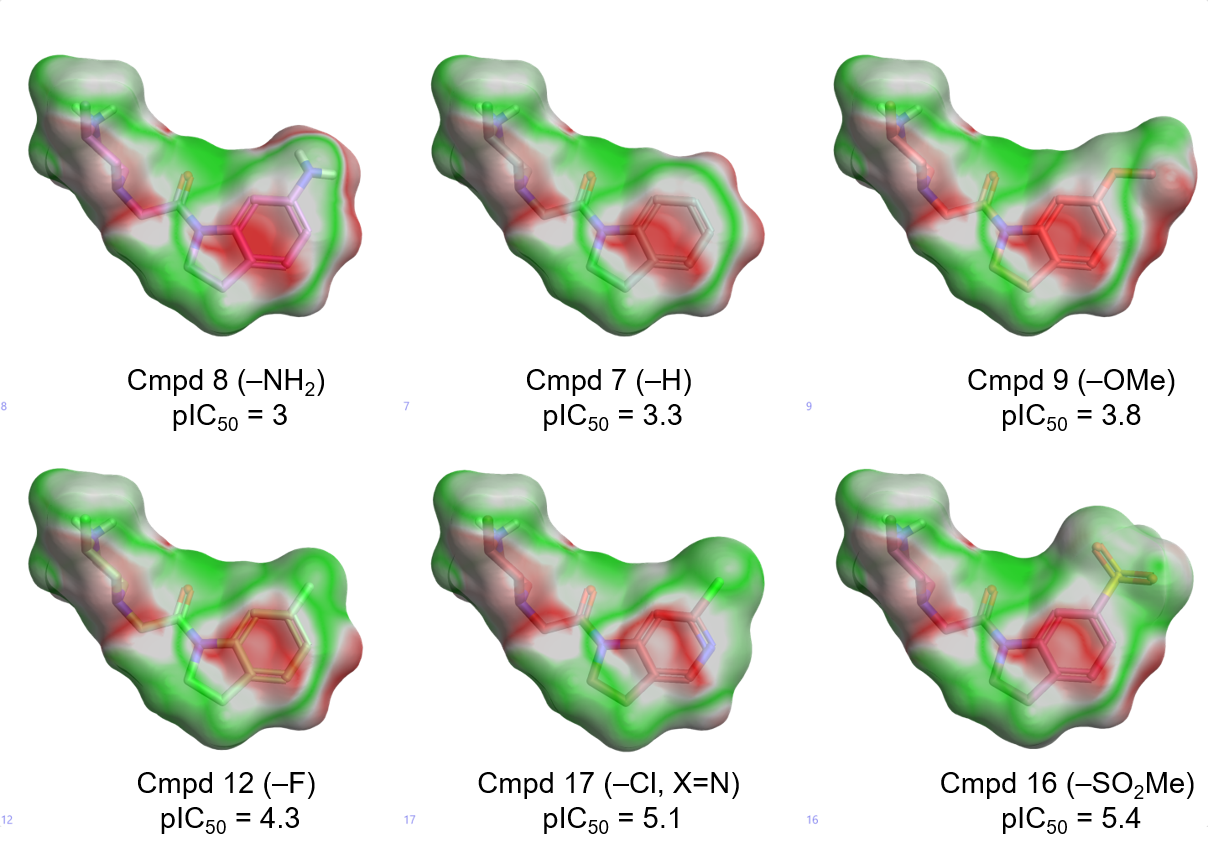
또한, 초기 가설에 따라 가장 효과가 좋은 화합물 3종의 치환기는 Lys297의 양성자화된 곁사슬과 유리한 상호작용을 나타내는 이들 자체의 음(-)의 리간드장(ligand field)과 관련이 있습니다(그림 4).
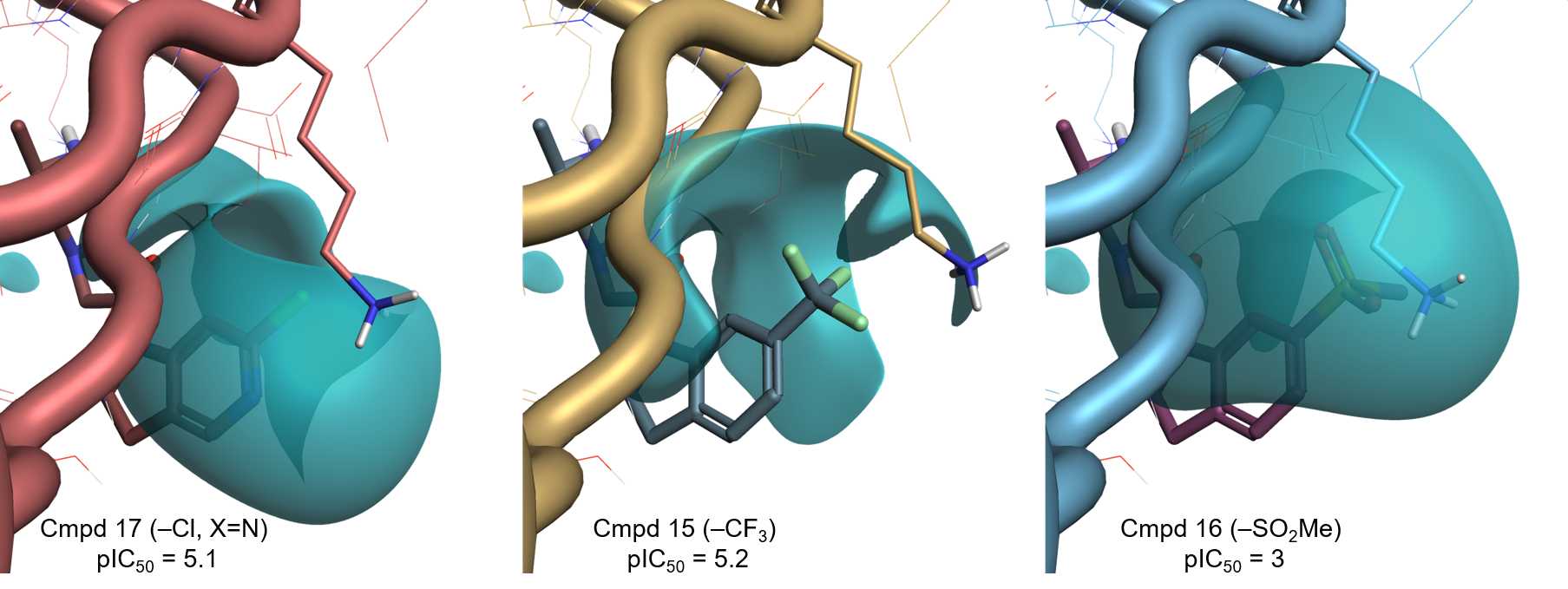
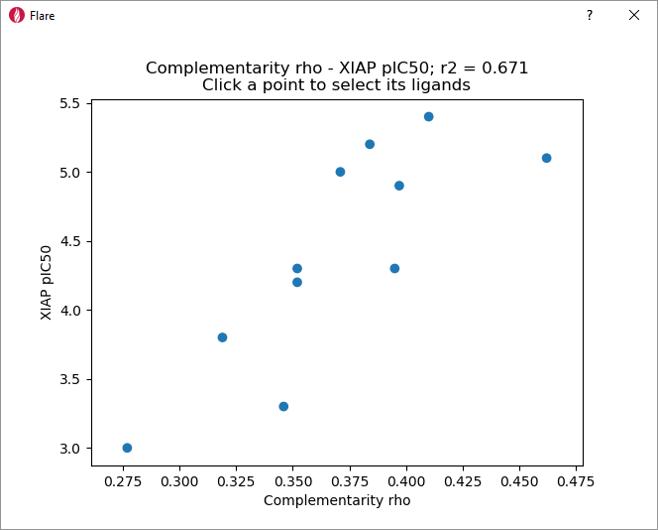
이러한 정성적 관찰은 그림 5에 나타낸 XIAP-BIR3 pIC50과 Complementarity rho 스코어 간의 좋은 상관 관계(r2 = 0.671)를 통해 확인할 수 있습니다.
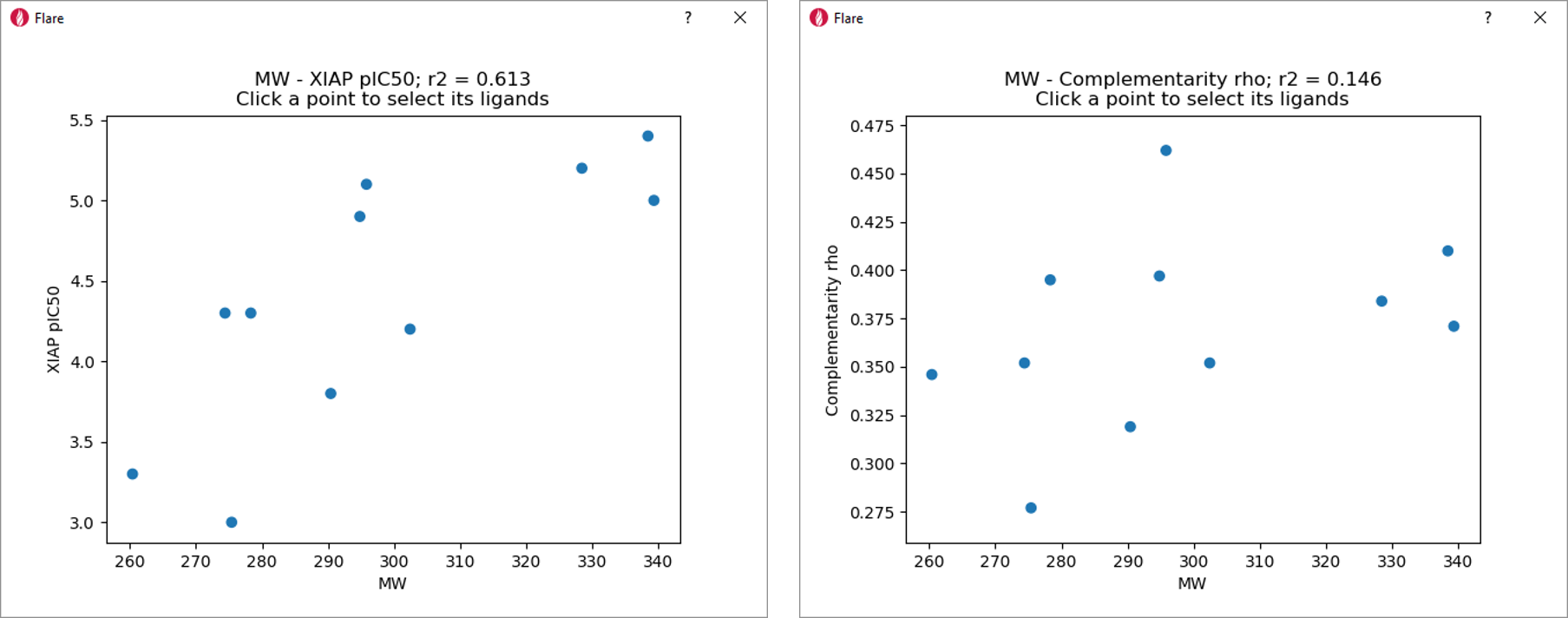
우리는 MW와 XIAP-BIR3 친화도/Complementarity rho 간의 상관 관계를 모니터링하여 Electrostatic Complementarity 스코어가 약물 설계에서 단순한 물리화학적 표현자(physico-chemical descriptor)를 이용하는 것 이상의 정보를 제공하는지 여부를 확인하였습니다.
MW와 XIAP-BIR3 pIC50 간의 상관 관계(r2 = 0.613, 그림 6 - 좌)는 데이터 세트에서 XIAP 친화도 변화에 대한 가장 단순한 해석으로 공간 채우기 효과를 가리킬 수 있습니다.
그러나, Complementarity rho와 MW 간의 낮은 상관 관계(그림 6 - 우)는 Electrostatic Complementarity 스코어가 분자 사이즈(분자량)와 무관함을 나타냅니다.
따라서, 정량적 SAR 모델링에 Electrostatic Complementarity 스코어를 이용하면 분자 사이즈 효과와 완전히 독립적인 추세를 생성할 수 있습니다.
또한, Electrostatic Complementarity 맵은 MW와 Hammett’s σp와 같은 종래의 단순한 물리화학적 표현자에서 얻을 수 없는 시각적 이해를 제공하므로 약물 설계에서 매우 유용한 정보를 제공합니다.
문헌에 보고된 XIAP-BIR3 데이터 세트에 Electrostatic Complementarity를 적용함으로써 우리의 방법이 생물활성 변화를 일으키는 XIAP 리간드 간의 정전기적 차이를 발견하고 이를 정량화 할 수 있음을 보였습니다. Cresset의 XED 힘장을 기반한 Electrostatic Complementarity 스코어 및 맵은 새로운 분자 설계에 대한 신속한 활성 예측과 시각적 피드백을 제공합니다. 또한, 리간드 결합과 SAR를 이해하는 데 유용한 정보를 제공하며 새로이 설계한 분자를 빠르게 순위 매김 하는 데 이용할 수 있습니다.