Improving PROTAC properties via single-point changes to linkers
We explore how computational methods can be applied to proteolysis targeting chimera (PROTAC) design, to effectively tackle some of the ...
News
Cresset의 CADD 워크벤치인 Flare V8에서는 리간드-단백질 복합체의 결합 자유 에너지 계산을 위한 신규 Molecular Mechanics/Generalized Born Surface Area (MM/GBSA) 방법, Flare FEP 적용 범위의 대폭 확대, GIST water analysis의 Grand Canonical Nonequilibrium Candidate Monte Carlo (GCNCMC), QM 계산의 HOMO/LUMO 오비탈 계산 및 시각화, library enumeration의 100가지 이상의 신규 반응, 단백질 및 homology 모델링의 새롭고 향상된 기능들을 포함한 신규 및 향상된 과학적 기능 및 방법이 제공됩니다.
이번 릴리즈에서는 Flare FEP 실험의 문제 해결과 분자동역학 trajectories의 분석을 위한 최신 시각화 도구들의 선택폭이 더욱 확대되었습니다. 분자동역학 및 FEP 시뮬레이션을 위한 리간드 커스텀 파라미터 생성 도구와 추가 연구를 위한 리간드 구조 준비 도구도 개발 및 개선되었습니다.
또한, 신규 Flare profiles는 Flare GUI를 현 라이선스 레벨에 맞춰 구성해주어 플랫폼을 더욱 효율적이고 쉽게 이용할 수 있습니다.
MM/GBSA1는 리간드-단백질 복합체의 결합 자유 에너지를 계산하는 방법입니다. 이 방법은 정확도와 계산 효율 사이의 우수한 균형을 제공합니다. 이 방법은 분자 역학(Molecular Mechanics)을 이용하고 있어 분자 도킹 등에서 이용되는 empirical scoring function에 비해 이론적으로 더 엄밀하며 다양한 계에 대해 더욱 정확한 결과를 제공합니다(그림 1). 또한, 이 방법은 Flare FEP 등에서와 같은 상대 결합 자유 에너지 시뮬레이션에 비해 계산 비용이 적게 들기 때문에 Flare GUI를 통해서는 수백 개의 화합물을 커맨드 라인을 통해서는 수천 개의 화합물을 빠르게 스코어링할 수 있습니다.
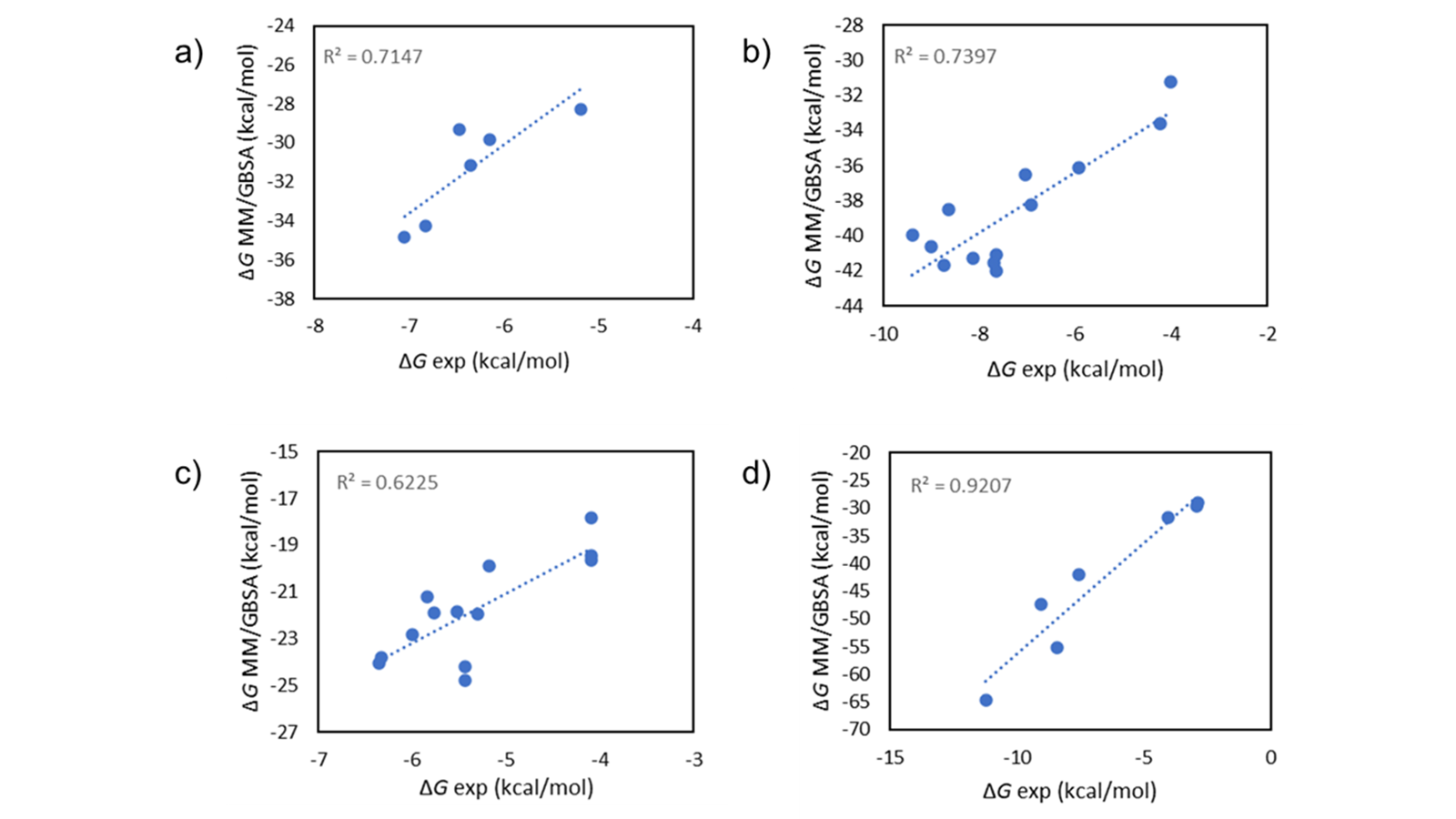 그림 1. Flare의 MM/GBSA single-point scoring을 이용한 FBDD 데이터 세트2의 실험 vs. 계산 ΔG 값
그림 1. Flare의 MM/GBSA single-point scoring을 이용한 FBDD 데이터 세트2의 실험 vs. 계산 ΔG 값
이러한 이유에서 MM/GBSA는 Flare docking 또는 SparkTM R-group replacement 실험 이후에 동종 리간드(congeneric ligands)에 대한 결합 친화도를 high-throughput으로 평가하는데 이용될 수 있으며 사용자는 이를 통해 설계된 분자들의 우선순위를 자신 있게 정할 수 있습니다.
Flare의 MM/GBSA 구현(그림 2)은 리간드 파라미터화에 OpenFF의 다양한 버전 및 AMBER GAFF/GAFF2 force field의 선택 옵션을 제공합니다. 다양한 implicit solvent model이 이용 가능하여 MM/GBSA를 관심있는 단백질-리간드 계에 대응하여 설정할 수 있습니다. 리간드 및 단백질 최적화 옵션은 단백질-리간드 계 내의 elastic strain 및 잠재적 electrostatic clashes를 줄여 보다 정확한 결합 자유 에너지 예측을 가능하게 합니다.
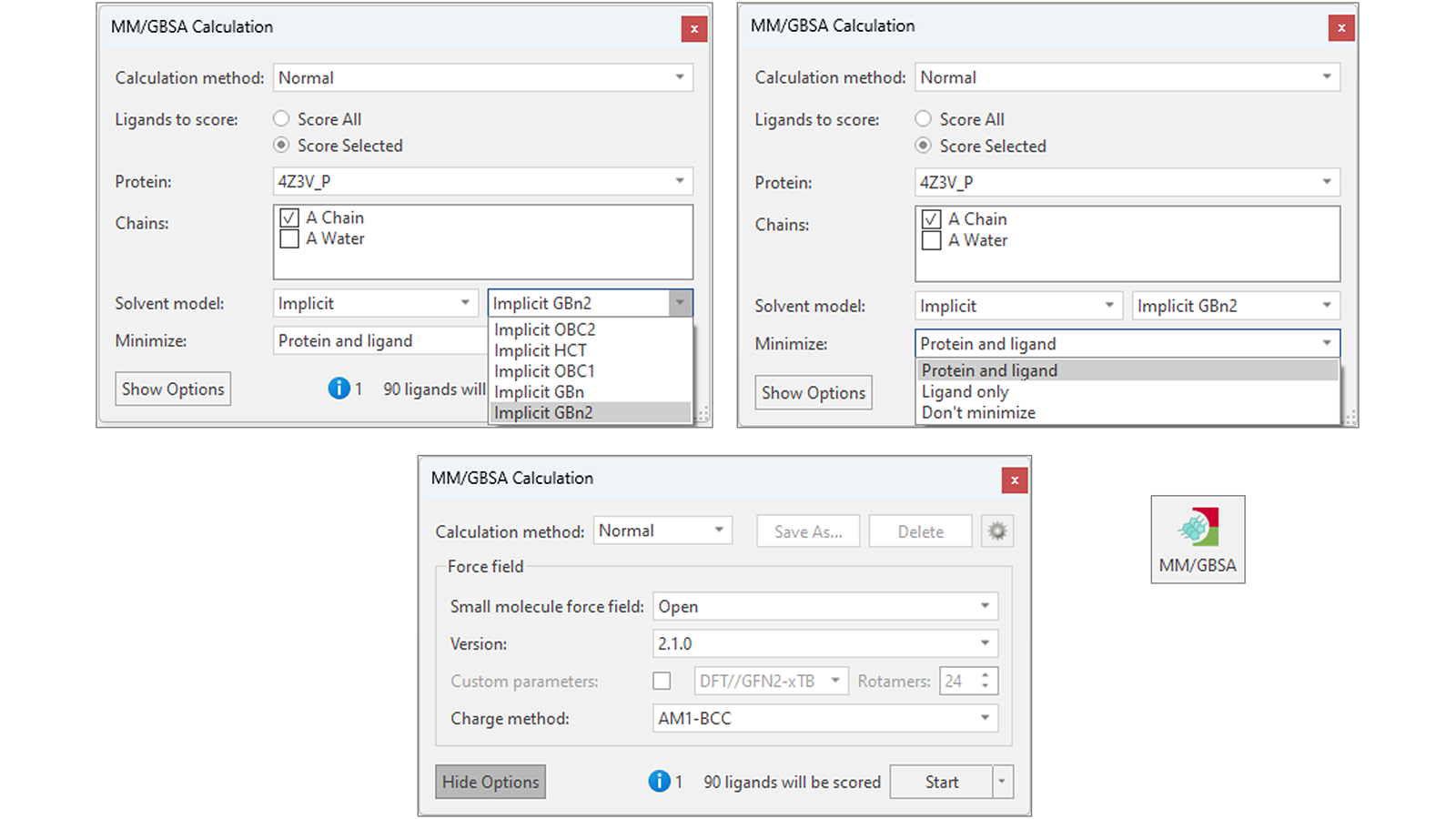
그림 2. Flare의 MM/GBSA 구현은 다양한 implicit solvent model(좌상단), 리간드 및 표적 단백질의 최적화를 위한 선택 옵션(우상단), 리간드 파라미터화를 위한 다양한 저분자 force field(하단)를 제공합니다.
이번 Flare 릴리즈의 Flare FEP는 순전하(net charge)가 다른 리간드를 포함한 프로젝트를 지원하여 실제 신약 개발 프로젝트에 Flare FEP의 적용 가능성을 크게 확대했습니다.
예측 성능은 이러한 네트워크에 대한 문헌의 데이터와 일치하며 그 예를 그림 3에 나타내었습니다.
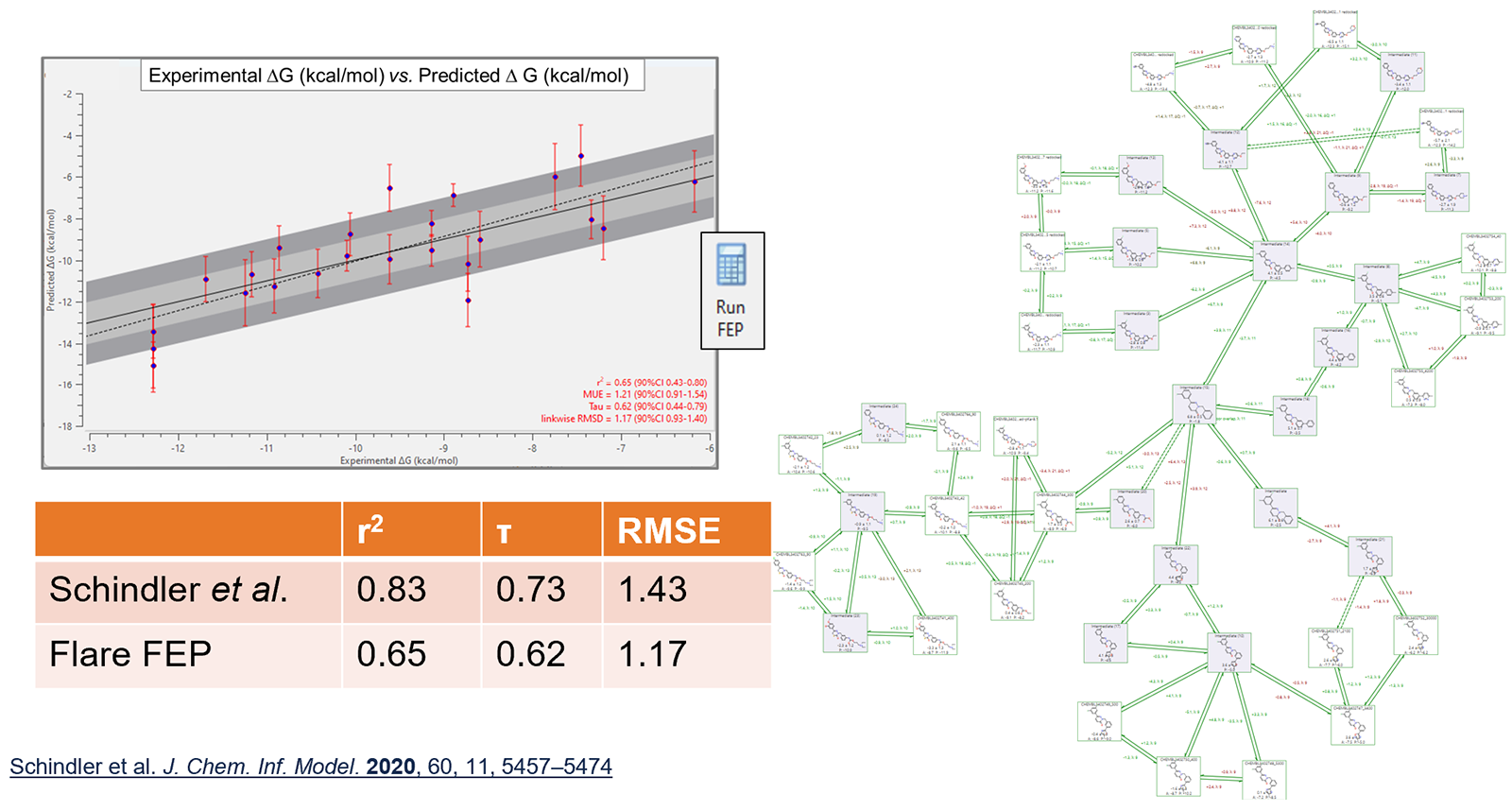
그림 3. c-Met 데이터 세트에 대한 Flare FEP의 예측 성능 및 Schindler 등 문헌3과의 비교
이번 릴리즈에는 Flare FEP 계산 결과를 이해하고 문제를 해결할 수 있는 고도의 시각적 도구가 추가되었습니다.
Torsion Plot(그림 4)은 특정 Flare FEP 변환에 관여하는 리간드의 각 회전 가능 결합에 의해 채택된 torsion 각도의 히스토그램을 자유 상태와 결합 상태 모두에서 변환의 각 프레임에 대해 계산된 것입니다. 이 Plot은 시작 분자와 마지막 분자 사이에서 발생하는 예상치 못한 conformation 변화를 시각화하는데 유용하며 이는 계가 샘플링 중이며 더 긴 시뮬레이션 기간 동안 다시 실행해야 할 수 있음을 나타내는 신호일 수 있습니다.
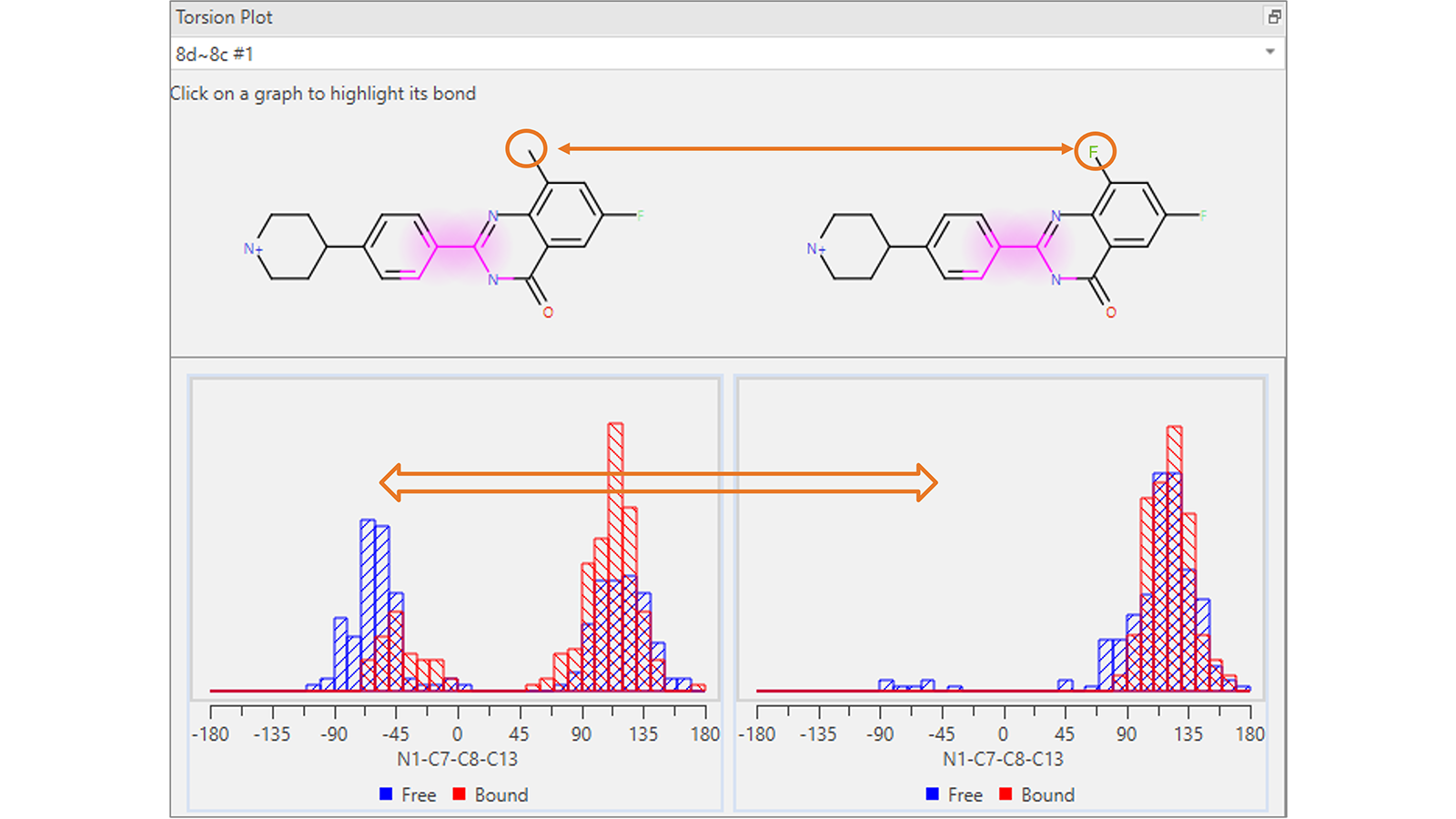
그림 4. Torsion Plot은 Flare FEP 변환에 관여된 분자의 공통 scaffold에서 강조 표시된 회전 가능 결합에 대한 torsion 각도 분포의 예기치 않은 차이를 보여줍니다.
Convergence Plot은 Flare FEP 프로젝트에서 각 변환의 시간 경과에 따른 FEP 시뮬레이션의 신뢰도 및 수렴을 평가하는데 유용합니다. 시뮬레이션 시간의 각 구간(순방향선)과 trajectory의 해당 마지막 구간(역방향선)에 ΔG 및 관련된 오차를 표시함으로써 그림 5에 나타낸 예와 같이 최적이 아닌 변환을 식별하는데 매우 효과적인 시각적 도구를 제공합니다. 최적으로 수렴된 변환에서는 순방향선과 역방향선이 Plot의 오른쪽보다 훨씬 앞서 서로 접근하지만 그림 5에 표시된 변환에서는 이러한 현상이 발생하지 않으므로 더 긴 시뮬레이션이 필요할 수 있습니다.
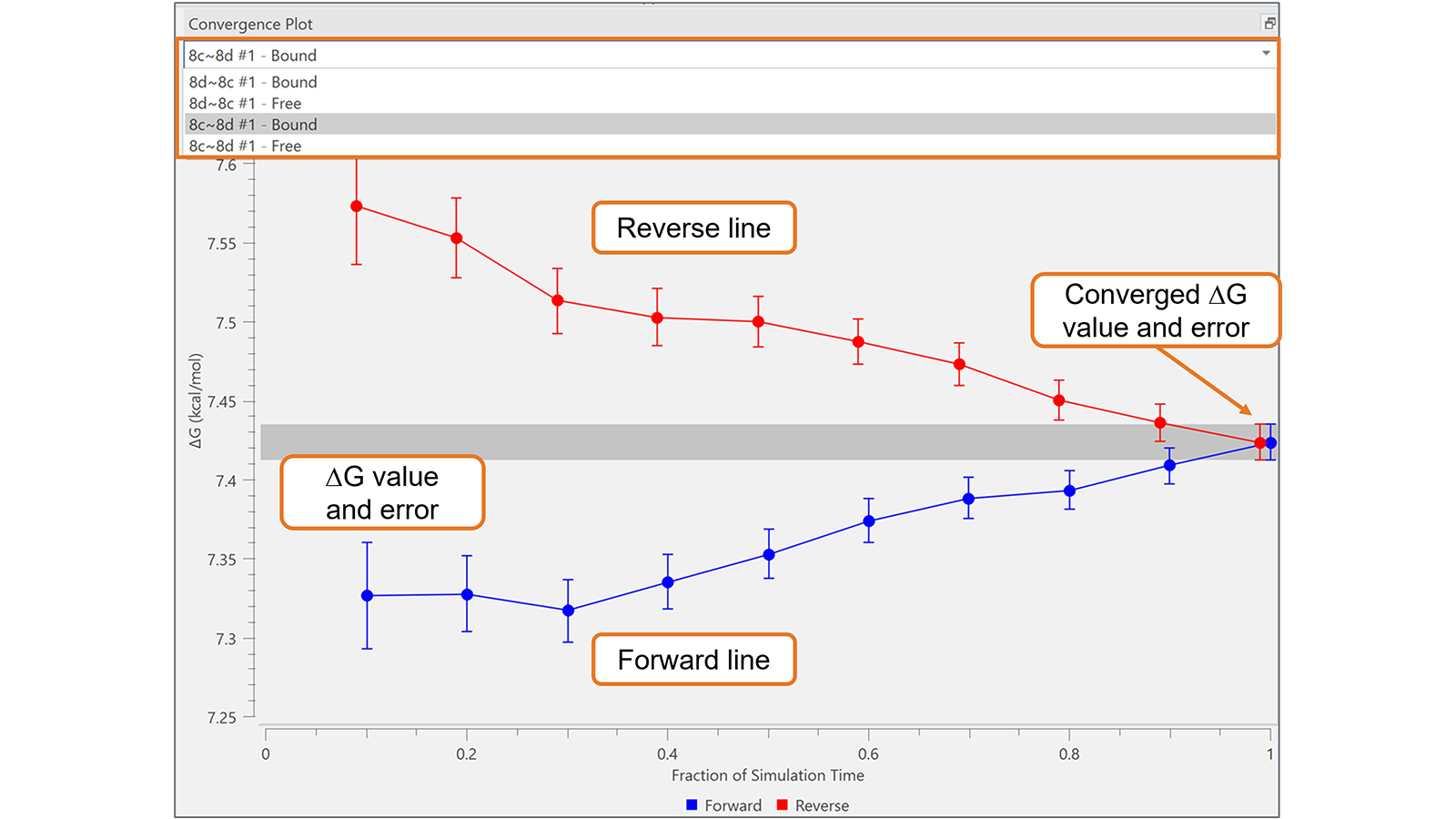
그림 5. Convergence Plot은 이 예에서와 같이 최적이 아닌 convergence로 변환을 모니터링하는데 유용합니다.
마지막으로 신규 Contacts 테이블(그림 6)은 변환의 각 lambda window에 변환 중인 리간드와 그 주변 분자(단백질, 물분자, 보조인자 등) 간에 형성되는 contact을 모니터링하는데 유용합니다. 예기치 않게 변하는 contact은 강조 표시되므로 주목할만한 contact 거동을 쉽게 식별할 수 있습니다.
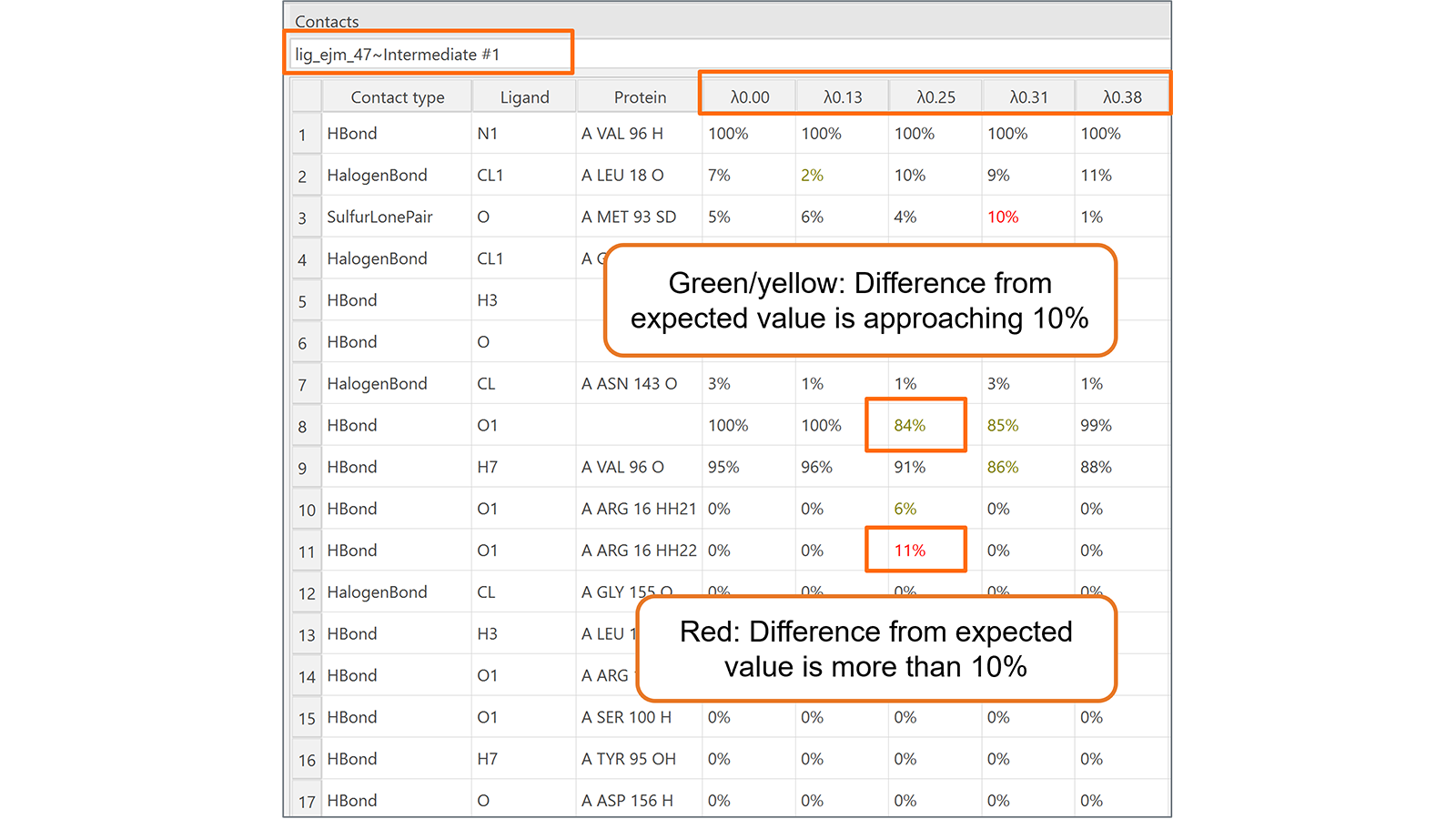
그림 6. Flare FEP의 신규 Contacts 테이블은 변환의 각 lambda window에 변환 중인 리간드와 그 주변 분자(단백질, 물분자, 보조인자 등) 간에 형성된 contact을 모니터링합니다.
Flare의 저분자에 대한 양자 역학 계산 구현은 더욱 확장되어 HOMO (Highest Occupied Molecular Orbital) 및 LUMO(Lowest Unoccupied Molecular Orbital)를 계산하고 이를 화면에 시각화할 수 있게 되었습니다. 이는 HOMO/LUMO gap 계산과 함께 저분자의 반응성 및 대사 거동을 이해하는데 매우 유용합니다.
그림 7의 상단 예에서 2,4-dichloro-5-methylpyrimidine의 piperidine에 대한 친핵성 치환은 2번 위치보다 4번 위치에서 선호적으로 발생하는 것이 문헌4을 통해 알려져 있습니다. 2,4-dichloro-5-methylpyrimidine의 LUMO 계산 및 시각화는 실험 거동을 설명합니다. C2와 C5는 관련된 LUMO lob가 없는 반면 C4와 C6는 가장 큰 LUMO lob가 존재합니다. 따라서, 친핵성 치환은 이들 위치 중 한 곳에서 선호됩니다.
그림 7 하단의 두번째 예5에서는 naphthalene의 HOMO를 조사하는 것으로 nitric acid의 친전자성 공격이 C2가 아닌 C1 위치에서 선호적으로 발생되는 현상에 대해 해당 위치가 전자가 더 풍부함을 나타내는 큰 HOMO lob와 관련되어 있음을 설명합니다.
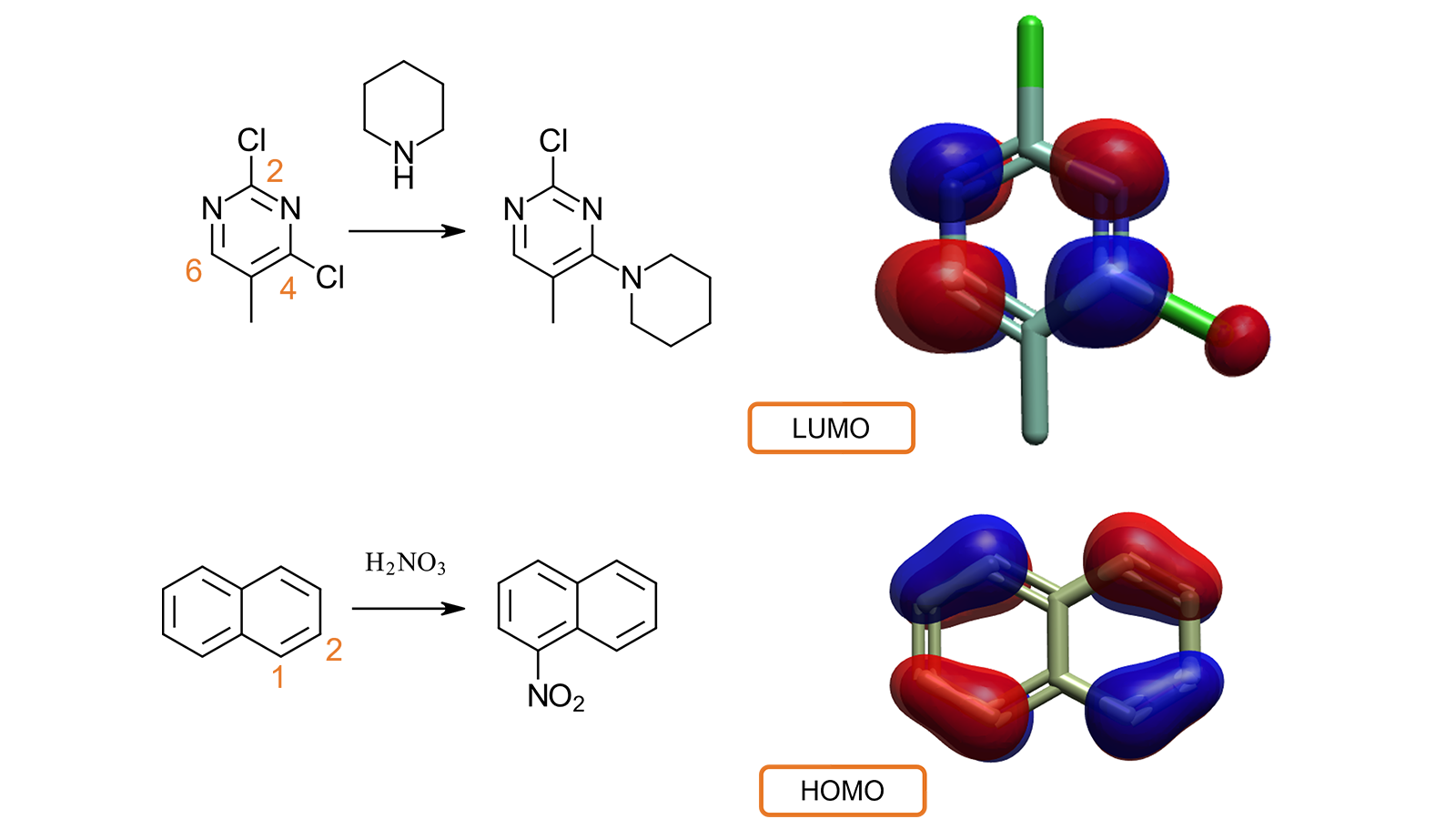
그림 7. LUMO 및 HOMO의 시각화는 저분자의 반응성 및 대사 거동을 이해하는데 도움이 됩니다.
Flare의 선-정의된 enumeration 반응은 대폭 확장되어 현재 150가지 이상의 반응을 제공합니다. 이들 반응은 합성 화학에서 널리 이용되는 것으로 bicyclic/monocyclic aromatic ring 및 non-aromatic ring, C-C 및 N|O|S-C 결합 형성, deprotection, oxidation 및 reduction 반응을 포함한 매우 다양한 변환을 다룹니다.
신규 ‘Search Reactions’ 기능도 원하는 반응을 쉽고 빠르게 찾을 수 있도록 추가되었습니다(그림 8).
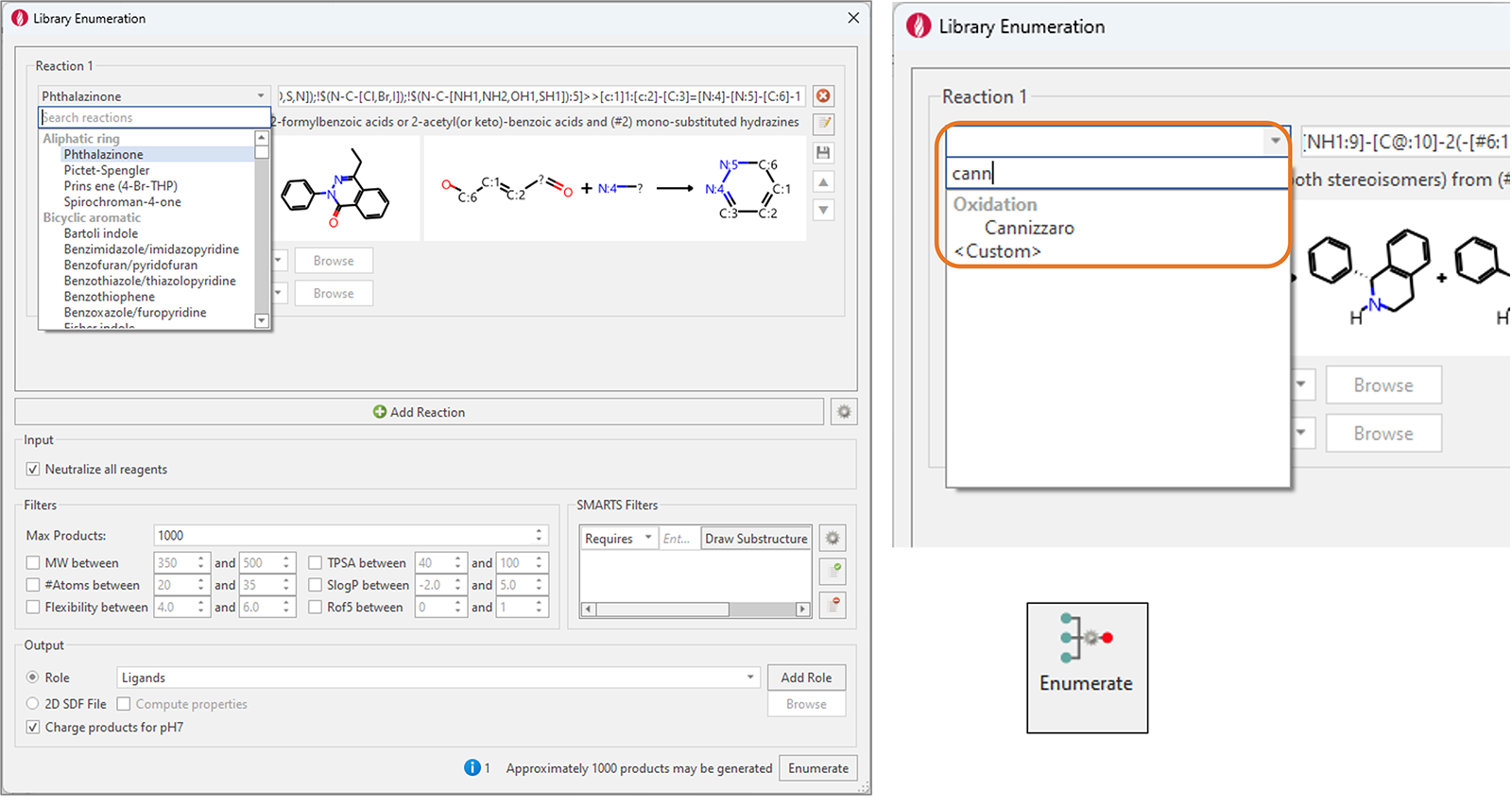
그림 8. 왼쪽: Flare V8의 Library Enumeration은 합성 화학에서 널리 이용되는 100가지 이상의 다양한 신규 반응을 제공합니다. 오른쪽: 신규 ‘Search Reaction’은 원하는 반응을 쉽게 찾을 수 있도록 도와줍니다.
Ligand preparation은 protonation 및 tautomeric state를 수정, salt를 제거, (필요한 경우) 정의되는 않은 stereocenter를 enumeration하는 등 추가 분자 모델링 연구를 위해 저분자를 준비하는 필수적이고 중요한 단계입니다.
Flare V8에서는 리간드에 대한 합리적인 3D 구조를 생성하는 신규 ‘Pop to 3D and minimize’ 옵션(그림 9)이 추가되었으며 이들을 Docking 및 Scoring 또는 리간드 기반 정렬과 같은 추가 계산에 이용할 수 있습니다.
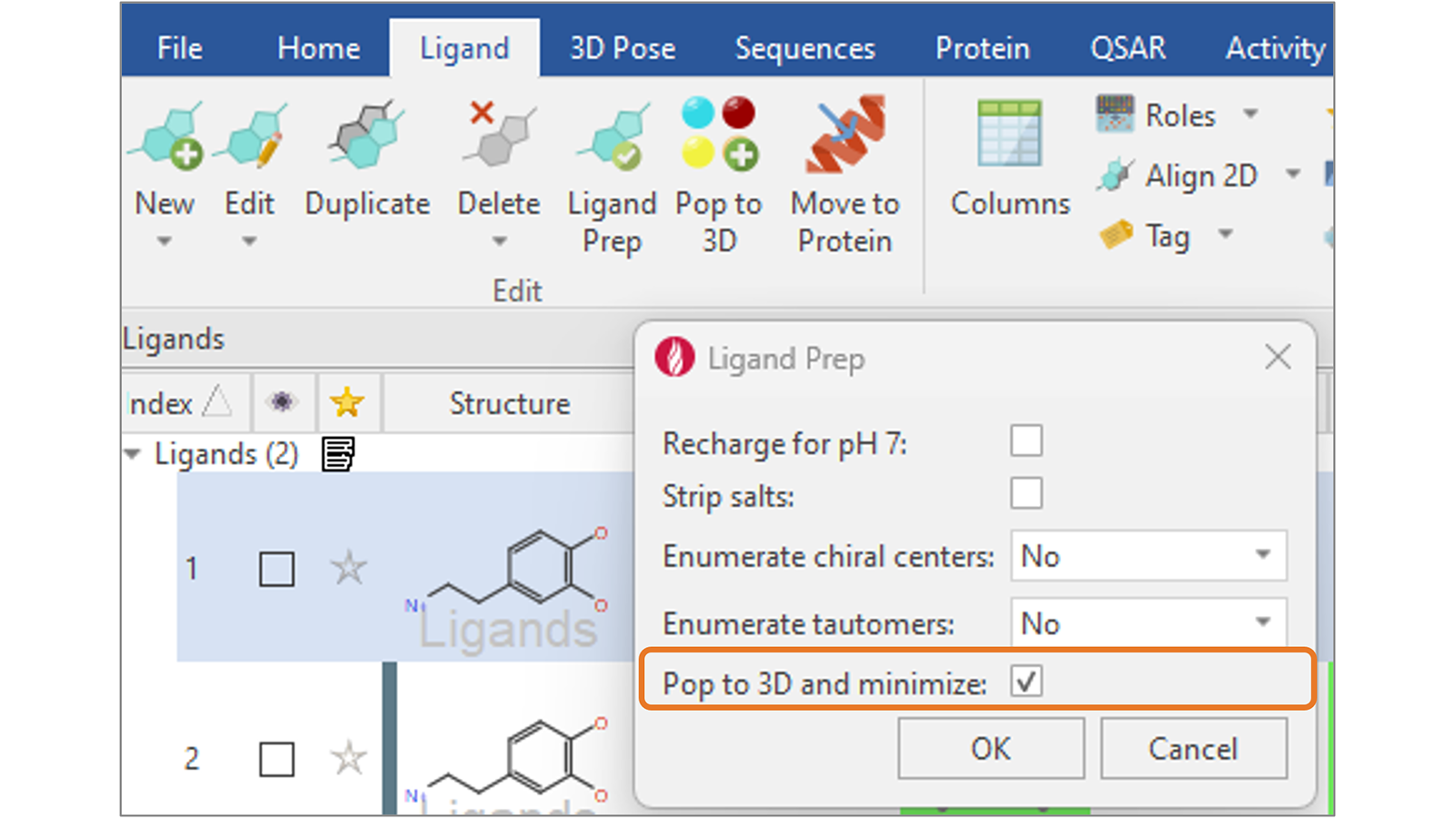
그림 9. 신규 ‘Pop to 3D and minimize 옵션은 리간드에 대한 합리적인 3D 구조를 생성합니다.
신규 ‘Radius of Gyration’ (RG) Plot(그림 10)은 분자동역학 연구의 결과를 분석하기 위해 Flare의 시각적 도구 모음에 추가되었습니다.
이 플롯은 단백질의 alpha carbon에 대한 RG 값을 시간의 함수로 나타내며 일반적으로 단백질 또는 펩타이드와 같은 생체 분자의 조밀도(compactness) 또는 크기를 분석하는데 이용할 수 있습니다. RG 값이 작을수록 더 조밀하거나 견고하게 접힌 구조를 나타내며 RG 값이 클수록 더 확장되거나 무질서한 conformation을 나타냅니다. 따라서, RG Plot은 생체 분자의 구조적인 변화, 유연성 및 동역학을 연구하는데 이용할 수 있으며 이는 생체 분자의 기능과 거동을 이해하는데 중요한 역할을 합니다.
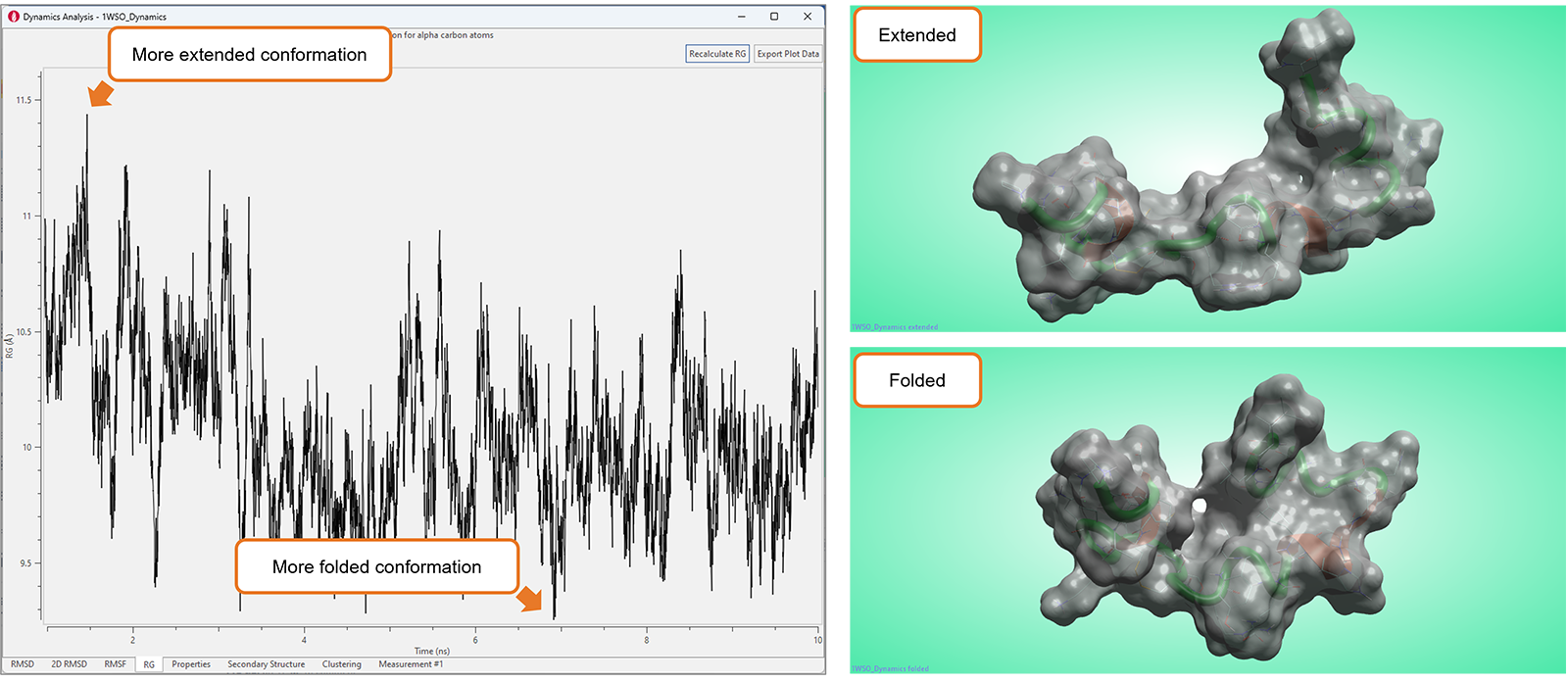
그림 10. RG Plot의 큰 요동(fluctuations)은 일반적으로 연구 중인 생체 분자의 conformation 변화를 매핑합니다.
또한 Flare V8의 Contacts 테이블은 시뮬레이션 중에 관찰되는 선호하는 리간드-단백질 상호작용에 대한 Contact 통계를 보고하며 물 분자와의 리간드 상호작용도 포함합니다.
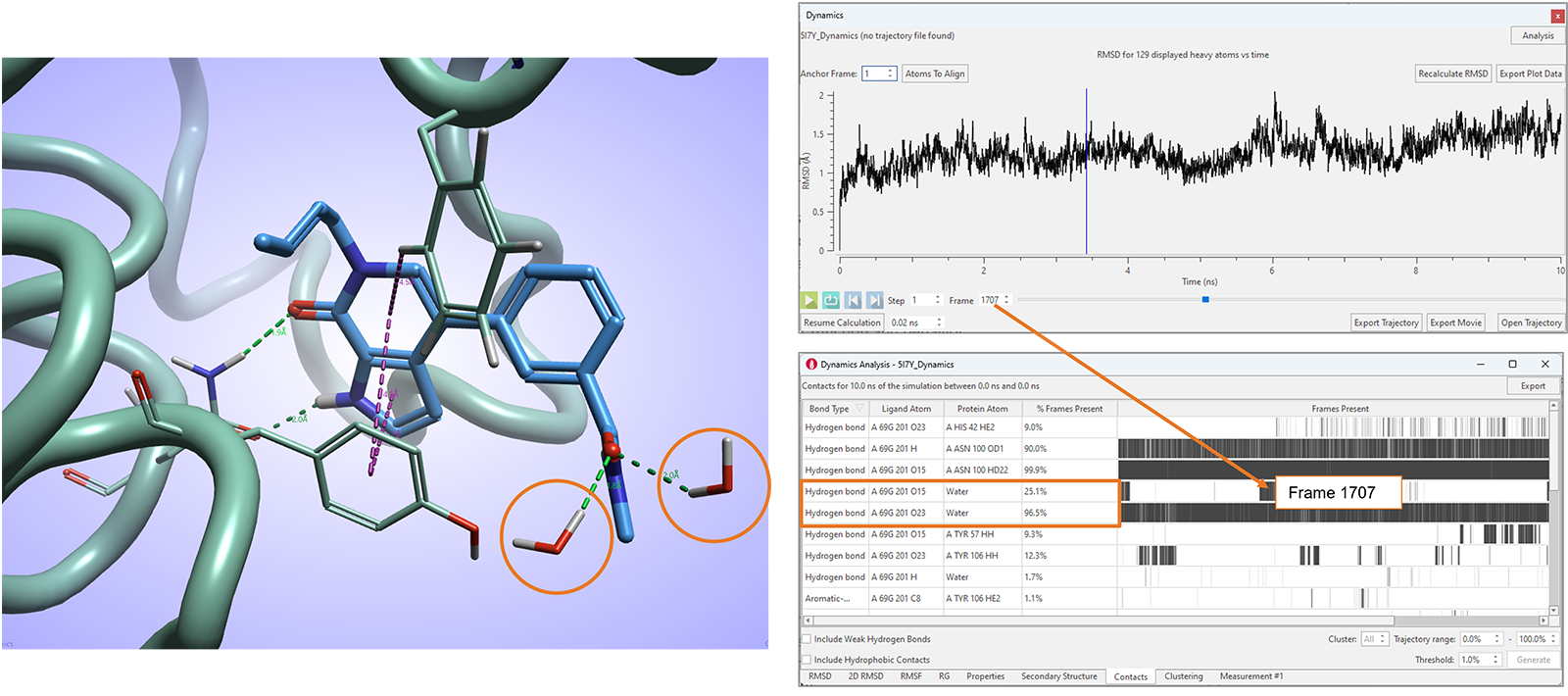
그림 11. Flare V8의 Contacts 분석 테이블은 선호하는 리간드-단백질 및 리간드-물분자 상호작용을 매핑합니다.
Flare의 분자동역학 계산에 대한 추가 개선 사항으로는 리간드가 없는 apo 활성 부위에 대해서도 Grand Canonical Nonequilibrium Candidate Monte Carlo (GCNCMC)6,7를 이용할 수 있는 기능과 분자동역학 trajectory의 불연속 프레임을 내보내는 기능을 포함합니다.
Flare V8의 리간드에 대한 정확한 OpenFF torsion 파라미터 생성은 “Custom Parameters” 기능으로 간단하게 수행할 수 있습니다. 하나 또는 여러 개의 리간드를 선택한 상태에서 이 버튼을 누르면 OpenFF 버전, 커스텀 파라미터 생성에 이용할 방법(DFT//GFN2-xTB, GFN2-xTB 또는 ANI-2X) 및 계산에 이용할 Rotamer의 개수를 선택할 수 있는 위젯이 표시됩니다(그림 12 상단 오른쪽).
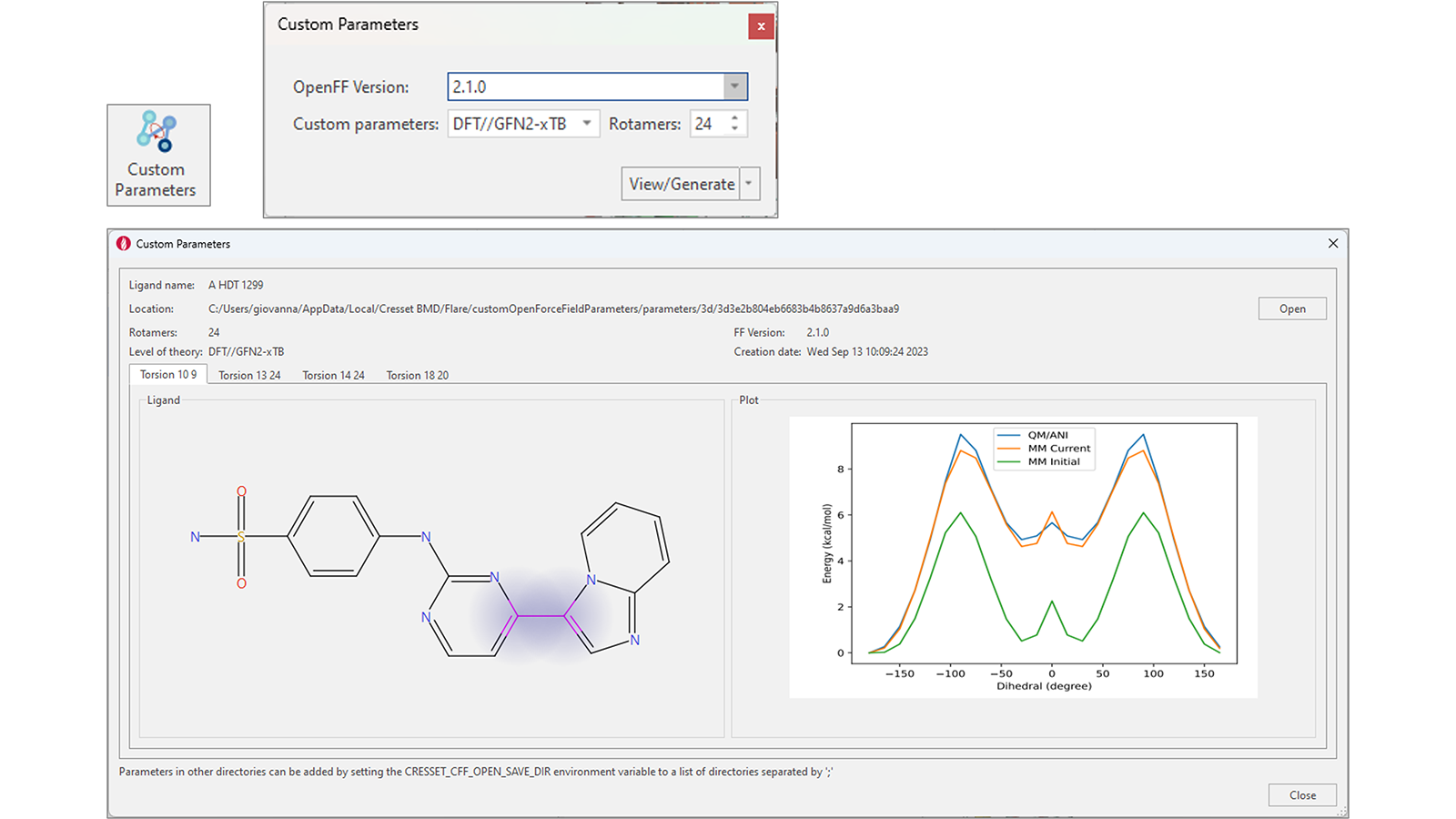
그림 12. 정확한 Torsion Force Field 파라미터 생성을 위한 향상된 워크플로
계산이 끝나면 커스텀 파라미터를 로컬 또는 공유 위치에 저장할 수 있으며 이후 분자동역학 및 Flare FEP 계산에 자동으로 이용됩니다.
커스텀 파라미터가 이미 생성된 경우, ‘View/Generate’ 버튼을 누르면 관심 리간드의 각 회전 가능 결합에 대한 계산 결과를 보여주는 창이 대신 열립니다(그림 12 하단).
이번 Flare 릴리즈에서는 단백질 구조 모델링을 용이하게 하는 신규 및 향상된 도구를 포함합니다.
‘Protein’ 탭의 ‘New Conformation’ 기능8은 선택 잔기의 곁사슬 conformation을 탐색하는 데 이용할 수 있습니다. 사용자는 버튼을 반복적으로 눌러 한 번에 하나의 곁사슬 conformation을 탐색하거나 옵션을 선택하여 나머지 단백질과 입체 충돌(steric clashes)을 피한 모든 conformation을 생성할 수 있습니다. 결과는 Alternate Conformations에 저장됩니다(그림 13).
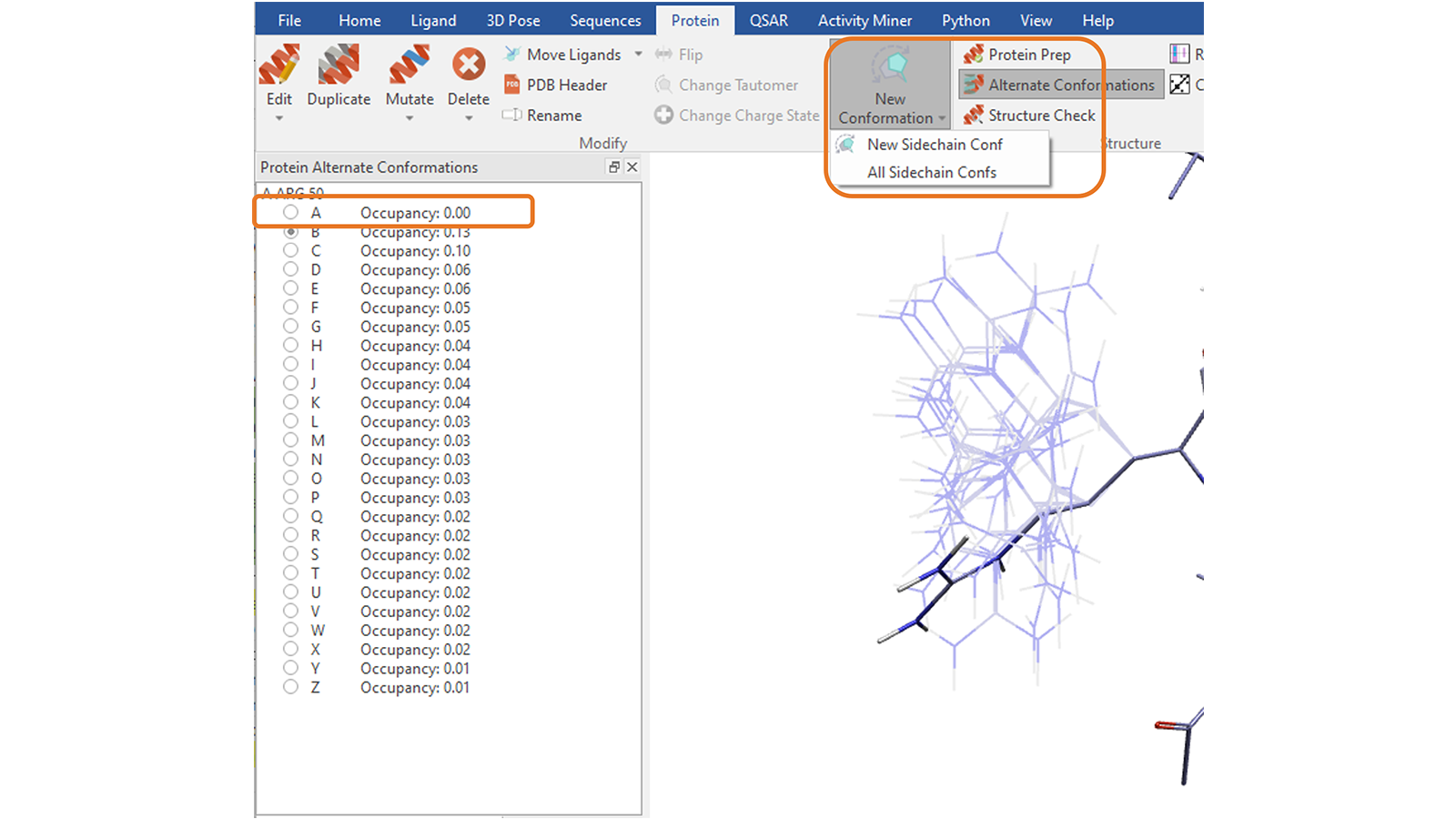
그림 13. 선택 잔기의 곁사슬에 대해 한 번에 하나의 conformation 또는 모든 conformation을 생성합니다. ‘All Sidechain Confs’ 옵션을 선택하면 결과를 Alternate Conformations에 저장합니다. 리스트의 첫 번째 엔트리는 original conformation에 해당합니다.
단백질 및 homology 모델링 관련 신규 기능이 ‘Sequences’ 탭에 추가(그림 14)되었으며 다음을 포함합니다.
- FASTA 및 ClustalW 파일 형태에서/로 시퀀스 가져오기 및 내보내기
- 선택 잔기 또는 전체 잔기를 다른 시퀀스-정렬된 단백질 사슬의 시퀀스와 일치하도록 변형(mutate)하기
- 선택 잔기 또는 전체 사슬의 번호를 다시 매기기, Gaps의 번호를 다시 매길지 여부를 선택하기, 중복 번호 매김을 피하기 위해 Insertion Codes를 지정하기
- 단백질 구조의 gaps를 수정하는 FREAD Loop 모델링 방법(Flare 이전 버전에서 이용 가능하였음)이 사용 편의성을 높이기 위해 이곳 탭으로 옮겨짐
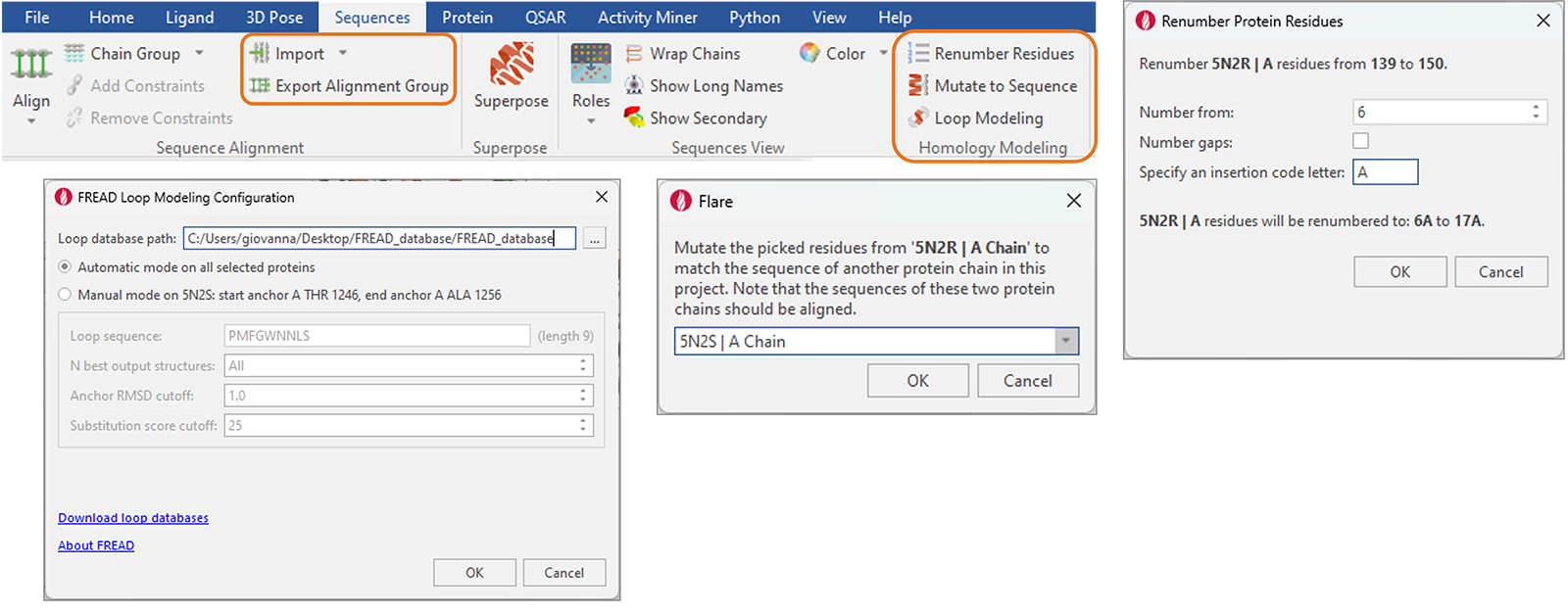
그림 14. 단백질 및 homology 모델링 관련 신규 및 향상된 기능은 Flare의 ‘Sequences’ 탭에서 이용 가능합니다.
마지막으로, Alignment 테이블에서는 시퀀스 정렬의 일치(consensus) 및 다양성을 시각적으로 매핑하는 신규 ‘Frequency Logo’가 표시됩니다(그림 15): 툴팁은 정렬된 시퀀스의 특정 위치에 존재하는 모든 잔기와 이들의 빈도를 보여줍니다.
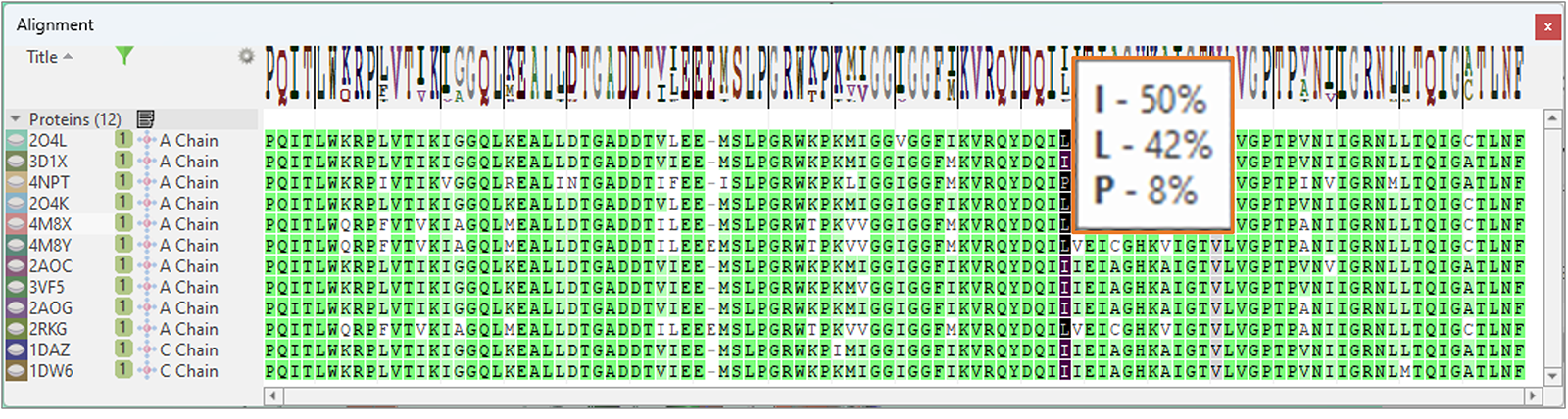
그림 15. 신규 ‘Frequency Logo’는 시퀀스 정렬의 일치(consensus) 및 다양성을 시각적으로 매핑합니다.
이번 릴리즈의 GIST water analysis 실험에서는 Grand Canonical Nonequilibrium Candidate Monte Carlo (GCNCMC)6,7를 이용한 향상된 water 샘플링의 이점을 제공하며 특히 단백질 활성 부위의 cryptic pockets에 대한 최적의 수화 상태(hydration)를 확보하는데 유용합니다.
그림 16에서 볼 수 있듯이 GCNCMC 단계는 실험에 이용되는 리간드를 중심으로 한 GCNCMC 구에 의해 정의된 영역에 대해 계산의 equilibration 단계에서 적용할 수 있습니다. 구의 사이즈와 GCNCMC 이동 빈도는 고급 옵션에서 미세 조정할 수 있습니다.
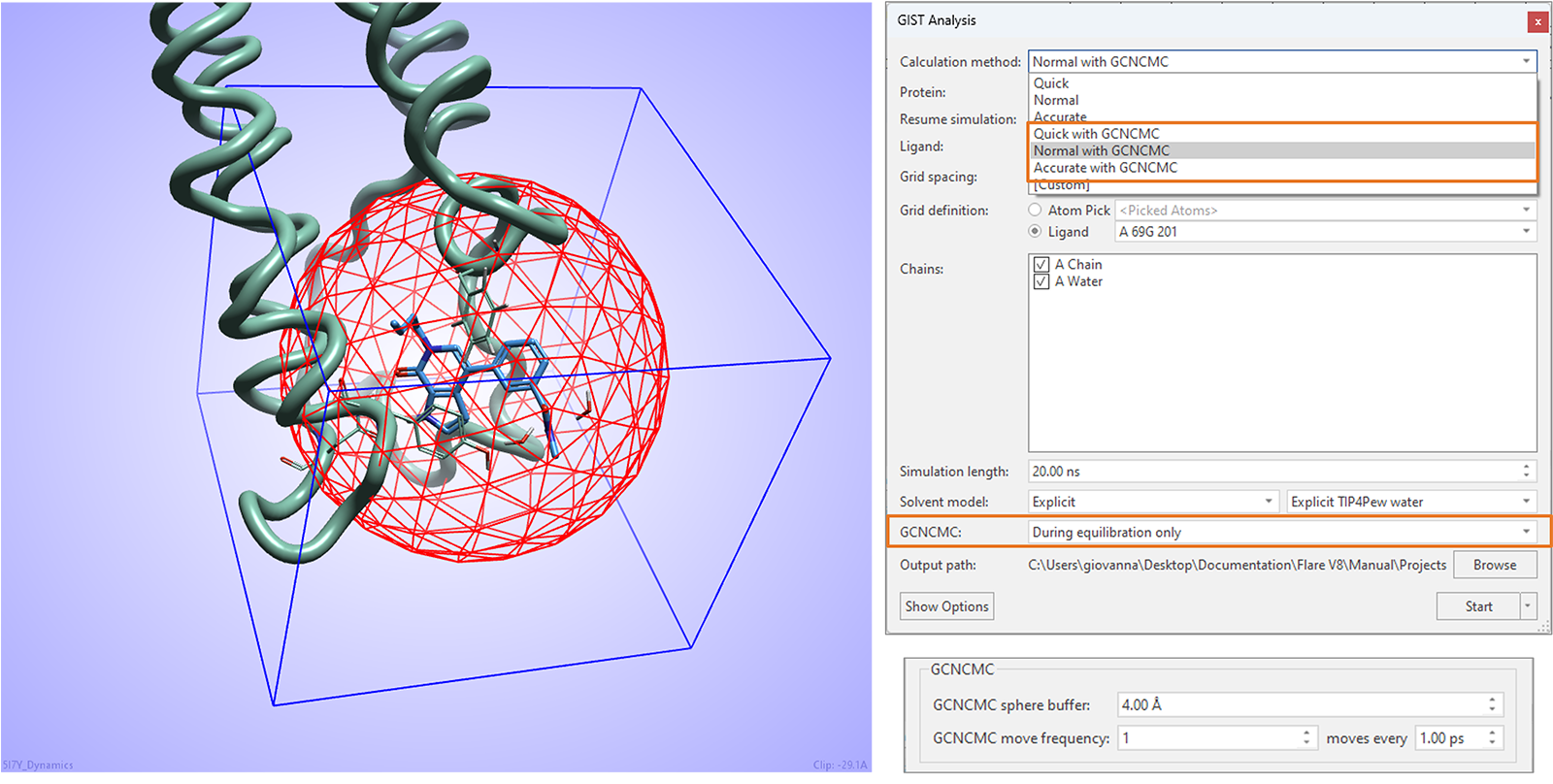
그림 16. Flare V8의 GIST water analysis는 equilibration 단계에서 GCNCMC의 이점을 활용할 수 있습니다.
Flare에서 이 신규 기능은 예를 들어 기본 Profiles(그림 17)에서 현 Flare 라이선스 레벨과 일치하는 것을 선택함으로써 Flare 리본 탭 메뉴를 커스텀 하는데 이용할 수 있습니다. 이러한 방식으로 현 라이선스 레벨에서 이용불가한 탭과 기능(버튼)이 숨겨지게 되며, 새 프로젝트를 열거나 Flare를 재시작할 때 선택한 profile이 유지됩니다. 언제라도 놓친 흥미로운 기능이 무엇인지 알고 싶다면 ‘Full’ profile을 선택하여 모든 Flare 탭과 버튼을 표시하게 합니다.
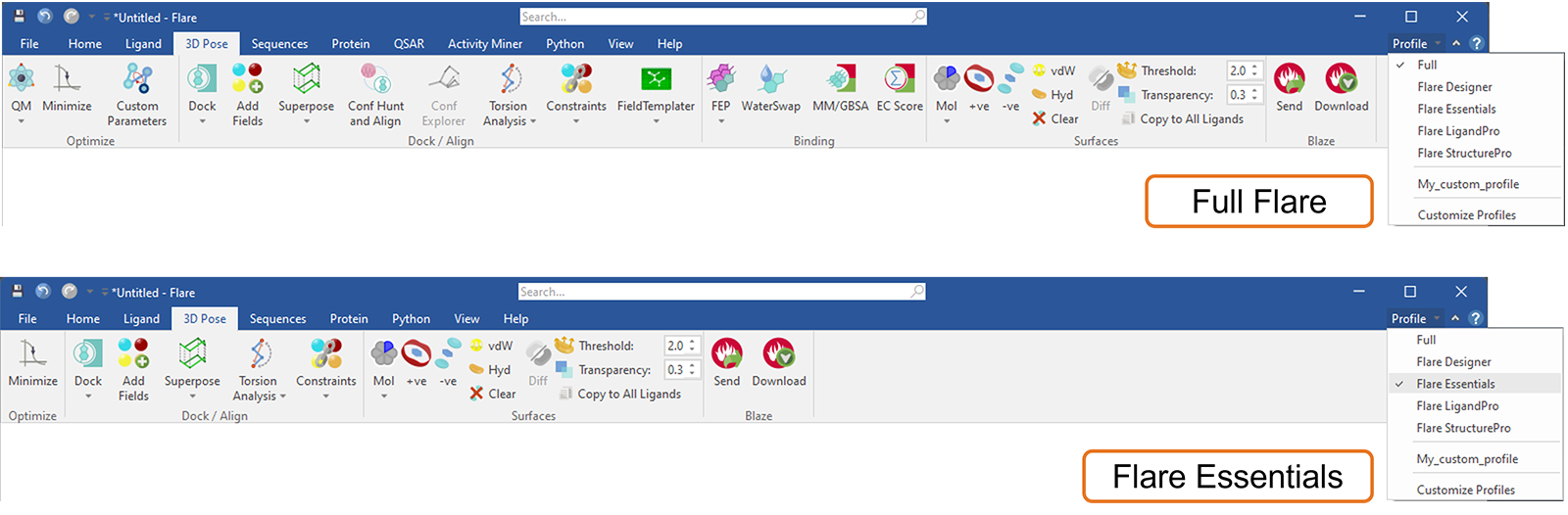
그림 17. Flare Essentials profile을 선택하면 해당 라이선스 레벨에서 이용가능한 탭과 버튼만 표시되도록 Flare 메뉴가 구성됩니다.
또한 자주 이용하는 탭과 버튼만 표시하고 이들의 appearance를 변경한 Custom profiles도 생성할 수 있습니다(그림 18).
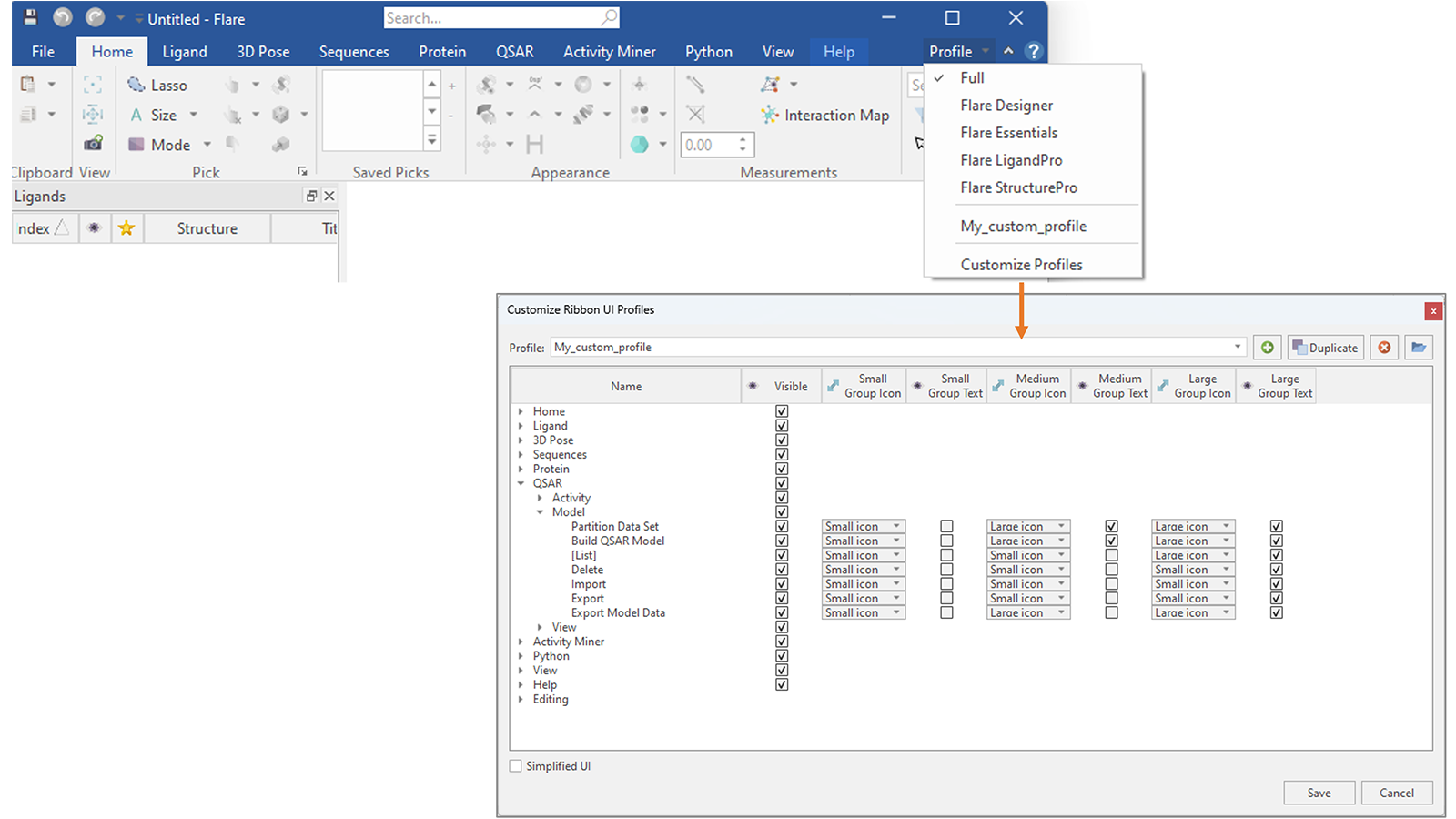
그림 18. Flare V8에서는 Flare 메뉴에 표시할 탭과 버튼을 결정하고 이들의 appearance를 변경하여 자신만의 Custom profiles를 생성할 수 있습니다.
Flare V8의 신규 기능 및 개선 사항에는 다음이 포함됩니다.
- Covalent inhibitors docking의 추가 warhead 정의
- Docking 및 Scoring의 신규 고급 옵션: substructure(예를 들어, co-crystallized ligand pose)를 지정하여 docking 실험 중에 입력 Conformation을 구속
- Ligands 테이블의 substructure filtering에 신규 ‘Is/Is Not’ 로직이 추가되어 검색 중인 구조와 정확히 일치 가능
- 커스텀 단백질 리본의 두께 및 너비 설정
- 펩타이드 및 단백질을 beta sheet 또는 alpha helix로 growing
- 하나 이상의 열을 선택하여 scatter plot에 주석을 달 때 레이블로 사용
- 가져온 RDKit descriptors를 재계산하는 Column & Activity Editor 내 신규 ‘Update RDKit Descriptors’ 기능
- Ligands 테이블의 열 값에 따라 리간드를 그룹화하는 신규 ‘Group By’ 기능
- Flare에서 Spark V10.7 프로젝트 파일 열기
- Pyflare 사용자를 위한 신규 및 향상된 스크립트
- 신규 및 향상된 Flare extensions
지금 바로 평가 사용을 요청하여 Flare V8의 신규 및 향상된 기능을 이용해 보세요. Flare는 다양한 라이선스 옵션이 이용 가능하여 계산화학자, 의약화학자 및 학술연구자에게 유연한 솔루션을 제공합니다. 평가 과정에서 생성된 결과는 문헌 등에 자유롭게 게제하고 추가 연구에 이용할 수 있습니다.