Improving PROTAC properties via single-point changes to linkers
We explore how computational methods can be applied to proteolysis targeting chimera (PROTAC) design, to effectively tackle some of the ...
News
리간드 기반 및 구조 기반 모델링을 위한 Cresset의 워크벤치인 Flare의 새로운 버전 7 릴리즈에는 분자동역학 및 Free Energy Perturbation(FEP) 계산을 위한 Grand Canonical Nonequilibrium Candidate Monte Carlo(GCNCMC1)를 이용한 향상된 수화(hydration) 패턴 샘플링, QM semi-empirical 계산, 새로운 ensemble covalent docking 방법, 예측 QSAR 모델의 구축을 위한 Consensus 모델 등 더욱 새롭고 향상된 과학적 기능 및 방법이 포함됩니다.
또한, 이번 릴리즈에서는 Flare FEP 실험의 문제 해결, 분자동역학 trajectories의 분석, 분자동역학 및 FEP 시뮬레이션을 위한 리간드 커스텀 파라미터의 생성, 추가 연구를 위한 리간드 구조의 준비 등에 이용할 수 있는 도구의 선택 폭이 확장되었습니다.
병렬 처리 및 새로운 계산 창을 통해 동일 Flare 프로젝트 내에서 다수의 작업을 개시, 실행 및 모니터링할 수 있게 되어 플랫폼이 더욱더 효율적이고 협업 친화적이며 사용하기에 간편해졌습니다.
Flare V7에서는 분자동역학 및 FEP 실험에 GCNCMC를 적용하여 물분자를 더 효율적으로 샘플링할 수 있게 되었습니다. 이 방법은 종래의 Grand Canonical Monte Carlo2(GCMC)와 Nonequilibrium Candidate Monte Carlo(NCMC)를 결합한 것으로 물분자의 삽입 및 제거를 점진적 비평형 방식으로 수행하여 단백질 활성 부위에 대한 수화 패턴의 샘플링을 향상시킵니다.
Flare FEP 계산의 평형화(equilibration) 단계에 GCNCMC를 이용하면 특히, 활성 부위가 일반적으로 수화되기 어려운 폐색 포켓(occluded pockets)을 특징으로 하는 단백질-리간드 복합체에서 방법의 예측 정확도를 크게 높일 수 있습니다(그림 1).
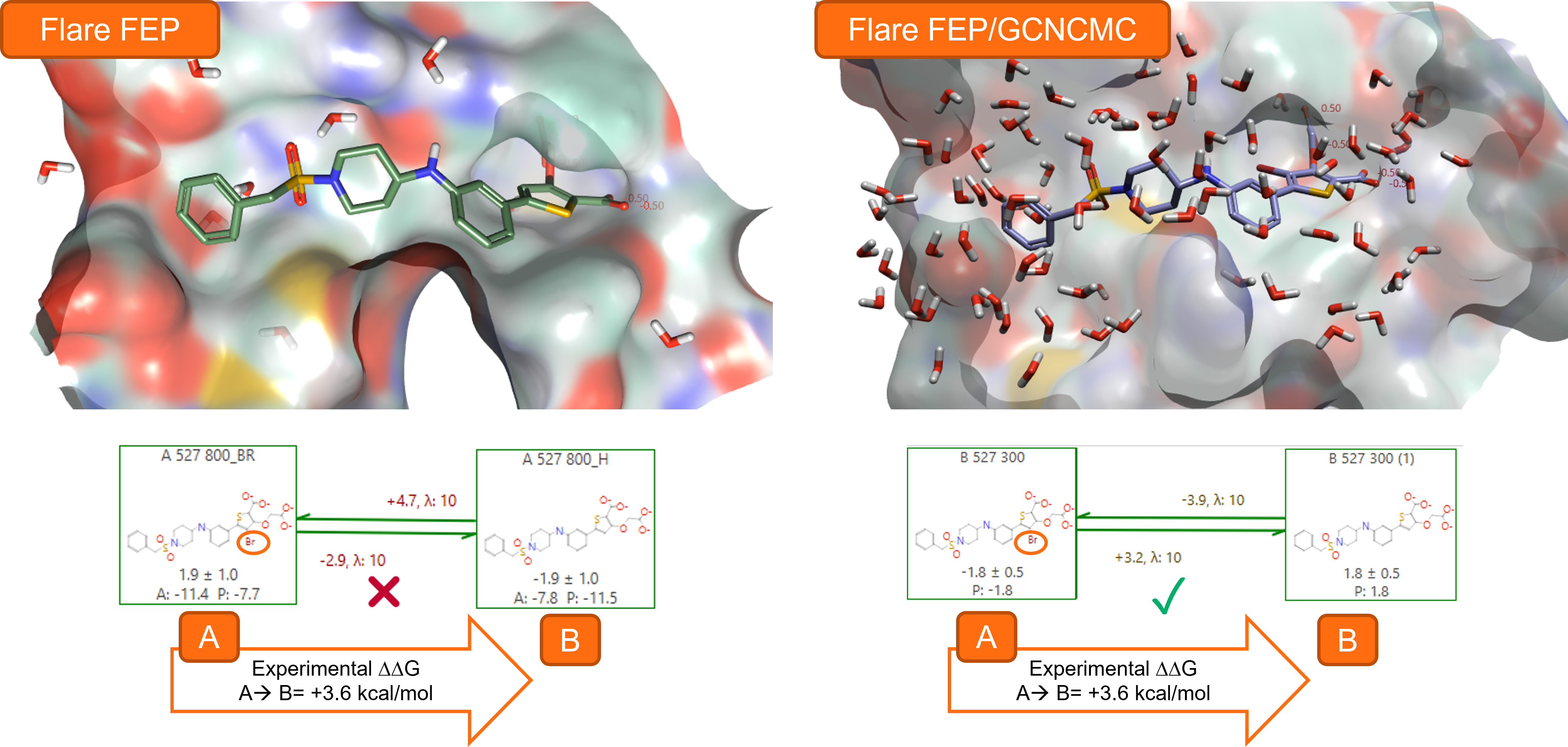
그림 1. Flare FEP 계산의 평형화 단계에 GCNCMC를 이용할 때 리간드 A(PDB: 2QBP, PTP1B Ki = 4nM3)에서 리간드 B(PTP1B Ki = 2000nM3)로 변환에 대한 FEP 예측 정확도가 크게 향상됩니다.
하위 그래프 분석은 Activity Plot의 새로운 기능이며 FEP 벤치마크 실험의 결과 분석을 위한 도구의 선택 폭을 더욱 확장합니다. 이 기능은 Flare FEP 프로젝트를 시작할 때 일상적으로 실행되어 시스템을 올바르게 설정하였는지 확인해 주거나 Activity Plot 내 각 리간드에 대한 실험 ΔG vs. 예측 ΔG를 비교하여 연구 중인 단백질 타겟/리간드 계열에 대한 방법의 예측력을 확인해 줍니다(그림 2).
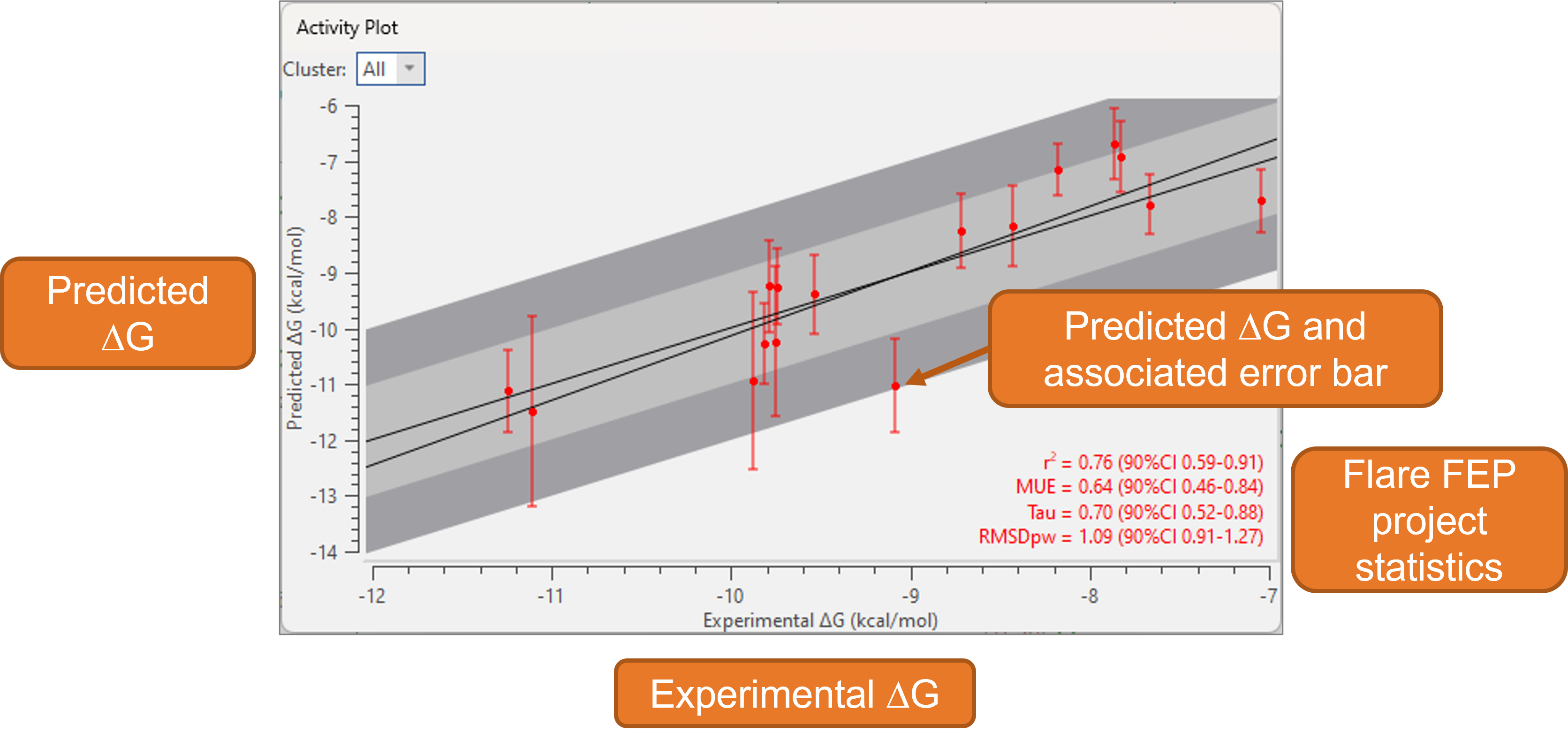
그림 2. Flare FEP 프로젝트의 Activity Plot.
실제 신약 개발 데이터로 수행된 Flare FEP 프로젝트의 perturbation networks에서는 입도(granularity, 매우 동종적이고 많은 상호 연결된 화합물들의 소그룹)를 관찰하는 것이 일반적입니다. 이들 그룹 내 분자에 대한 상대적인 ΔΔG 예측은 일반적으로 정확하며 신뢰할 수 있습니다. 그러나, 나머지 네트워크와의 연결 링크에 문제가 있거나 네트워크에 예측 정확도가 낮은 화합물도 포함하는 경우에 전체 데이터세트의 통계가 영향을 받아 잘 정의된(well-behaved) 그룹 내 화합물에 대해 과대평가된 예측 오류가 발생하며 대신 상대적인 ΔΔG 예측은 전적으로 신뢰할 수 있습니다.
이 경우에 하위 그래프 분석은 FEP 프로젝트 내 예측이 더 신뢰할 수 있는 내부 오차 통계가 낮은 화합물 클러스터를 확인하거나 큰 통계 오차를 유발하는 문제가 있는 변환을 중점적으로 확인하는데 유용할 수 있습니다. 예를 들어, 그림 3의 프로젝트에서는 두 개의 화합물 클러스터가 확인됩니다. 클러스터 1(보라색)의 화합물에 대한 예측은 Activity Plot의 큰 오차 막대로 알 수 있듯이 덜 정확합니다. 이는 많은 링크가 어느 정도의 hysteresis에 영향을 받기 때문입니다. 대신에, 클러스터 2(녹색)의 분자에 대한 예측은 더 정확합니다(오차 막대가 더 작음). 결과적으로 클러스터 2의 분자에 대한 상대적인 ΔΔG 예측은 매우 신뢰할 수 있지만 클러스터 1 또는 클러스터 1과2 사이의 분자에 대한 예측은 연결된 링크가 또한 hysteresis(주황색 타원)를 나타내고 있기 때문에 그다지 신뢰할 수 없습니다.
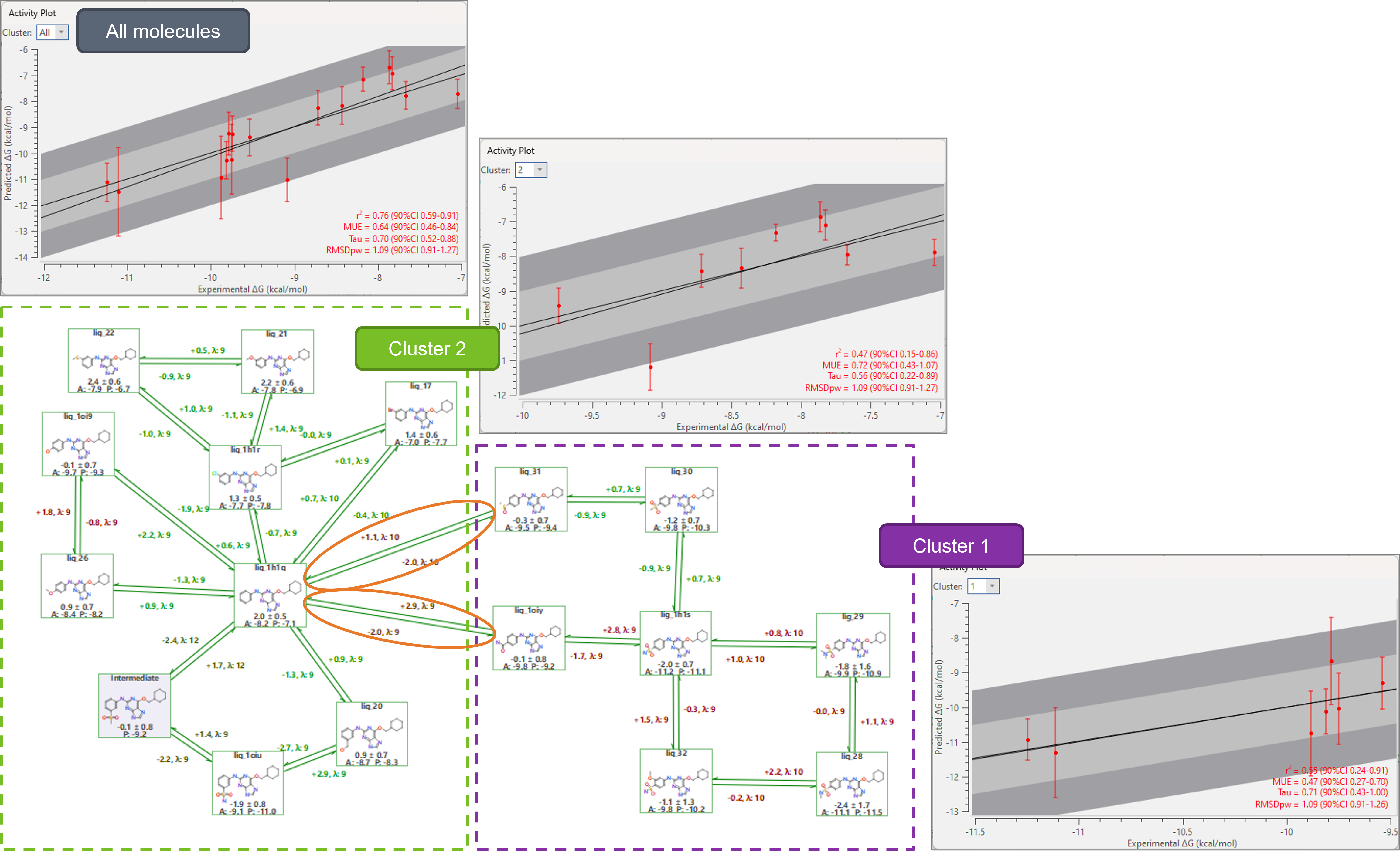
그림 3. Flare V7의 하위 그래프 분석은 FEP 프로젝트 내 예측이 더 신뢰할 수 있는 내부 오차 통계가 낮은 화합물 클러스터와 큰 통계 오차를 유발하는 문제가 있는 변환을 확인하는데 유용합니다.
Flare V7에서는 semi-empirical tight-binding 방법인 GFN2-xTB4,5을 새롭게 도입하여 양자 역학(QM) 계산을 수행하기 위한 옵션의 선택 폭을 더욱 확장했습니다. 이 방법은 사이즈가 큰 리간드의 계산에 권장되며, 적당한 정확도로 geometry optimization과 single-point energy 계산을 모두 수행할 수 있습니다. 또한, 빠른 semi-empirical minimization을 실행한 다음, DFT 수준에서 single-point energy를 계산하여 QM 워크플로의 계산 효율을 최적화할 수 있습니다(그림 4).
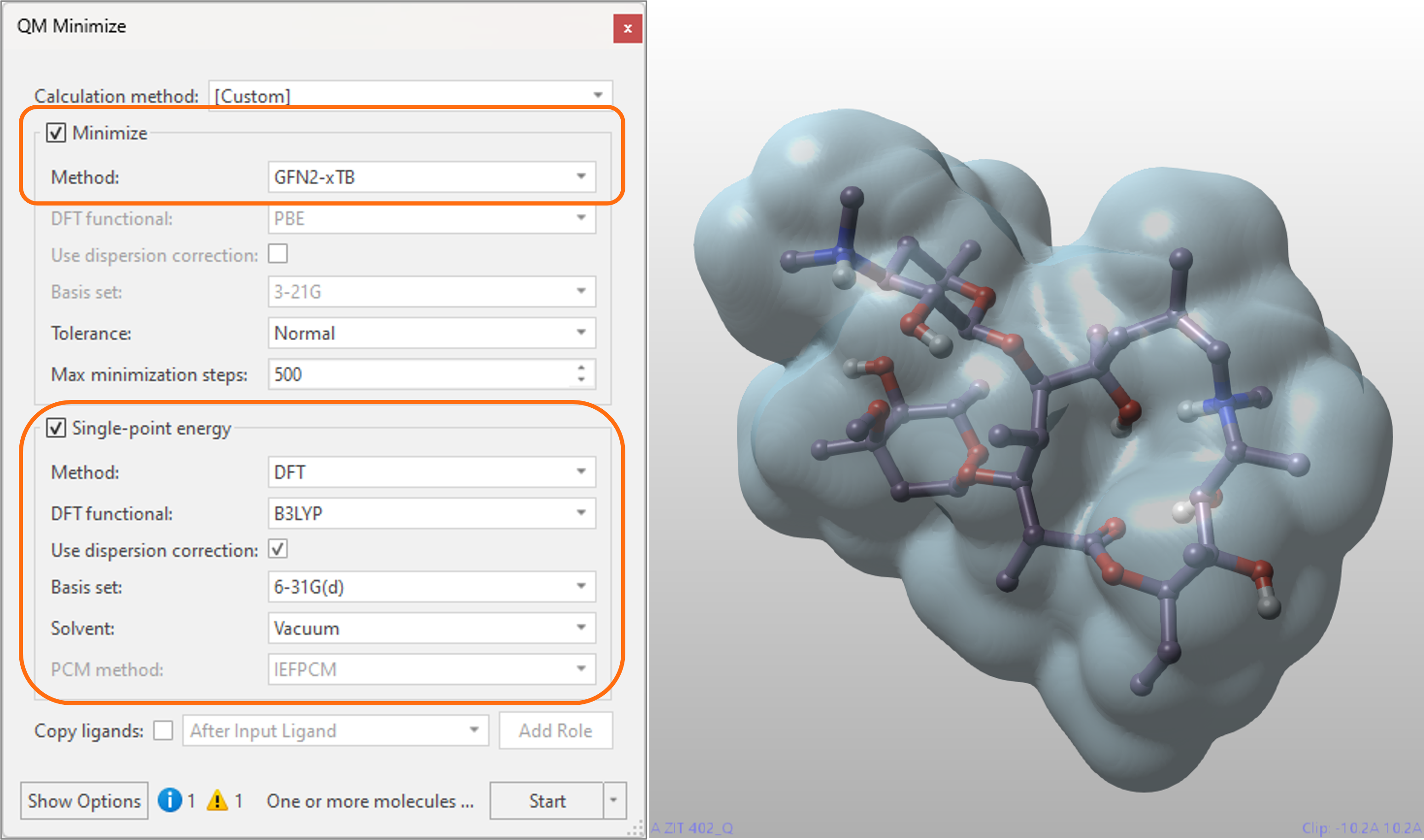
그림 4. Macrolide Azithromycin 분자의 semi-empirical 수준에서의 geometry optimization 계산과 기체상 또는 용매상에서의 DFT 수준 single-point energy의 후속 계산의 설정. 계산은 노트북에서 단 몇 분 만에 완료되며 큰 리간드에 대한 계산 효율이 높은 워크플로를 지원합니다. 시안색 = QM 전자 밀도
pyflare 사용자는 새로운‘qm.py’ 스크립트를 이용하여 명령줄에서 QM 계산을 효율적으로 수행할 수 있게 되었습니다.
분자동역학 실험의 경우, 평형화 단계 또는 시뮬레이션 전체에 GCNCMC 옵션을 활성화하여 시뮬레이션에 포함된 리간드를 둘러싼 단백질 활성 부위 영역의 수화 패턴 샘플링을 향상시킬 수 있습니다(그림 5). GCNCMC 구의 크기와 이동 빈도는 고급 옵션을 통해 미세 조정할 수 있습니다.
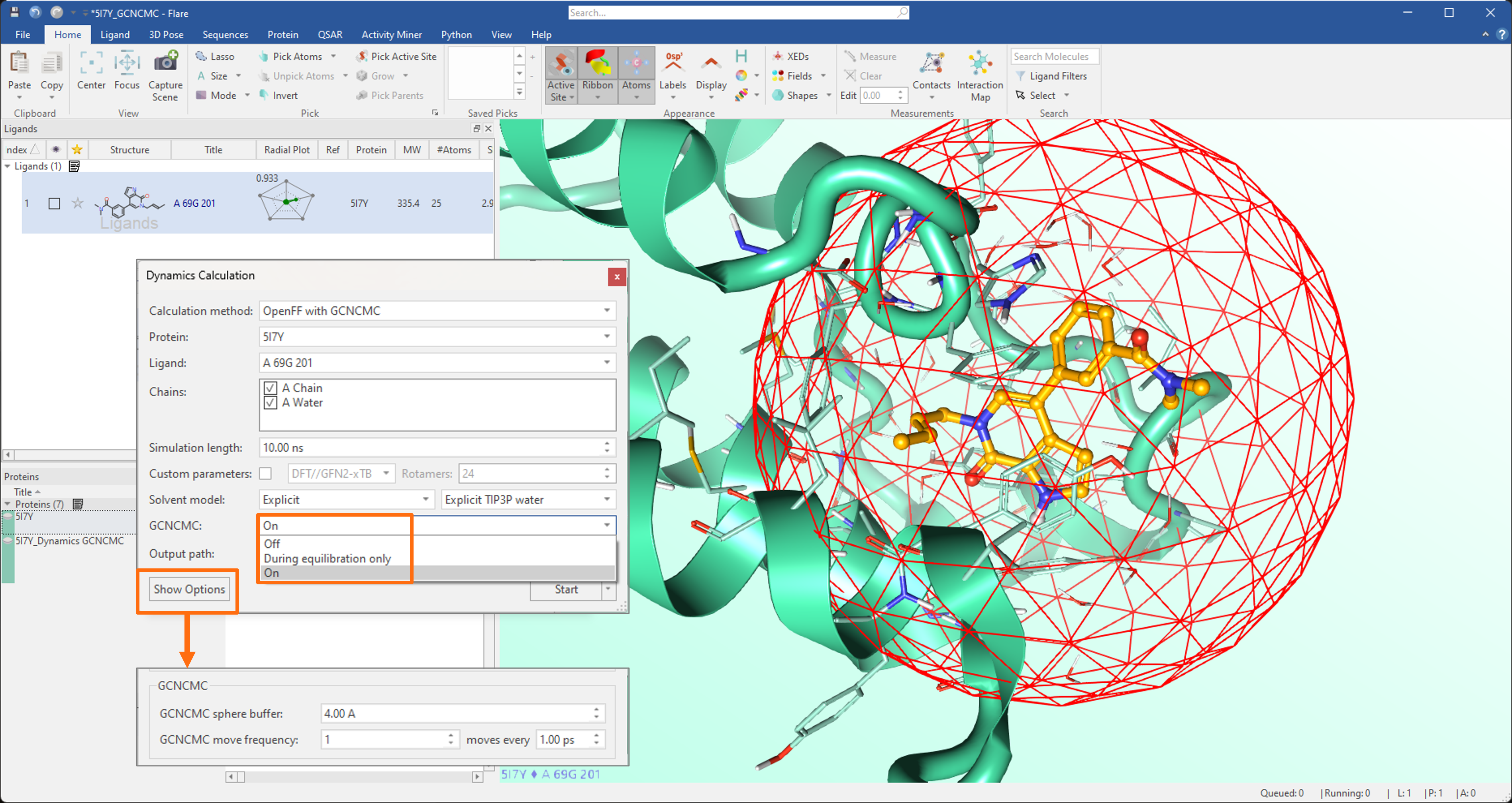
그림 5. 분자동역학 시뮬레이션에서 GCNCMC 옵션은 평형화 단계에서만 또는 시뮬레이션 전체에 활성화할 수 있습니다.
새로운 'Export Trajectory' 버튼(그림 6 – 왼쪽)을 이용하여 전체 분자동역학 trajectory를 내보내거나 원하는 프레임 범위만 내보낼 수 있어 관심 있는 범위의 프레임만 포함하도록 trajectory를 'trimming'하는 효율적인 워크플로를 제공합니다. 그런 다음 'Open Trajectory' 버튼을 눌러 트리밍된 trajectory를 Flare로 다시 가져와서 내보낸 단백질에 연결할 수 있습니다(그림 6 – 오른쪽).
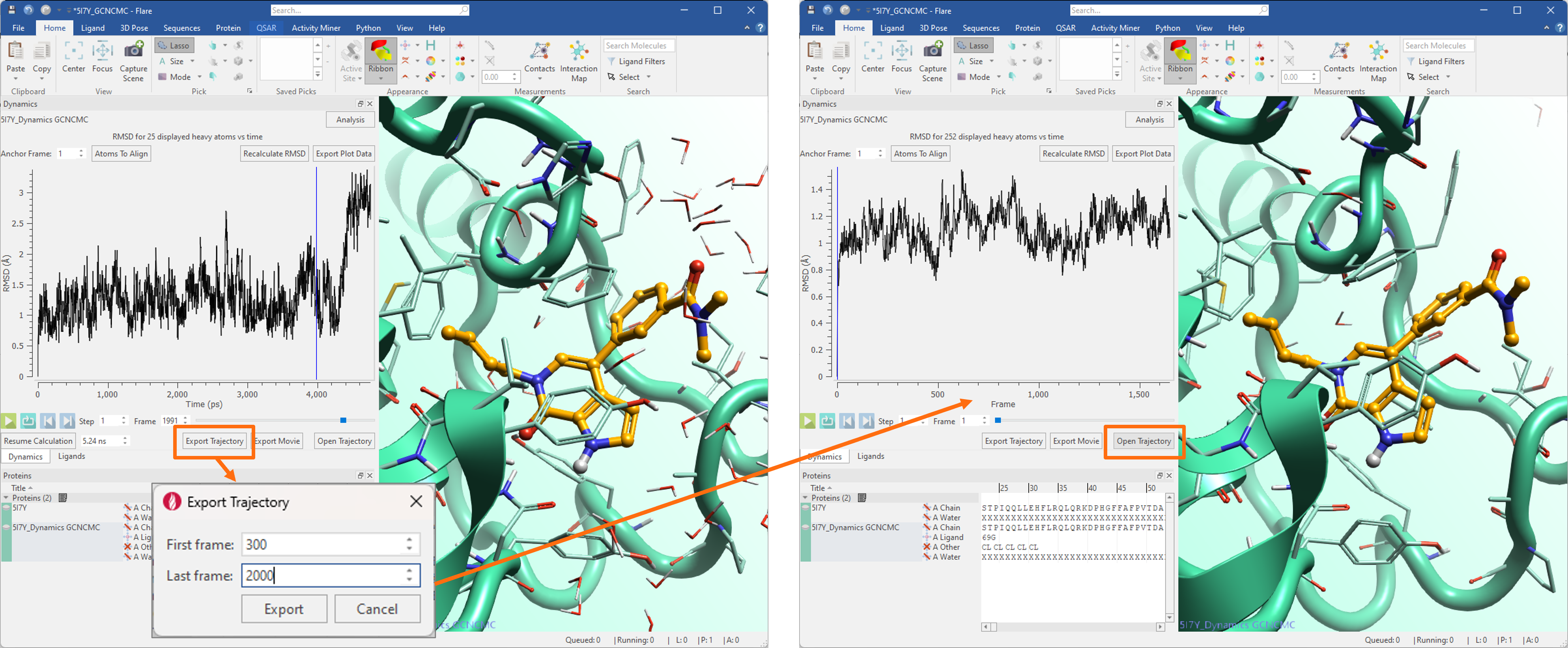
그림 6. 왼쪽: 새로운 ‘Export Trajectory’ 버튼을 이용하면 전체 trajectory 또는 관심 영역 내 프레임만을 내보낼 수 있습니다. 오른쪽: 내보낸 trajectory는 ‘Open Trajectory’ 버튼을 눌러 다시 Flare로 가져올 수 있습니다.
선택한 프레임 범위에서만 분석을 수행할 수 있게 되어 분자동역학 trajectories의 클러스터링도 향상되었습니다.
Flare V7에서는 새로운 hybrid DFT//GFN2-xTB 방법을 도입하여 분자동역학 및 Flare FEP 계산의 선택적 커스텀 torsion 파라미터 생성을 위한 알고리즘의 선택 폭을 더욱 확장하였습니다(그림 7).
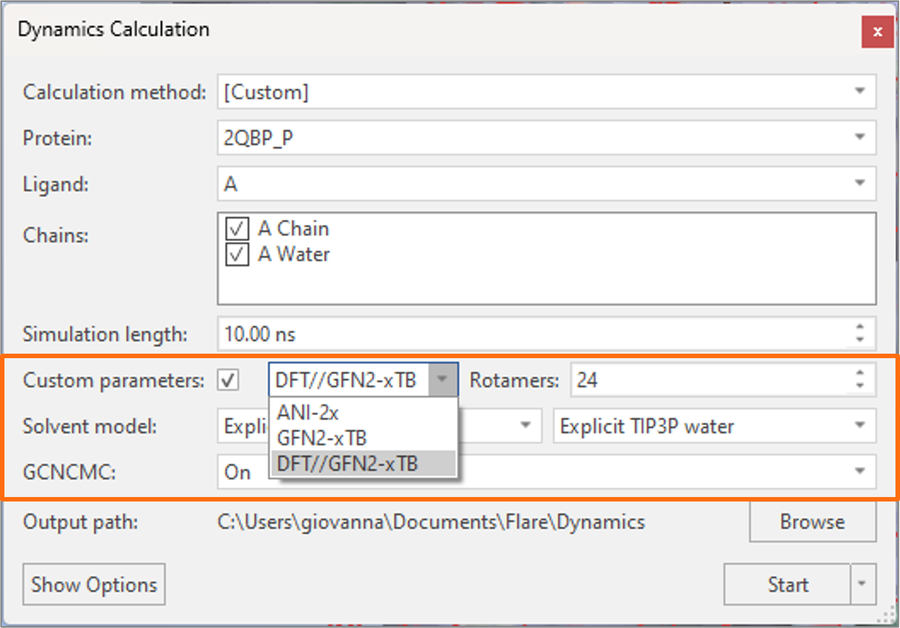
그림 7. Flare V7은 Open Force Field의 커스텀 torsion 파라미터 생성 알고리즘의 확장된 선택을 제공합니다.
이 방법은 모든 저분자(중성 및 하전 상태6)에 강력히 권장되며 GFN2-xTB tight-binding semi-empirical 방법으로 얻은 geometries에 대해 B3LYP-D3BJ/DZVP 수준에서 DFT single-point energies를 계산합니다. 이처럼, 빠른 semi-empirical geometry optimization을 통해 우수한 계산 성능을 달성하는 한편 양질의 에너지 계산 수행을 위해 ab-initio 방법이 이용됩니다.
기존 ANI-2X7 및 GFN2-xTB5 방법도 향상된 fragmentation 알고리즘으로 업데이트되었습니다.
새로운 ‘Consensus’ 회귀 및 분류 모델이 Flare의 머신러닝(ML) 방법의 기존 패널에 추가되었습니다(그림 8). Consensus 회귀 모델(그림 8 – 왼쪽)은 Gaussian Process, MultiLayer Perceptron, Random Forest and Support Vector Machine 모델을 실행하고 각 개별 모델의 예측을 평균내어 화합물의 활성을 예측합니다. 대신에, Consensus 분류(그림 8 – 오른쪽)는 MultiLayer Perceptron과 Random Forest 분류 모델에서 예측 확률의 합이 가장 높은 클래스로 분자의 클래스/카테고리를 예측합니다.
이 모델은 예측을 위한 ‘consensus’를 생성하며, 모든 회귀 또는 분류 모델이 동의하는 리간드의 우선순위를 정할 수 있습니다.
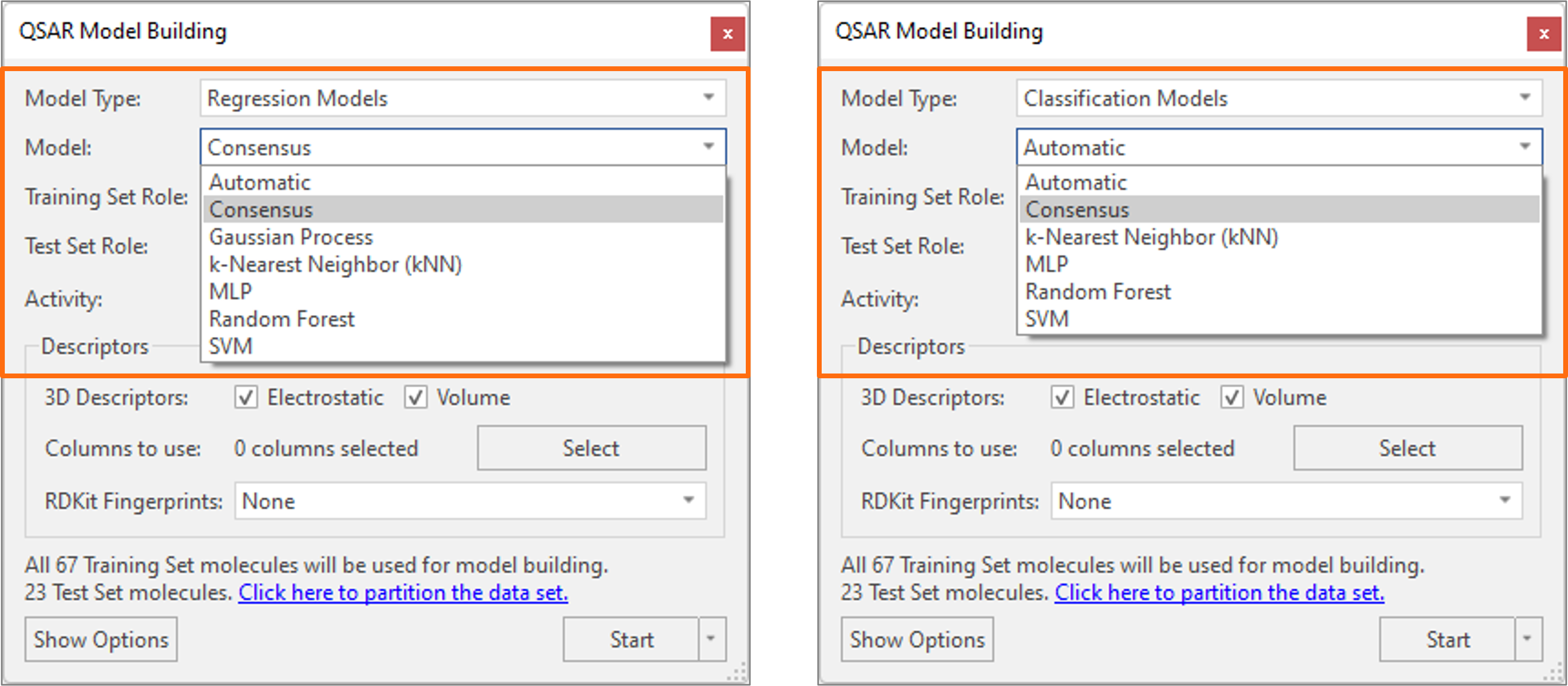
그림 8. Consensus 회귀(왼쪽) 및 분류(오른쪽) 모델은 모든 머신러닝 회귀 또는 분류 모델이 동의하는 리간드의 우선 순위를 정할 수 있습니다.
Flare V7에서는 머신러닝 회귀 및 분류 모델을 여러 RDKit fingerprint들8 중 하나를 선택하여 손쉽게(seamlessly) 구축할 수 있습니다(그림 9 – 왼쪽).
또한, 일반적으로 사용되는 물리화학적 표현자9를 Column & Activity Editor를 이용하여 RDKit에서 리간드 테이블로 가져올 수 있으며(그림 9 – 오른쪽), 활성 및 ADMET 특성 예측 QSAR 모델을 구축시 Cresset 3D 표현자(정전기 및 형상)를 대신하여 이용할 수 있습니다.
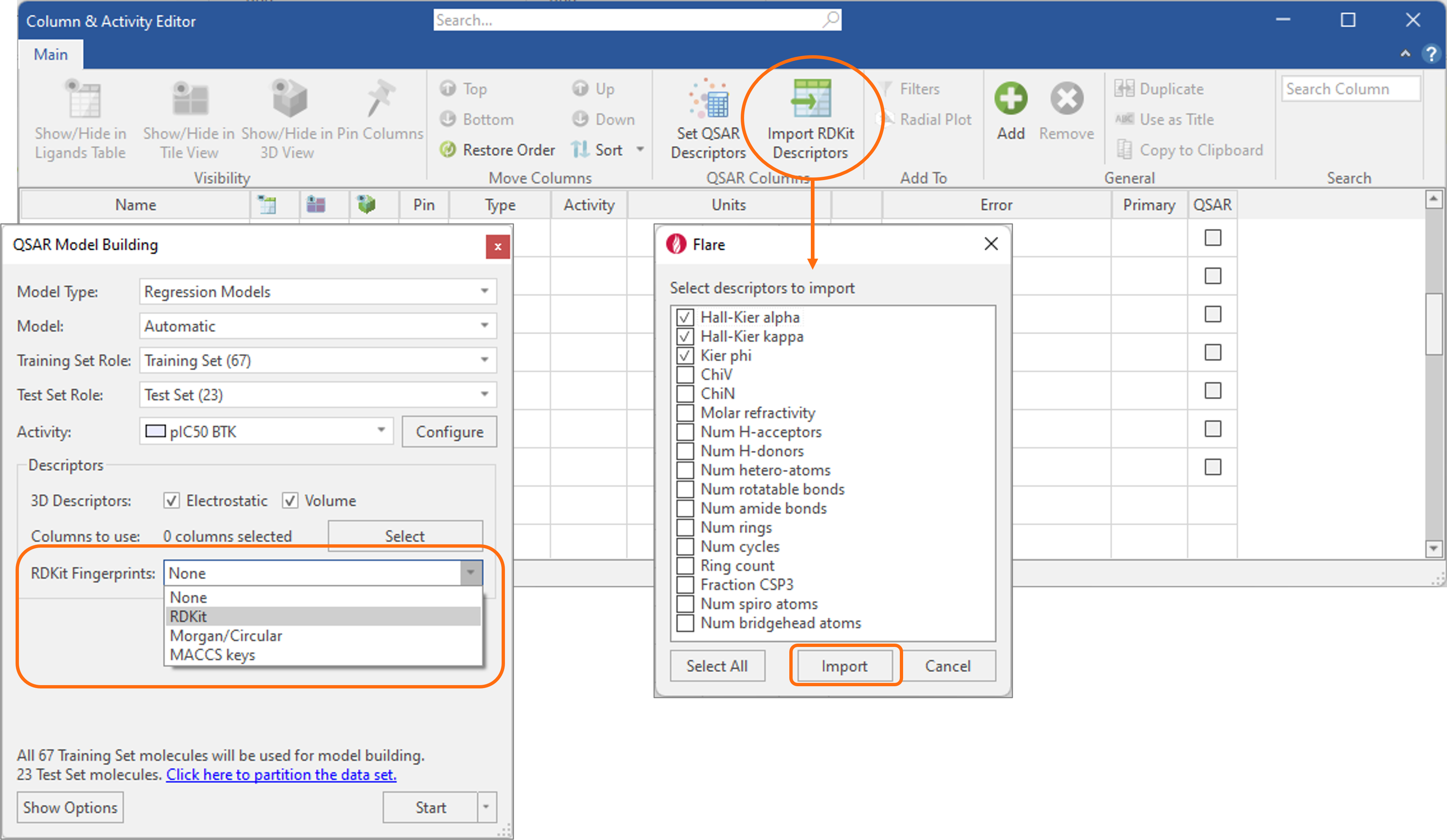
그림 9. Flare V7에서는 RDKit fingerprint 및 물리화학적 표현자를 이용하여 활성 및 ADMET 특성 예측 QSAR 모델을 구축할 수 있습니다.
마지막으로, Gaussian Process 회귀 모델은 이제 각 예측치에 대한 표준 편차 값을 계산하므로 화합물을 합성하고 테스트하기 전에 Flare의 예측 신뢰성에 대한 확신을 얻을 수 있습니다.
Flare V7의 새로운 ‘Ensemble covalent’ 방법은 일반적인 두 가지 docking 방법(구체적으로, 공유 결합 억제제의 결합 포즈를 정밀하게 예측하는 covalent docking과 단일 docking 실험에서 동일 단백질의 대체 가능 활성 부위 conformation들을 포함시켜 활성 부위의 유연성을 고려한 ensemble docking)을 통합한 것입니다.
Covalent docking과 마찬가지로 docking할 리간드는 Flare가 지원하는 covalent warhead들(20개 이상) 중 하나를 반드시 가지고 있어야 합니다. 만일, 사용하려는 covalent warhead가 지원되지 않는 경우에는 이를 쉽게 Flare에 추가하고 다른 동료들과 공유할 수 있습니다(자세한 내용은 Cresset 지원팀에 문의하십시오). Ensemble docking과 마찬가지로 연구에 이용할 단백질 conformation들은 3D로 다같이 정렬(align)되어야 합니다(그림 10).
Docking grid(리간드의 docking 영역)는 알려진 결정학적 리간드의 위치를 기반으로 하거나 원하는 단백질 원자를 수동으로 선택하여 쉽게 정의할 수 있습니다.
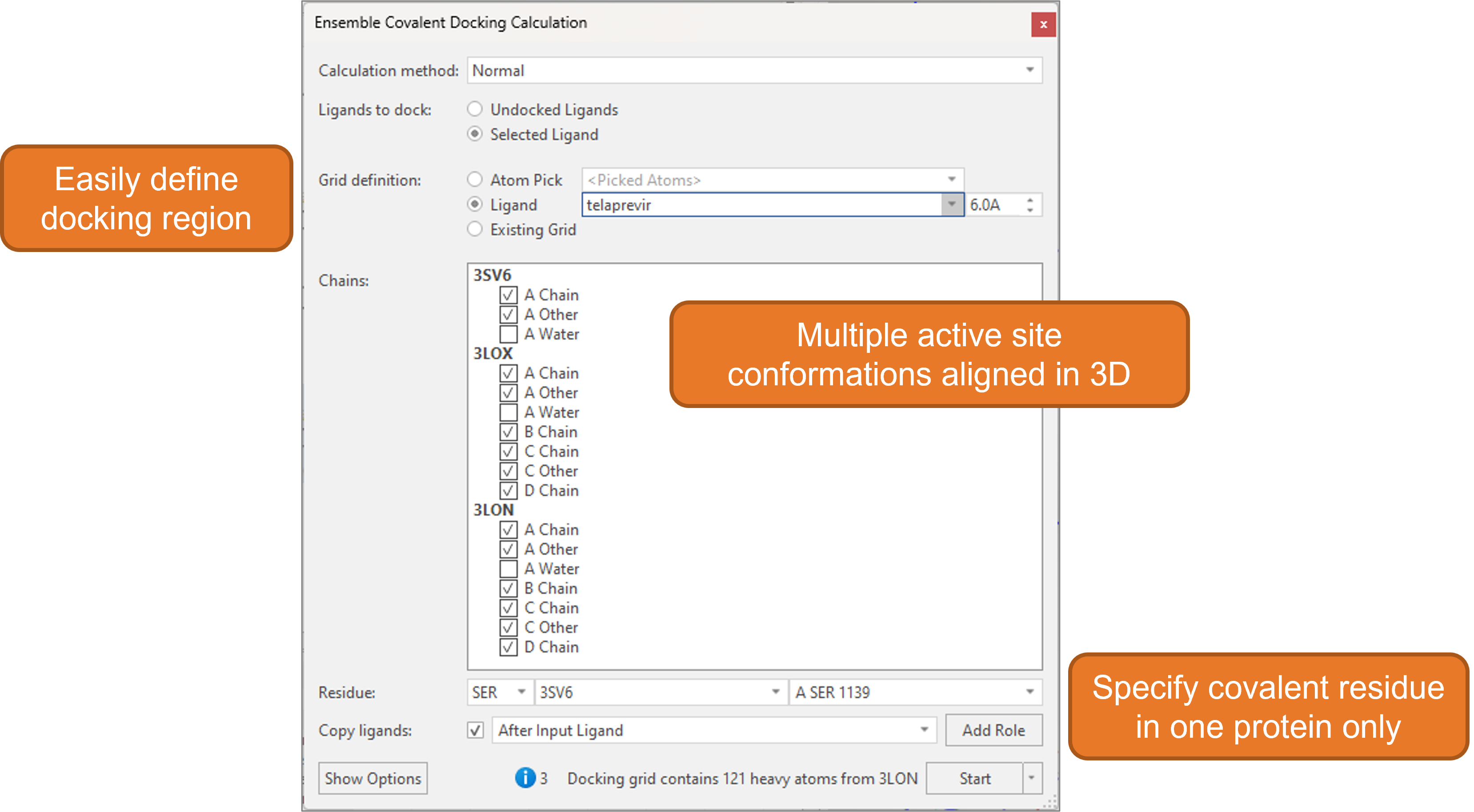
그림 10. Flare의 계산 패널을 통해 ensemble covalent docking 실험을 쉽게 설정할 수 있습니다.
여러 단백질 중 단 하나의 covalent 잔기만을 정의하는 것으로 충분합니다. Flare는 중첩(superimpose)된 다른 단백질 구조의 해당 covalent 잔기를 자동으로 감지합니다. 대신에, 각 단백질에서 covalent 잔기를 개별적으로 수동 선택도 가능합니다.
Ensemble covalent docking 실험이 완료되면 Flare의 covalent docking 알고리즘에 의해 예측된 결합 포즈가 Ligands 테이블에 저장됩니다. 가장 낮은 Rank Score를 갖는 포즈는 결정학적 포즈를 재현할 가능성이 가장 높습니다(그림 11).
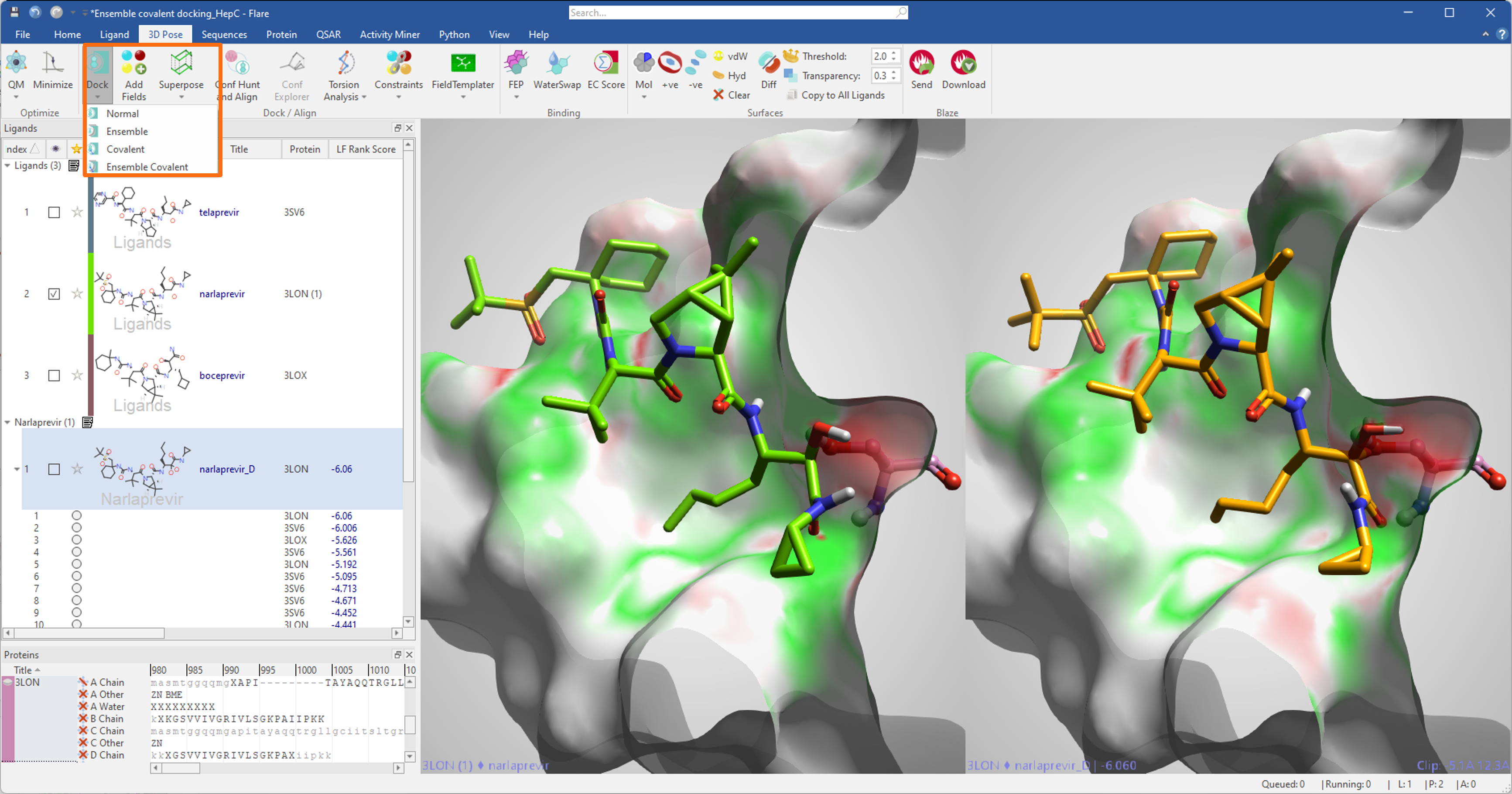
그림 11. HCV NS3/4A protease 복합체의 covalent inhibitors에 대한 ensemble covalent docking 실험 결과. HCV NS3/4A protease의 세 가지 다른 conformation들(PDBs 3SV6, 3LON, 3LOX)에 대한 ensemble covalent docking 실험에는 narlaprevir의 2D 구조가 사용됨. Flare의 최고 득점 포즈(오른쪽 그림, 주황색)는 PDB: 3LON의 narlaprevir의 결정학적 포즈(왼쪽 그림, 녹색)를 잘 재현합니다. 단백질 표면은 Electrostatic Complementarity에 따라 색상으로 표시함(녹색 = 양호한 정전기적 상보성; 빨강 = 정전기적 충돌).
Flare V7에서는 covalent docking의 새로운 고급 옵션을 통해 실험에 사용할 warhead를 SMARTS 정의를 입력하여 지정할 수 있습니다. 이 옵션은 docking하려는 리간드가 하나 이상의 잠재 covalent warhead를 가지고 있는 경우와 사용자가 화학 계열의 지식을 이용하여 반응 그룹을 제어하려는 경우에 유용합니다.
Flare의 모든 docking 방법에 대한 또 다른 새로운 고급 옵션으로 사용자는 도킹 실험 중에 활성 부위의 어떤 H-bond donor 단백질 잔기(Serine, Threonine, Tyrosine 및 Lysine 중 하나 또는 모두 선택 가능)가 회전하도록 허용할지 지정할 수 있습니다. 이 옵션을 통해 docking 실험에 추가적인 방식으로 곁사슬의 유연성을 허용할 수 있으며 단백질 활성 부위에 최적의 상호작용을 형성할 수 있습니다.
이번 릴리즈에서는 다수의 작업을 동시에 실행할 수 있도록 하여 계산 효율을 크게 향상시켰습니다(그림 12).
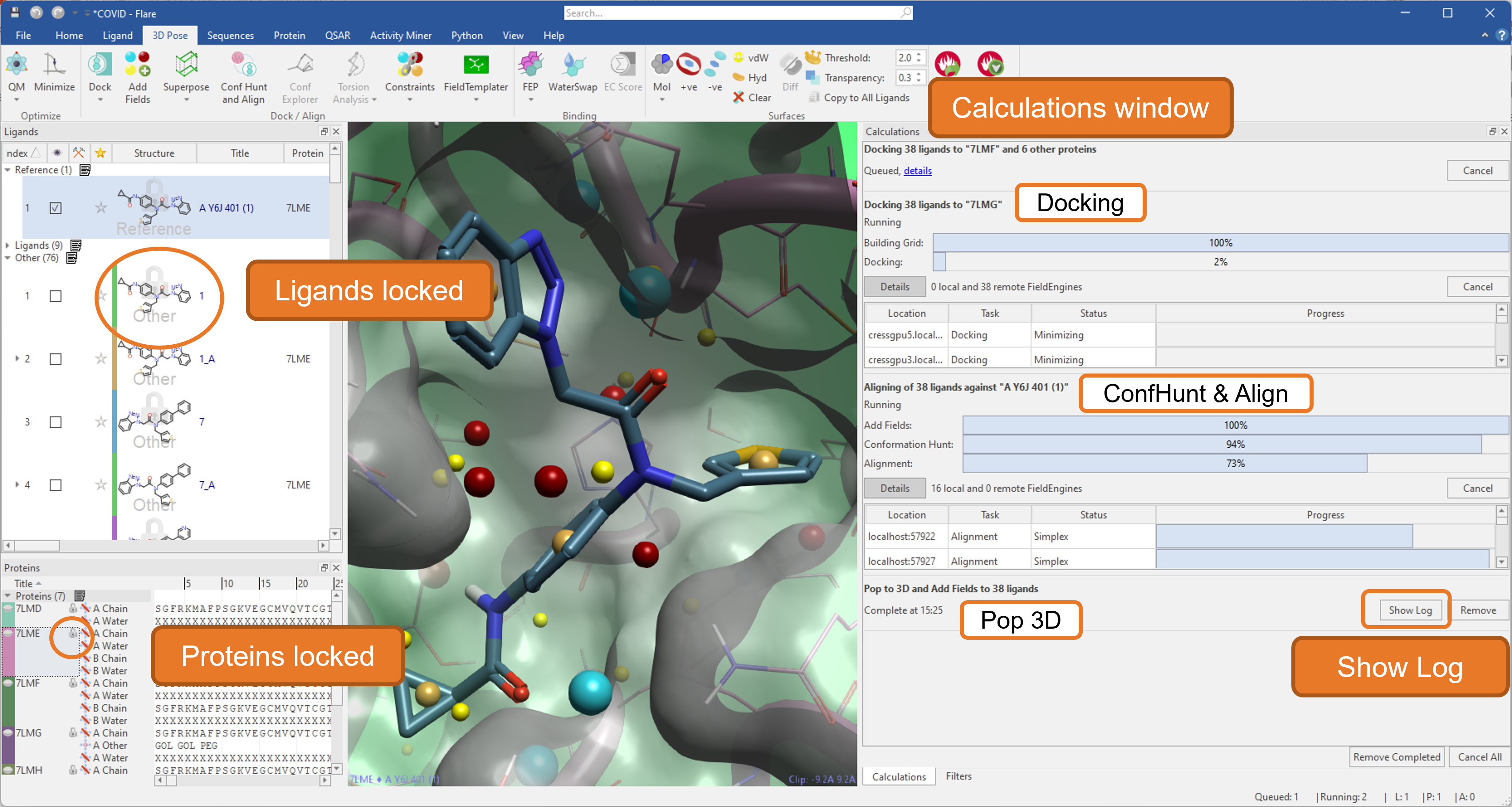
그림 12. Flare의 다중 작업
지능형 잠금 시스템은 작업이 완료될 때까지 작업에 이용된 리간드 및 단백질을 의도치 않은 삭제 또는 수정으로부터 보호합니다. 또한, 새롭게 디자인된 Calculations 창에서는 실행 중, 대기 중, 완료된 모든 작업을 모니터링 할 수 있으며, 각 계산의 로그에 쉽게 접근할 수 있게 되었습니다.
이번 릴리즈의 Ligand Prep 기능에서는 tautomer enumeration에 새로운 옵션이 추가되어 Cresset 규칙을 기반으로 canonical tautomer만을 생성할지 또는 수용액에서 가능성 높은 것에서 낮은 것까지의 tautomer를 생성할지를 선택할 수 있게 되었습니다(그림 13).
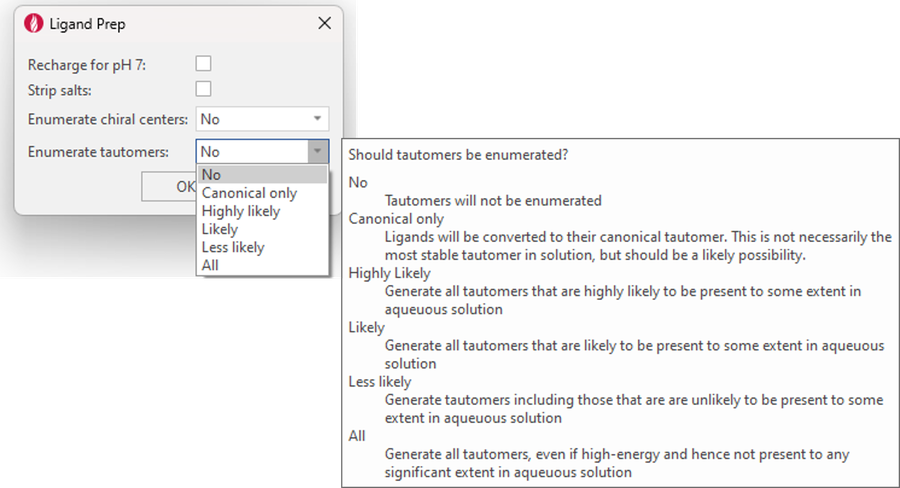
그림 13. Ligand Prep의 새로운 tautomer enumeration 옵션
Flare V7에서는 Protein Prep 기능에 새로운 옵션이 추가되어 입력 PDB파일에 기재된 대로 생물학적 어셈블리를 자동화된 방식으로 쉽게 가져올 수 있게 되었습니다.
그림 14의 왼쪽은 기본 옵션을 적용하여 prep한 PDB:6TOH(확산성 거대 B세포 림프종 치료를 위한 잠재 치료 타겟인 BCL6에 결합된 CCT36538610의 결정 구조)를 나타낸 것입니다. 오른쪽 그림은 ‘Create biological assembly’ 옵션을 이용하여 prep한 PDB:6TOH입니다. BCL6의 기능성 이량체 구조가 자동으로 가져오기 되어 결합 부위 내 물 충전 포켓이 명확하게 보여집니다.
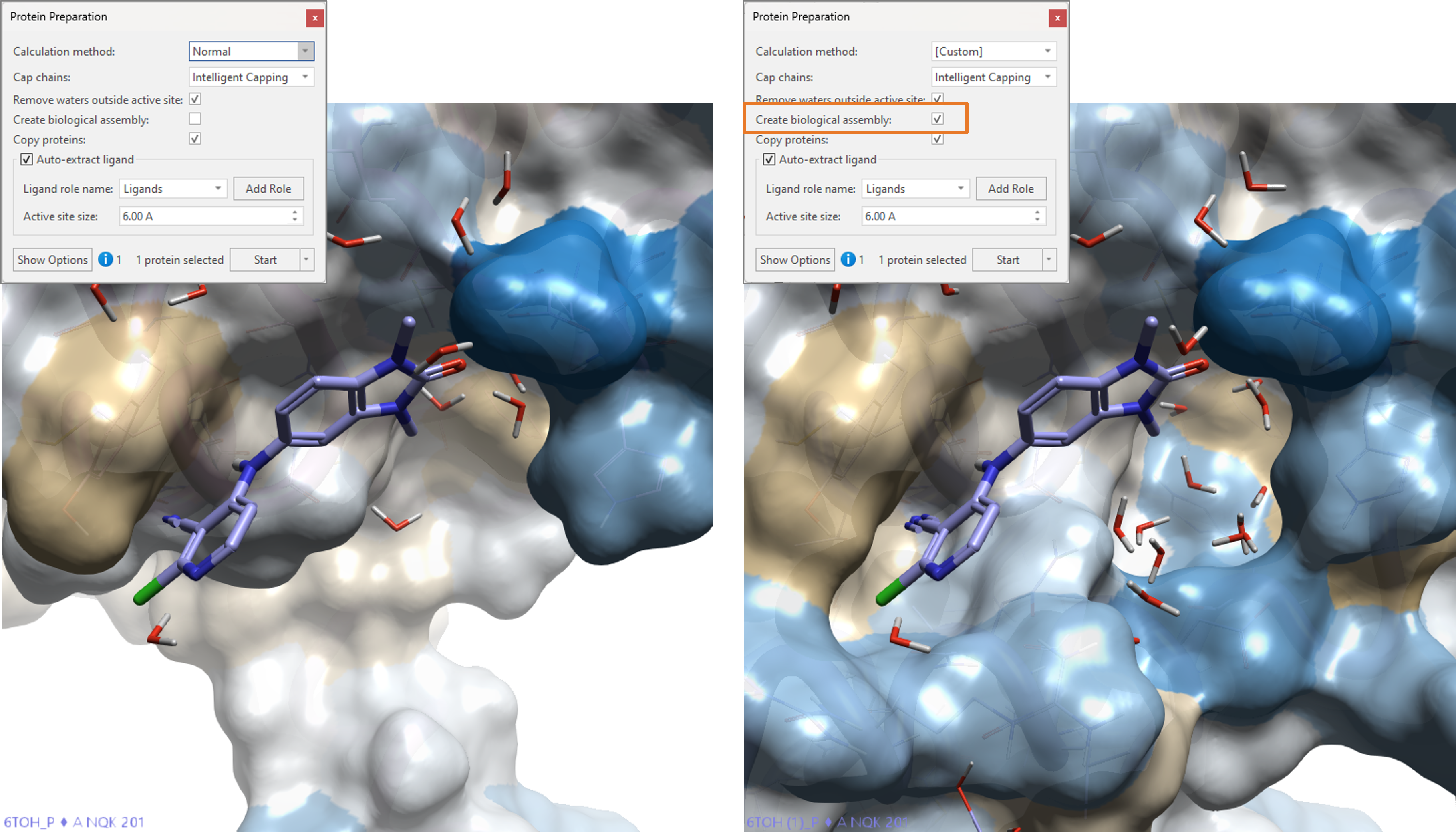
그림 14. 기본 옵션(왼쪽)과 ‘Create biological assembly’ 옵션(오른쪽)을 이용하여 prep한 PDB: 6TOH. 오른쪽 그림의 경우, BCL6의 기능성 이량체 형태가 자동으로 생성됨을 확인할 수 있음. 활성 부위의 표면은 소수성(hydrophobicity)에 따라 색상으로 표시함11.
Flare V7의 그 외 새로운 기능 및 개선 사항은 다음과 같습니다.
# Role을 이용하여 Proteins and Alignments 테이블을 구성하는 기능
# 향상된 단백질 모델링:
# 편집기 내에서 펩타이드를 구축하고 단백질을 성장시키는 기능
# ‘Copy/Paste Loop’ 기능으로 다른 단백질 구조에서 루프를 복사하여 관심 단백질의 틈(gaps) 메우기가 가능
# 원하는 위치에 일부 시퀀스를 삽입하는 기능
# 3D 좌표가 없는 단백질 시퀀스 내 잔기를 위한 기능으로 잔기 선택, 삭제, 새 사슬로 배치 및 사슬 분할이 가능
# Single-point mutations 워크플로를 이용하여 mutated residues만을 prep할지 또는 전체 단백질을 prep할지 선택 가능
#Proteins and Alignments 테이블의 interactive sequence ruler bar를 이용하여 전체 단백질의 동일 위치에 대한 모든 잔기의 강조 표시/선택 가능
# Library enumeration 기능의 향상으로 명확한 명명과 확장된 반응 정의 가능 및 구조 필터의 내보내기/가져오기 옵션 지원
# Mogul torsion 분석 방법의 새로운 인터페이스 지원(유효한 CSD 라이선스 필요)
# Flare GUI 상단에 ‘Search’ 상자를 새롭게 추가하여 ribbon tab 버튼, 메뉴 항목, dockable window 등 원하는 Flare 기능을 빠르게 검색 가능
# Spin/Rock Movie를 내보내는 기능을 새롭게 추가
# Interactive Radial Plots
# 태그를 기반으로 AND/OR 로직을 이용하여 리간드 필터링 가능
# Hit Expander의 치환체 선택 폭 확장
# R-Group Analysis 결과의 향상된 시각화
# Storyboard 내 기존 scenes에 대한 업데이트 가능
# Scatter plots의 리간드 타이틀의 새로운 영구 주석
리간드 기반 또는 구조 기반 방법 또는 두 방법 모두에 관심이 있는 분들에게 Flare는 계산 화학자, 의약 화학자 및 학계 종사자에 적합한 유연한 라이선스 옵션을 제공합니다. 프로젝트에 Flare V7을 테스트해 보고 싶으시다면 지금 바로 평가판을 요청해 보십시오.
평가 과정의 일환으로 플랫폼을 설치하고 다양한 기능에 액세스하는 데 필요한 모든 지원을 받을 수 있으며, 생성된 결과를 자유롭게 게시하고 추가 연구에 사용할 수 있습니다.